+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of activated transcription complex Pol II-DSIF-PAF-SPT6, KOW1 selected particles (Map C) | |||||||||
 マップデータ マップデータ | Global refinement of KOW1 selected EC* particles. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.49 Å | |||||||||
 データ登録者 データ登録者 | Vos SM / Farnung L / Boehning M / Linden A / Wigge C / Urlaub H / Cramer P | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Structure of activated transcription complex Pol II-DSIF-PAF-SPT6. 著者: Seychelle M Vos / Lucas Farnung / Marc Boehning / Christoph Wigge / Andreas Linden / Henning Urlaub / Patrick Cramer /  要旨: Gene regulation involves activation of RNA polymerase II (Pol II) that is paused and bound by the protein complexes DRB sensitivity-inducing factor (DSIF) and negative elongation factor (NELF). Here ...Gene regulation involves activation of RNA polymerase II (Pol II) that is paused and bound by the protein complexes DRB sensitivity-inducing factor (DSIF) and negative elongation factor (NELF). Here we show that formation of an activated Pol II elongation complex in vitro requires the kinase function of the positive transcription elongation factor b (P-TEFb) and the elongation factors PAF1 complex (PAF) and SPT6. The cryo-EM structure of an activated elongation complex of Sus scrofa Pol II and Homo sapiens DSIF, PAF and SPT6 was determined at 3.1 Å resolution and compared to the structure of the paused elongation complex formed by Pol II, DSIF and NELF. PAF displaces NELF from the Pol II funnel for pause release. P-TEFb phosphorylates the Pol II linker to the C-terminal domain. SPT6 binds to the phosphorylated C-terminal-domain linker and opens the RNA clamp formed by DSIF. These results provide the molecular basis for Pol II pause release and elongation activation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|---|
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0032.map.gz emd_0032.map.gz | 140.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0032-v30.xml emd-0032-v30.xml emd-0032.xml emd-0032.xml | 23.5 KB 23.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0032_fsc.xml emd_0032_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0032.png emd_0032.png | 76.7 KB | ||
| マスクデータ |  emd_0032_msk_1.map emd_0032_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0032_additional.map.gz emd_0032_additional.map.gz emd_0032_half_map_1.map.gz emd_0032_half_map_1.map.gz emd_0032_half_map_2.map.gz emd_0032_half_map_2.map.gz | 15.3 MB 140.8 MB 140.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0032 http://ftp.pdbj.org/pub/emdb/structures/EMD-0032 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0032 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0032 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0032_validation.pdf.gz emd_0032_validation.pdf.gz | 447.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0032_full_validation.pdf.gz emd_0032_full_validation.pdf.gz | 446.8 KB | 表示 | |
| XML形式データ |  emd_0032_validation.xml.gz emd_0032_validation.xml.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0032 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0032 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0032 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0032 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0032.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0032.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Global refinement of KOW1 selected EC* particles. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.049 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_0032_msk_1.map emd_0032_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
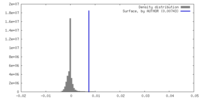
| 密度ヒストグラム |
-追加マップ: Postprocessed map of global refinement for KOW1 selected...
| ファイル | emd_0032_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map of global refinement for KOW1 selected particles with an applied B factor of -90.57(Map C). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
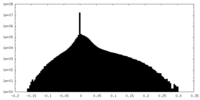
| 密度ヒストグラム |
-ハーフマップ: Half map 2 of global refinement for KOW1 selected particles (Map C).
| ファイル | emd_0032_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 of global refinement for KOW1 selected particles (Map C). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
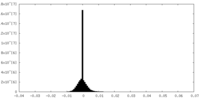
| 密度ヒストグラム |
-ハーフマップ: Half map 1 of global refinement for KOW1 selected particles (Map C).
| ファイル | emd_0032_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 of global refinement for KOW1 selected particles (Map C). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
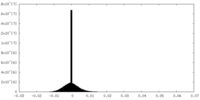
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA Polymerase II-DSIF-PAF-SPT6 elongation complex (EC*)
| 全体 | 名称: RNA Polymerase II-DSIF-PAF-SPT6 elongation complex (EC*) |
|---|---|
| 要素 |
|
-超分子 #1: RNA Polymerase II-DSIF-PAF-SPT6 elongation complex (EC*)
| 超分子 | 名称: RNA Polymerase II-DSIF-PAF-SPT6 elongation complex (EC*) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#21, #23 |
|---|---|
| 分子量 | 理論値: 1.257 MDa |
-超分子 #2: RNA Polymerase II
| 超分子 | 名称: RNA Polymerase II / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#9, #11, #10, #12 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: associated proteins
| 超分子 | 名称: associated proteins / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #13, #16, #18, #23, #19-#22 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-超分子 #4: Nucleic acids
| 超分子 | 名称: Nucleic acids / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #14-#15, #17 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種: synthetic construct (人工物) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 10s wait prior to blotting, blotting 8.5s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 10.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)