+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDCW8 |
|---|---|
 試料 試料 | Human Rev7 monomer (R124A mutant) in complex with Rev3 peptide @ 4mg/mL
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報somatic diversification of immunoglobulins involved in immune response / DNA damage response, signal transduction resulting in transcription / zeta DNA polymerase complex / positive regulation of isotype switching / negative regulation of transcription by competitive promoter binding / negative regulation of cell-cell adhesion mediated by cadherin / JUN kinase binding / negative regulation of epithelial to mesenchymal transition / positive regulation of double-strand break repair via nonhomologous end joining / mitotic spindle assembly checkpoint signaling ...somatic diversification of immunoglobulins involved in immune response / DNA damage response, signal transduction resulting in transcription / zeta DNA polymerase complex / positive regulation of isotype switching / negative regulation of transcription by competitive promoter binding / negative regulation of cell-cell adhesion mediated by cadherin / JUN kinase binding / negative regulation of epithelial to mesenchymal transition / positive regulation of double-strand break repair via nonhomologous end joining / mitotic spindle assembly checkpoint signaling / negative regulation of ubiquitin protein ligase activity / telomere maintenance in response to DNA damage / positive regulation of peptidyl-serine phosphorylation / error-prone translesion synthesis / negative regulation of double-strand break repair via homologous recombination / actin filament organization / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI / regulation of cell growth / negative regulation of canonical Wnt signaling pathway / double-strand break repair via homologous recombination / negative regulation of protein catabolic process / DNA-templated DNA replication / spindle / transcription corepressor activity / double-strand break repair / site of double-strand break / chromosome / 4 iron, 4 sulfur cluster binding / DNA-directed DNA polymerase / RNA polymerase II-specific DNA-binding transcription factor binding / DNA-directed DNA polymerase activity / cell division / nucleotide binding / positive regulation of DNA-templated transcription / chromatin / nucleolus / negative regulation of transcription by RNA polymerase II / DNA binding / zinc ion binding / nucleoplasm / nucleus / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Rev7 dimerization is important for assembly and function of the Rev1/Polζ translesion synthesis complex. 著者: Alessandro A Rizzo / Faye-Marie Vassel / Nimrat Chatterjee / Sanjay D'Souza / Yunfeng Li / Bing Hao / Michael T Hemann / Graham C Walker / Dmitry M Korzhnev /  要旨: The translesion synthesis (TLS) polymerases Polζ and Rev1 form a complex that enables replication of damaged DNA. The Rev7 subunit of Polζ, which is a multifaceted HORMA (Hop1, Rev7, Mad2) protein ...The translesion synthesis (TLS) polymerases Polζ and Rev1 form a complex that enables replication of damaged DNA. The Rev7 subunit of Polζ, which is a multifaceted HORMA (Hop1, Rev7, Mad2) protein with roles in TLS, DNA repair, and cell-cycle control, facilitates assembly of this complex by binding Rev1 and the catalytic subunit of Polζ, Rev3. Rev7 interacts with Rev3 by a mechanism conserved among HORMA proteins, whereby an open-to-closed transition locks the ligand underneath the "safety belt" loop. Dimerization of HORMA proteins promotes binding and release of this ligand, as exemplified by the Rev7 homolog, Mad2. Here, we investigate the dimerization of Rev7 when bound to the two Rev7-binding motifs (RBMs) in Rev3 by combining in vitro analyses of Rev7 structure and interactions with a functional assay in a Rev7 cell line. We demonstrate that Rev7 uses the conventional HORMA dimerization interface both to form a homodimer when tethered by the two RBMs in Rev3 and to heterodimerize with other HORMA domains, Mad2 and p31 Structurally, the Rev7 dimer can bind only one copy of Rev1, revealing an unexpected Rev1/Polζ architecture. In cells, mutation of the Rev7 dimer interface increases sensitivity to DNA damage. These results provide insights into the structure of the Rev1/Polζ TLS assembly and highlight the function of Rev7 homo- and heterodimerization. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-Data source
| SASBDBのページ |  SASDCW8 SASDCW8 |
|---|
-関連構造データ
- 外部リンク
外部リンク
| 「今月の分子」の関連する項目 |
|---|
-モデル
| モデル #1624 |  タイプ: atomic / ダミー原子の半径: 1.90 A コメント: residues at N- and C-termini that were missing in PDB were built in extended conformation カイ2乗値: 2.67595863267  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|
- 試料
試料
 試料 試料 | 名称: Human Rev7 monomer (R124A mutant) in complex with Rev3 peptide @ 4mg/mL 試料濃度: 4 mg/ml / Entity id: 854 / 855 |
|---|---|
| バッファ | 名称: 20 mM Tris, 150 mM NaCl, 1 mM EDTA, 10 mM DTT, 5% glycerol pH: 8.4 |
| 要素 #854 | 名称: hRev7-R124A / タイプ: protein / 記述: Mitotic spindle assembly checkpoint protein MAD2B / 分子量: 24.306 / 分子数: 1 / 由来: Homo sapiens / 参照: UniProt: Q9UI95 配列: GMTTLTRQDL NFGQVVADVL CEFLEVAVHL ILYVREVYPV GIFQKRKKYN VPVQMSCHPE LNQYIQDTLH CVKPLLEKND VEKVVVVILD KEHRPVEKFV FEITQPPLLS ISSDSLLSHV EQLLAAFILK ISVCDAVLDH NPPGCTFTVL VHTREAATRN MEKIQVIKDF ...配列: GMTTLTRQDL NFGQVVADVL CEFLEVAVHL ILYVREVYPV GIFQKRKKYN VPVQMSCHPE LNQYIQDTLH CVKPLLEKND VEKVVVVILD KEHRPVEKFV FEITQPPLLS ISSDSLLSHV EQLLAAFILK ISVCDAVLDH NPPGCTFTVL VHTREAATRN MEKIQVIKDF PWILADEQDV HMHDPRLIPL KTMTSDILKM QLYVEERAHK GS |
| 要素 #855 | 名称: Rev3-RBM2 / タイプ: protein / 記述: DNA polymerase zeta catalytic subunit / 分子量: 3.273 / 分子数: 1 / 由来: Homo sapiens / 参照: UniProt: O60673 配列: MEDKKIVIMP CKCAPSRQLV QVWLQAKE |
-実験情報
| ビーム | 設備名称: Cornell High Energy Synchrotron Source (CHESS) G1 地域: Ithaca, NY / 国: USA  / 線源: X-ray synchrotron / 波長: 0.1267 Å / 線源: X-ray synchrotron / 波長: 0.1267 Å | |||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Finger Lakes CCD / タイプ: CCD | |||||||||||||||||||||||||||||||||||||||
| スキャン | 測定日: 2016年5月14日 / 保管温度: 4 °C / セル温度: 4 °C / 照射時間: 1 sec. / フレーム数: 10 / 単位: 1/A /
| |||||||||||||||||||||||||||||||||||||||
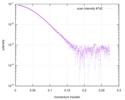
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー