+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDAE7 |
|---|---|
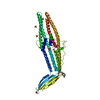
 試料 試料 | Structure of a complex between full length and truncated CS74L endolysin
|
| 機能・相同性 | : / Ami_3 / N-acetylmuramoyl-L-alanine amidase, catalytic domain / N-acetylmuramoyl-L-alanine amidase / N-acetylmuramoyl-L-alanine amidase activity / viral release from host cell by cytolysis / peptidoglycan catabolic process / Endolysin CS74L 機能・相同性情報 機能・相同性情報 |
| 生物種 |  Clostridium phage phi8074-B1 (ファージ) Clostridium phage phi8074-B1 (ファージ) |
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2016 ジャーナル: J Biol Chem / 年: 2016タイトル: Crystal Structure of the CTP1L Endolysin Reveals How Its Activity Is Regulated by a Secondary Translation Product. 著者: Matthew Dunne / Stefan Leicht / Boris Krichel / Haydyn D T Mertens / Andrew Thompson / Jeroen Krijgsveld / Dmitri I Svergun / Natalia Gómez-Torres / Sonia Garde / Charlotte Uetrecht / Arjan ...著者: Matthew Dunne / Stefan Leicht / Boris Krichel / Haydyn D T Mertens / Andrew Thompson / Jeroen Krijgsveld / Dmitri I Svergun / Natalia Gómez-Torres / Sonia Garde / Charlotte Uetrecht / Arjan Narbad / Melinda J Mayer / Rob Meijers /     要旨: Bacteriophages produce endolysins, which lyse the bacterial host cell to release newly produced virions. The timing of lysis is regulated and is thought to involve the activation of a molecular ...Bacteriophages produce endolysins, which lyse the bacterial host cell to release newly produced virions. The timing of lysis is regulated and is thought to involve the activation of a molecular switch. We present a crystal structure of the activated endolysin CTP1L that targets Clostridium tyrobutyricum, consisting of a complex between the full-length protein and an N-terminally truncated C-terminal cell wall binding domain (CBD). The truncated CBD is produced through an internal translation start site within the endolysin gene. Mutants affecting the internal translation site change the oligomeric state of the endolysin and reduce lytic activity. The activity can be modulated by reconstitution of the full-length endolysin-CBD complex with free CBD. The same oligomerization mechanism applies to the CD27L endolysin that targets Clostridium difficile and the CS74L endolysin that targets Clostridium sporogenes. When the CTP1L endolysin gene is introduced into the commensal bacterium Lactococcus lactis, the truncated CBD is also produced, showing that the alternative start codon can be used in other bacterial species. The identification of a translational switch affecting oligomerization presented here has implications for the design of effective endolysins for the treatment of bacterial infections. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #257 |  タイプ: dummy / ダミー原子の半径: 3.00 A / カイ2乗値: 0.775  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|---|
| モデル #258 |  タイプ: mix / ソフトウェア: (2.0) / ダミー原子の半径: 1.90 A コメント: Ensemble Component: Heterotetramer (compact) カイ2乗値: 0.664 / P-value: 0.051000  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #259 |  タイプ: mix / ダミー原子の半径: 1.90 A / コメント: Ensemble Component: Heterotetramer (extended) / カイ2乗値: 0.664 / P-value: 0.051000  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #260 | 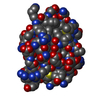 タイプ: mix / ソフトウェア: (2.0) / ダミー原子の半径: 1.90 A / コメント: Ensemble Component: Free CTD of endolysin / カイ2乗値: 0.664 / P-value: 0.051000  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
- 試料
試料
 試料 試料 | 名称: Structure of a complex between full length and truncated CS74L endolysin 試料濃度: 1.00-6.80 |
|---|---|
| バッファ | 名称: 20 mM HEPES / pH: 7.4 |
| 要素 #156 | 名称: CS74L / タイプ: protein / 記述: Endolysin CS74L / 分子量: 31.1 / 由来: Clostridium phage phi8074-B1 / 参照: UniProt: I1TJX3 配列: MGSSHHHHHH SSGLVPRGSH MKIGIDMGHT LSGADYGVVG LRPESVLTRE VGTKVIYKLQ KLGHVVVNCT VDKASSVSES LYTRYYRANQ ANVDLFISIH FNATPGGTGT EVYTYAGRQL GEATRIRQEF KSLGLRDRGT KDGSGLAVIR NTKAKAMLVE CCFCDNPNDM ...配列: MGSSHHHHHH SSGLVPRGSH MKIGIDMGHT LSGADYGVVG LRPESVLTRE VGTKVIYKLQ KLGHVVVNCT VDKASSVSES LYTRYYRANQ ANVDLFISIH FNATPGGTGT EVYTYAGRQL GEATRIRQEF KSLGLRDRGT KDGSGLAVIR NTKAKAMLVE CCFCDNPNDM KLYNSESFSN AIVKGITGKL PNGESGNNNQ GGNKVKAVVI YNEGADRRGA EYLADYLNCP TISNSRTFDY SCVEHVYAVG GKKEQYTKYL KTLLSGANRY DTMQQILNFI NGGK |
-実験情報
| ビーム | 設備名称:  DORIS III X33 DORIS III X33  / 地域: Hamburg / 国: Germany / 地域: Hamburg / 国: Germany  / 形状: 0.6 / 線源: X-ray synchrotron / 波長: 0.15 Å / スペクトロメータ・検出器間距離: 2.7 mm / 形状: 0.6 / 線源: X-ray synchrotron / 波長: 0.15 Å / スペクトロメータ・検出器間距離: 2.7 mm | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M-W / Pixsize x: 0.172 mm | |||||||||||||||||||||
| スキャン |
| |||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー


 SASDAE7
SASDAE7