+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8grq | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of BRCA1/BARD1 bound to H2AK127-UbcH5c-Ub nucleosome | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | NUCLEAR PROTEIN / nucleosome / BRCA1/BARD1 / BRCA1 / BARD1 / H2AK127 / H2AK127-UbcH5c-Ub | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Inhibition of DNA recombination at telomere / negative regulation of mRNA 3'-end processing / Deposition of new CENPA-containing nucleosomes at the centromere / DNA Damage/Telomere Stress Induced Senescence / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Condensation of Prophase Chromosomes / Metalloprotease DUBs / Defective DNA double strand break response due to BRCA1 loss of function / Defective DNA double strand break response due to BARD1 loss of function ...Inhibition of DNA recombination at telomere / negative regulation of mRNA 3'-end processing / Deposition of new CENPA-containing nucleosomes at the centromere / DNA Damage/Telomere Stress Induced Senescence / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Condensation of Prophase Chromosomes / Metalloprotease DUBs / Defective DNA double strand break response due to BRCA1 loss of function / Defective DNA double strand break response due to BARD1 loss of function / HDACs deacetylate histones / BRCA1-BARD1 complex / BRCA1-C complex / PRC2 methylates histones and DNA / BRCA1-B complex / UCH proteinases / BRCA1-A complex / random inactivation of X chromosome / negative regulation of centriole replication / sex-chromosome dosage compensation / negative regulation of intracellular estrogen receptor signaling pathway / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / gamma-tubulin ring complex / RMTs methylate histone arginines / nuclear ubiquitin ligase complex / DNA strand resection involved in replication fork processing / chordate embryonic development / cellular response to indole-3-methanol / negative regulation of fatty acid biosynthetic process / lateral element / homologous recombination / tissue homeostasis / protein K6-linked ubiquitination / regulation of DNA damage checkpoint / Ub-specific processing proteases / regulation of phosphorylation / Impaired BRCA2 binding to PALB2 / XY body / mitotic G2/M transition checkpoint / negative regulation of protein export from nucleus / postreplication repair / DNA repair complex / RNA polymerase binding / centrosome cycle / Defective homologous recombination repair (HRR) due to BRCA1 loss of function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA1 binding function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA2/RAD51/RAD51C binding function / Homologous DNA Pairing and Strand Exchange / Resolution of D-loop Structures through Synthesis-Dependent Strand Annealing (SDSA) / Resolution of D-loop Structures through Holliday Junction Intermediates / negative regulation of gene expression via chromosomal CpG island methylation / intracellular non-membrane-bounded organelle / HDR through Single Strand Annealing (SSA) / Impaired BRCA2 binding to RAD51 / DNA-binding transcription activator activity / response to ionizing radiation / Transcriptional Regulation by E2F6 / mitotic G2 DNA damage checkpoint signaling / Presynaptic phase of homologous DNA pairing and strand exchange / negative regulation of cell cycle / localization / positive regulation of vascular endothelial growth factor production / negative regulation of reactive oxygen species metabolic process / protein autoubiquitination / regulation of DNA repair / SUMOylation of DNA damage response and repair proteins / negative regulation of extrinsic apoptotic signaling pathway via death domain receptors / ubiquitin ligase complex / nucleosomal DNA binding / Meiotic synapsis / positive regulation of DNA repair / tubulin binding / male germ cell nucleus / chromosome segregation / cellular response to ionizing radiation / TP53 Regulates Transcription of DNA Repair Genes / Nonhomologous End-Joining (NHEJ) / heterochromatin formation / double-strand break repair via homologous recombination / RING-type E3 ubiquitin transferase / HDR through Homologous Recombination (HRR) / G2/M DNA damage checkpoint / negative regulation of cell growth / Metalloprotease DUBs / Meiotic recombination / kinase binding / fatty acid biosynthetic process / cytoplasmic ribonucleoprotein granule / positive regulation of protein catabolic process / structural constituent of chromatin / positive regulation of angiogenesis / ubiquitin-protein transferase activity / UCH proteinases / intrinsic apoptotic signaling pathway in response to DNA damage / KEAP1-NFE2L2 pathway / nucleosome / double-strand break repair / nucleosome assembly / p53 binding / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.87 Å | ||||||
 データ登録者 データ登録者 | Ai, H.S. / Zebin, T. / Zhiheng, D. / Jiakun, T. / Liying, Z. / Jia-Bin, L. / Man, P. / Liu, L. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: Chem / 年: 2023 ジャーナル: Chem / 年: 2023タイトル: Synthetic E2-Ub-nucleosome conjugates for studying nucleosome ubiquitination. 著者: Ai, H.S. / Tong, Z. / Deng, Z. / Tian, J. / Zhang, L. / Sun, M. / Du, Y. / Xu, Z. / Shi, Q. / Liang, L. / Zheng, Q. / Li, J.B. / Pan, M. / Liu, L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8grq.cif.gz 8grq.cif.gz | 353.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8grq.ent.gz pdb8grq.ent.gz | 266.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8grq.json.gz 8grq.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8grq_validation.pdf.gz 8grq_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8grq_full_validation.pdf.gz 8grq_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  8grq_validation.xml.gz 8grq_validation.xml.gz | 46.6 KB | 表示 | |
| CIF形式データ |  8grq_validation.cif.gz 8grq_validation.cif.gz | 71.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gr/8grq https://data.pdbj.org/pub/pdb/validation_reports/gr/8grq ftp://data.pdbj.org/pub/pdb/validation_reports/gr/8grq ftp://data.pdbj.org/pub/pdb/validation_reports/gr/8grq | HTTPS FTP |
-関連構造データ
| 関連構造データ | 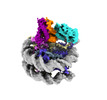 34212MC  8grmC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 7種, 11分子 AEBFCGDHKMN
| #1: タンパク質 | 分子量: 11530.447 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CALMAC_LOCUS17614 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CALMAC_LOCUS17614 / 発現宿主:  #2: タンパク質 | 分子量: 9123.692 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: Dana\GF27365, Dana_GF27365, GF27365 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: Dana\GF27365, Dana_GF27365, GF27365 / 発現宿主:  #3: タンパク質 | 分子量: 12066.128 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: H2ac12, Hist1h2ah / 発現宿主: Homo sapiens (ヒト) / 遺伝子: H2ac12, Hist1h2ah / 発現宿主:  #4: タンパク質 | 分子量: 20937.998 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  #7: タンパク質 | | 分子量: 10626.693 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: BRCA1, RNF53 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: BRCA1, RNF53 / 発現宿主:  #8: タンパク質 | | 分子量: 10348.078 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: BARD1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: BARD1 / 発現宿主:  #9: タンパク質 | | 分子量: 16657.938 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: UBE2D3, hCG_2028213 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: UBE2D3, hCG_2028213 / 発現宿主:  |
|---|
-DNA鎖 , 2種, 2分子 IJ
| #5: DNA鎖 | 分子量: 45604.047 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  |
|---|---|
| #6: DNA鎖 | 分子量: 45145.754 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  |
-非ポリマー , 1種, 4分子 
| #10: 化合物 | ChemComp-ZN / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex of BRCA1/BARD1 and H2AK127-UbcH5c-Ub nucleosome タイプ: COMPLEX / Entity ID: #9, #1-#3, #5-#6, #4 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.87 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 68006 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj
















































