+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8ejl | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
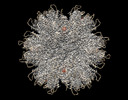
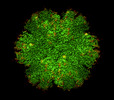
| タイトル | Structure of HIV-1 capsid declination in complex with CPSF6-FG peptide | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | VIRAL PROTEIN / HIV-1 / capsid / declination / pentamer / hexamer | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exon-exon junction complex binding / : / positive regulation of RNA export from nucleus / mRNA cleavage factor complex / interchromatin granule / Processing of Intronless Pre-mRNAs / perichromatin fibrils / mRNA cleavage and polyadenylation specificity factor complex / mRNA alternative polyadenylation / paraspeckles ...exon-exon junction complex binding / : / positive regulation of RNA export from nucleus / mRNA cleavage factor complex / interchromatin granule / Processing of Intronless Pre-mRNAs / perichromatin fibrils / mRNA cleavage and polyadenylation specificity factor complex / mRNA alternative polyadenylation / paraspeckles / mRNA 3'-end processing / mRNA 3'-end processing / Signaling by cytosolic FGFR1 fusion mutants / RNA Polymerase II Transcription Termination / protein heterotetramerization / viral budding via host ESCRT complex / ribosomal large subunit binding / Processing of Capped Intron-Containing Pre-mRNA / localization / Signaling by FGFR1 in disease / protein tetramerization / mRNA processing / ISG15 antiviral mechanism / host multivesicular body / viral nucleocapsid / nuclear speck / ribonucleoprotein complex / mRNA binding / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / RNA binding / zinc ion binding / nucleoplasm / membrane / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Pornillos, O. / Ganser-Pornillos, B.K. / Schirra, R.T. / dos Santos, N.F.B. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: A molecular switch modulates assembly and host factor binding of the HIV-1 capsid. 著者: Randall T Schirra / Nayara F B Dos Santos / Kaneil K Zadrozny / Iga Kucharska / Barbie K Ganser-Pornillos / Owen Pornillos /   要旨: The HIV-1 capsid is a fullerene cone made of quasi-equivalent hexamers and pentamers of the viral CA protein. Typically, quasi-equivalent assembly of viral capsid subunits is controlled by a ...The HIV-1 capsid is a fullerene cone made of quasi-equivalent hexamers and pentamers of the viral CA protein. Typically, quasi-equivalent assembly of viral capsid subunits is controlled by a molecular switch. Here, we identify a Thr-Val-Gly-Gly motif that modulates CA hexamer/pentamer switching by folding into a 3 helix in the pentamer and random coil in the hexamer. Manipulating the coil/helix configuration of the motif allowed us to control pentamer and hexamer formation in a predictable manner, thus proving its function as a molecular switch. Importantly, the switch also remodels the common binding site for host factors that are critical for viral replication and the new ultra-potent HIV-1 inhibitor lenacapavir. This study reveals that a critical assembly element also modulates the post-assembly and viral replication functions of the HIV-1 capsid and provides new insights on capsid function and inhibition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8ejl.cif.gz 8ejl.cif.gz | 116 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8ejl.ent.gz pdb8ejl.ent.gz | 86.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8ejl.json.gz 8ejl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8ejl_validation.pdf.gz 8ejl_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8ejl_full_validation.pdf.gz 8ejl_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  8ejl_validation.xml.gz 8ejl_validation.xml.gz | 32.5 KB | 表示 | |
| CIF形式データ |  8ejl_validation.cif.gz 8ejl_validation.cif.gz | 46.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ej/8ejl https://data.pdbj.org/pub/pdb/validation_reports/ej/8ejl ftp://data.pdbj.org/pub/pdb/validation_reports/ej/8ejl ftp://data.pdbj.org/pub/pdb/validation_reports/ej/8ejl | HTTPS FTP |
-関連構造データ
| 関連構造データ |  28186MC  7urnC  7urtC  8eepC  8eetC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 | x 5
|
| 2 |
|
| 3 | 
|
| 対称性 | 点対称性: (シェーンフリース記号: C5 (5回回転対称)) |
- 要素
要素
| #1: タンパク質 | 分子量: 25630.426 Da / 分子数: 4 / 由来タイプ: 組換発現 由来: (組換発現)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: NL4-3 / 遺伝子: Gag-pol / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 1708.952 Da / 分子数: 2 / Fragment: UNP residues 313-327 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) / 参照: UniProt: Q16630 Homo sapiens (ヒト) / 参照: UniProt: Q16630 |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Capsid-like particles of in vitro assembled HIV-1 CA protein in complex with CPSF6-FG peptide タイプ: COMPLEX 詳細: Capsids (0.27 mM CA) were incubated with peptide (3 mM) for 1 hour in ice Entity ID: all / 由来: MULTIPLE SOURCES |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 由来(組換発現) | 生物種:  |
| ウイルスについての詳細 | 中空か: YES / エンベロープを持つか: NO / 単離: OTHER / タイプ: VIRUS-LIKE PARTICLE |
| 緩衝液 | pH: 6 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE / 詳細: Manual plunge-freezing |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 500 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.20.1_4487: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 166463 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1 / Source name: PDB / タイプ: experimental model
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj





