+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8b1t | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RecBCD-DNA in complex with the phage protein Abc2 | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN / Homologous recombination / DNA repair / phage / Helicase / Nuclease / Inhibitor / Protein complex / Enzyme / DNA mimic | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exodeoxyribonuclease V / exodeoxyribonuclease V activity / exodeoxyribonuclease V complex / clearance of foreign intracellular DNA / DNA 5'-3' helicase / single-stranded DNA helicase activity / DNA 3'-5' helicase / recombinational repair / 3'-5' DNA helicase activity / DNA helicase activity ...exodeoxyribonuclease V / exodeoxyribonuclease V activity / exodeoxyribonuclease V complex / clearance of foreign intracellular DNA / DNA 5'-3' helicase / single-stranded DNA helicase activity / DNA 3'-5' helicase / recombinational repair / 3'-5' DNA helicase activity / DNA helicase activity / isomerase activity / helicase activity / double-strand break repair via homologous recombination / response to radiation / 5'-3' DNA helicase activity / DNA recombination / DNA damage response / magnesium ion binding / ATP hydrolysis activity / DNA binding / ATP binding / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Salmonella phage P22 (ファージ) Salmonella phage P22 (ファージ)synthetic construct (人工物) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Wilkinson, M. / Wilkinson, O.J. / Feyerherm, C. / Fletcher, E.E. / Wigley, D.B. / Dillingham, M.S. | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Structures of RecBCD in complex with phage-encoded inhibitor proteins reveal distinctive strategies for evasion of a bacterial immunity hub. 著者: Martin Wilkinson / Oliver J Wilkinson / Connie Feyerherm / Emma E Fletcher / Dale B Wigley / Mark S Dillingham /  要旨: Following infection of bacterial cells, bacteriophage modulate double-stranded DNA break repair pathways to protect themselves from host immunity systems and prioritise their own recombinases. Here, ...Following infection of bacterial cells, bacteriophage modulate double-stranded DNA break repair pathways to protect themselves from host immunity systems and prioritise their own recombinases. Here, we present biochemical and structural analysis of two phage proteins, gp5.9 and Abc2, which target the DNA break resection complex RecBCD. These exemplify two contrasting mechanisms for control of DNA break repair in which the RecBCD complex is either inhibited or co-opted for the benefit of the invading phage. Gp5.9 completely inhibits RecBCD by preventing it from binding to DNA. The RecBCD-gp5.9 structure shows that gp5.9 acts by substrate mimicry, binding predominantly to the RecB arm domain and competing sterically for the DNA binding site. Gp5.9 adopts a parallel coiled-coil architecture that is unprecedented for a natural DNA mimic protein. In contrast, binding of Abc2 does not substantially affect the biochemical activities of isolated RecBCD. The RecBCD-Abc2 structure shows that Abc2 binds to the Chi-recognition domains of the RecC subunit in a position that might enable it to mediate the loading of phage recombinases onto its single-stranded DNA products. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8b1t.cif.gz 8b1t.cif.gz | 543.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8b1t.ent.gz pdb8b1t.ent.gz | 431 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8b1t.json.gz 8b1t.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8b1t_validation.pdf.gz 8b1t_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8b1t_full_validation.pdf.gz 8b1t_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  8b1t_validation.xml.gz 8b1t_validation.xml.gz | 79.8 KB | 表示 | |
| CIF形式データ |  8b1t_validation.cif.gz 8b1t_validation.cif.gz | 120.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b1/8b1t https://data.pdbj.org/pub/pdb/validation_reports/b1/8b1t ftp://data.pdbj.org/pub/pdb/validation_reports/b1/8b1t ftp://data.pdbj.org/pub/pdb/validation_reports/b1/8b1t | HTTPS FTP |
-関連構造データ
| 関連構造データ | 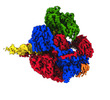 15804MC  8b1rC  8b1uC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-RecBCD enzyme subunit ... , 3種, 3分子 BCD
| #1: タンパク質 | 分子量: 134066.625 Da / 分子数: 1 / 変異: D1080A / 由来タイプ: 組換発現 / 詳細: Nuclease-dead mutant D1080A / 由来: (組換発現)  遺伝子: recB, BHS81_16925, BJI68_00835, BON89_01565, BON93_20715, BvCmsHHP056_02020, C5N07_06355, C5Y87_19730, C9114_10810, DAH36_05180, DAH37_01130, E2134_07940, E5M02_13555, E5P30_22870, E5P31_ ...遺伝子: recB, BHS81_16925, BJI68_00835, BON89_01565, BON93_20715, BvCmsHHP056_02020, C5N07_06355, C5Y87_19730, C9114_10810, DAH36_05180, DAH37_01130, E2134_07940, E5M02_13555, E5P30_22870, E5P31_10440, E5P32_13260, E5P36_03520, E5P40_11525, E5P51_08340, E5S39_06465, E5S44_07560, EI021_14360, ELX85_18530, EYV17_10125, EYV18_15060, F0L67_06605, F2N31_08115, F9V24_07045, FOI11_01100, FOI11_022080, GF699_18635, GKF89_09600, GRW05_08565, HMV95_04145, HX136_04845, IH772_15520, JNP96_21695, NCTC13216_00866, NCTC9117_01550, NCTC9706_03254, RG28_07470 プラスミド: pRSFduet / 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 128974.102 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
| #3: タンパク質 | 分子量: 66990.367 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
-DNA鎖 / タンパク質 , 2種, 2分子 XA
| #4: DNA鎖 | 分子量: 21440.707 Da / 分子数: 1 / 由来タイプ: 合成 詳細: Synthesised hairpin substrate with unpaired ssDNA tails 由来: (合成) synthetic construct (人工物) |
|---|---|
| #5: タンパク質 | 分子量: 11640.210 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Salmonella phage P22 (ファージ) / 遺伝子: abc2 / プラスミド: pET22b / 発現宿主: Salmonella phage P22 (ファージ) / 遺伝子: abc2 / プラスミド: pET22b / 発現宿主:  |
-非ポリマー , 2種, 2分子 


| #6: 化合物 | ChemComp-ANP / |
|---|---|
| #7: 化合物 | ChemComp-MG / |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | |||||||||||||||||||||||||||||||||||
| 由来(天然) |
| |||||||||||||||||||||||||||||||||||
| 由来(組換発現) |
| |||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | |||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 0.1 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: RecBCD-Abc2 mixed with 1.5x molar excess of synthesised DNA substrate, 2 mM ADPNP and 5 mM MgCl2 prior to making grids | |||||||||||||||||||||||||||||||||||
| 試料支持 | 詳細: Mixture of graphene oxide with 0.3 mM DDM detergent applied directly to grids twice before application of sample グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: C-flat-2/1 | |||||||||||||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 90 % / 詳細: 1.5s blot time |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2400 nm / 最小 デフォーカス(公称値): 1200 nm / Cs: 2.7 mm / C2レンズ絞り径: 50 µm |
| 試料ホルダ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 12 sec. / 電子線照射量: 56 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2500 |
| 電子光学装置 | エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 467613 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 119163 / 対称性のタイプ: POINT | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 72 / プロトコル: FLEXIBLE FIT / 空間: REAL / Target criteria: Correlation coefficient | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1 / Source name: PDB / タイプ: experimental model
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj


















































