+ データを開く
データを開く
- 基本情報
基本情報
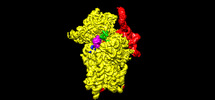
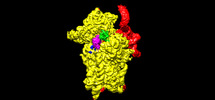
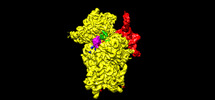
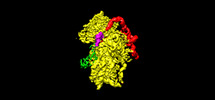
| 登録情報 | データベース: PDB / ID: 7syr | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
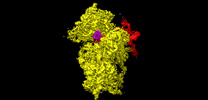
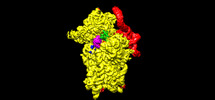
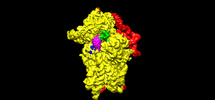
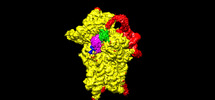
| タイトル | Structure of the wt IRES eIF2-containing 48S initiation complex, closed conformation. Structure 12(wt). | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | RIBOSOME / HCV / IRES / 40S | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of translation in response to endoplasmic reticulum stress / translation initiation ternary complex / glial limiting end-foot / response to kainic acid / Cellular response to mitochondrial stress / response to manganese-induced endoplasmic reticulum stress / positive regulation of type B pancreatic cell apoptotic process / negative regulation of translational initiation in response to stress / Response of EIF2AK1 (HRI) to heme deficiency / Recycling of eIF2:GDP ...regulation of translation in response to endoplasmic reticulum stress / translation initiation ternary complex / glial limiting end-foot / response to kainic acid / Cellular response to mitochondrial stress / response to manganese-induced endoplasmic reticulum stress / positive regulation of type B pancreatic cell apoptotic process / negative regulation of translational initiation in response to stress / Response of EIF2AK1 (HRI) to heme deficiency / Recycling of eIF2:GDP / PERK-mediated unfolded protein response / PERK regulates gene expression / eukaryotic translation initiation factor 2 complex / regulation of translational initiation in response to stress / multi-eIF complex / eukaryotic 43S preinitiation complex / translation factor activity, RNA binding / ribosomal subunit / eukaryotic 48S preinitiation complex / Formation of the ternary complex, and subsequently, the 43S complex / laminin receptor activity / Translation initiation complex formation / Ribosomal scanning and start codon recognition / mammalian oogenesis stage / activation-induced cell death of T cells / positive regulation of signal transduction by p53 class mediator / ubiquitin ligase inhibitor activity / Formation of a pool of free 40S subunits / phagocytic cup / Response of EIF2AK4 (GCN2) to amino acid deficiency / L13a-mediated translational silencing of Ceruloplasmin expression / GTP hydrolysis and joining of the 60S ribosomal subunit / endonucleolytic cleavage to generate mature 3'-end of SSU-rRNA from (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / TOR signaling / T cell proliferation involved in immune response / ribosomal small subunit export from nucleus / erythrocyte development / translation regulator activity / cytosolic ribosome / stress granule assembly / laminin binding / rough endoplasmic reticulum / endonucleolytic cleavage in ITS1 to separate SSU-rRNA from 5.8S rRNA and LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / gastrulation / MDM2/MDM4 family protein binding / translational initiation / translation initiation factor activity / cellular response to amino acid starvation / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / ribosome assembly / rescue of stalled ribosome / response to endoplasmic reticulum stress / 90S preribosome / maturation of SSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / cellular response to leukemia inhibitory factor / maturation of SSU-rRNA / small-subunit processome / positive regulation of apoptotic signaling pathway / protein kinase C binding / positive regulation of protein-containing complex assembly / placenta development / PKR-mediated signaling / ABC-family proteins mediated transport / cytoplasmic ribonucleoprotein granule / cytoplasmic stress granule / modification-dependent protein catabolic process / spindle / G1/S transition of mitotic cell cycle / rRNA processing / protein tag activity / cellular response to UV / rhythmic process / positive regulation of canonical Wnt signaling pathway / ribosome binding / glucose homeostasis / virus receptor activity / regulation of translation / ribosomal small subunit biogenesis / ribosomal small subunit assembly / cellular response to heat / small ribosomal subunit / small ribosomal subunit rRNA binding / cellular response to oxidative stress / cell body / T cell differentiation in thymus / perikaryon / cytosolic small ribosomal subunit / mitochondrial inner membrane / tRNA binding / cytoplasmic translation / postsynaptic density / cell differentiation / rRNA binding / ribosome / protein ubiquitination / structural constituent of ribosome / ribonucleoprotein complex / translation / positive regulation of protein phosphorylation 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) Hepatitis C virus (ウイルス) Hepatitis C virus (ウイルス) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||
 データ登録者 データ登録者 | Brown, Z.P. / Abaeva, I.S. / De, S. / Hellen, C.U.T. / Pestova, T.V. / Frank, J. | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2022 ジャーナル: EMBO J / 年: 2022タイトル: Molecular architecture of 40S translation initiation complexes on the hepatitis C virus IRES. 著者: Zuben P Brown / Irina S Abaeva / Swastik De / Christopher U T Hellen / Tatyana V Pestova / Joachim Frank /  要旨: Hepatitis C virus mRNA contains an internal ribosome entry site (IRES) that mediates end-independent translation initiation, requiring a subset of eukaryotic initiation factors (eIFs). Biochemical ...Hepatitis C virus mRNA contains an internal ribosome entry site (IRES) that mediates end-independent translation initiation, requiring a subset of eukaryotic initiation factors (eIFs). Biochemical studies revealed that direct binding of the IRES to the 40S ribosomal subunit places the initiation codon into the P site, where it base pairs with eIF2-bound Met-tRNAiMet forming a 48S initiation complex. Subsequently, eIF5 and eIF5B mediate subunit joining, yielding an elongation-competent 80S ribosome. Initiation can also proceed without eIF2, in which case Met-tRNAiMet is recruited directly by eIF5B. However, the structures of initiation complexes assembled on the HCV IRES, the transitions between different states, and the accompanying conformational changes have remained unknown. To fill these gaps, we now obtained cryo-EM structures of IRES initiation complexes, at resolutions up to 3.5 Å, that cover all major stages from the initial ribosomal association, through eIF2-containing 48S initiation complexes, to eIF5B-containing complexes immediately prior to subunit joining. These structures provide insights into the dynamic network of 40S/IRES contacts, highlight the role of IRES domain II, and reveal conformational changes that occur during the transition from eIF2- to eIF5B-containing 48S complexes and prepare them for subunit joining. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7syr.cif.gz 7syr.cif.gz | 1.8 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7syr.ent.gz pdb7syr.ent.gz | 1.4 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7syr.json.gz 7syr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7syr_validation.pdf.gz 7syr_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7syr_full_validation.pdf.gz 7syr_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  7syr_validation.xml.gz 7syr_validation.xml.gz | 162.9 KB | 表示 | |
| CIF形式データ |  7syr_validation.cif.gz 7syr_validation.cif.gz | 271 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/sy/7syr https://data.pdbj.org/pub/pdb/validation_reports/sy/7syr ftp://data.pdbj.org/pub/pdb/validation_reports/sy/7syr ftp://data.pdbj.org/pub/pdb/validation_reports/sy/7syr | HTTPS FTP |
-関連構造データ
| 関連構造データ |  25538MC  7syiC  7syjC  7sykC  7sylC  7syoC  7sypC  7syqC  7sysC  7sytC  7syuC  7syvC  7sywC  7syxC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-RNA鎖 , 3種, 3分子 2iz
| #1: RNA鎖 | 分子量: 603100.938 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #36: RNA鎖 | 分子量: 24231.510 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #39: RNA鎖 | 分子量: 128746.109 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Hepatitis C virus (isolate 1) (C型肝炎ウイルス) Hepatitis C virus (isolate 1) (C型肝炎ウイルス)発現宿主:  |
-Eukaryotic translation initiation factor ... , 2種, 2分子 Aj
| #2: タンパク質 | 分子量: 16488.449 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: EIF1AX, EIF1A, EIF4C / 発現宿主: Homo sapiens (ヒト) / 遺伝子: EIF1AX, EIF1A, EIF4C / 発現宿主:  |
|---|---|
| #37: タンパク質 | 分子量: 36161.180 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: EIF2S1, EIF2A / 発現宿主: Homo sapiens (ヒト) / 遺伝子: EIF2S1, EIF2A / 発現宿主:  |
+タンパク質 , 33種, 33分子 BCDEFGHIJKLMNOPQRSTUVWXYZabcde...
-タンパク質・ペプチド / 非ポリマー , 2種, 2分子 n

| #38: タンパク質・ペプチド | 分子量: 3473.451 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #40: 化合物 | ChemComp-ZN / |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: 40S ribosomal small subunit with HCV IRES / タイプ: RIBOSOME / Entity ID: #1-#39 / 由来: MULTIPLE SOURCES |
|---|---|
| 分子量 | 値: 2 MDa / 実験値: NO |
| 緩衝液 | pH: 7.5 |
| 試料 | 濃度: 7.5E-5 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: H2/O2 mixture for 25 seconds at 25W power / グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R0.6/1 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K / 詳細: 4 second blot time, force 3 |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TECNAI F30 |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 52000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs: 2.26 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN |
| 撮影 | 平均露光時間: 4 sec. / 電子線照射量: 70.9 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.18.2_3874: / 分類: 精密化 | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 46904 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー
























 PDBj
PDBj









































