+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6zwv | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
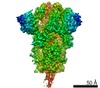
| タイトル | Cryo-EM structure of SARS-CoV-2 Spike Proteins on intact virions: 3 Closed RBDs | ||||||||||||||||||
 要素 要素 | Spike glycoprotein | ||||||||||||||||||
 キーワード キーワード | VIRAL PROTEIN / SARS-CoV-2 / Spike Protein / Virions / Glycoprotein | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / membrane fusion / Attachment and Entry / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Ke, Z. / Qu, K. / Nakane, T. / Xiong, X. / Cortese, M. / Zila, V. / Scheres, S.H.W. / Briggs, J.A.G. | ||||||||||||||||||
| 資金援助 | European Union,  英国, 英国,  ドイツ, ドイツ,  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structures and distributions of SARS-CoV-2 spike proteins on intact virions. 著者: Zunlong Ke / Joaquin Oton / Kun Qu / Mirko Cortese / Vojtech Zila / Lesley McKeane / Takanori Nakane / Jasenko Zivanov / Christopher J Neufeldt / Berati Cerikan / John M Lu / Julia Peukes / ...著者: Zunlong Ke / Joaquin Oton / Kun Qu / Mirko Cortese / Vojtech Zila / Lesley McKeane / Takanori Nakane / Jasenko Zivanov / Christopher J Neufeldt / Berati Cerikan / John M Lu / Julia Peukes / Xiaoli Xiong / Hans-Georg Kräusslich / Sjors H W Scheres / Ralf Bartenschlager / John A G Briggs /   要旨: Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) virions are surrounded by a lipid bilayer from which spike (S) protein trimers protrude. Heavily glycosylated S trimers bind to the ...Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) virions are surrounded by a lipid bilayer from which spike (S) protein trimers protrude. Heavily glycosylated S trimers bind to the angiotensin-converting enzyme 2 receptor and mediate entry of virions into target cells. S exhibits extensive conformational flexibility: it modulates exposure of its receptor-binding site and subsequently undergoes complete structural rearrangement to drive fusion of viral and cellular membranes. The structures and conformations of soluble, overexpressed, purified S proteins have been studied in detail using cryo-electron microscopy, but the structure and distribution of S on the virion surface remain unknown. Here we applied cryo-electron microscopy and tomography to image intact SARS-CoV-2 virions and determine the high-resolution structure, conformational flexibility and distribution of S trimers in situ on the virion surface. These results reveal the conformations of S on the virion, and provide a basis from which to understand interactions between S and neutralizing antibodies during infection or vaccination. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6zwv.cif.gz 6zwv.cif.gz | 546.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6zwv.ent.gz pdb6zwv.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  6zwv.json.gz 6zwv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zw/6zwv https://data.pdbj.org/pub/pdb/validation_reports/zw/6zwv ftp://data.pdbj.org/pub/pdb/validation_reports/zw/6zwv ftp://data.pdbj.org/pub/pdb/validation_reports/zw/6zwv | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  11497MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10492 (タイトル: Cryo-EM structure of SARS-CoV-2 Spike Proteins on intact virions EMPIAR-10492 (タイトル: Cryo-EM structure of SARS-CoV-2 Spike Proteins on intact virionsData size: 2.1 TB / Data #1: Unaligned movies [micrographs - multiframe]) |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 141239.391 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  参照: UniProt: P0DTC2 #2: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose #3: 糖 | ChemComp-NAG / 研究の焦点であるリガンドがあるか | N | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Severe acute respiratory syndrome coronavirus 2 / タイプ: VIRUS 詳細: sucrose-cushion purified SARS-CoV-2 virions produced from infected VeroE6 cells. Entity ID: #1 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  株: Germany/BavPat1/2020 |
| ウイルスについての詳細 | 中空か: NO / エンベロープを持つか: YES / 単離: STRAIN / タイプ: VIRION |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドのタイプ: C-flat-2/2 |
| 急速凍結 | 装置: LEICA EM GP / 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs: 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: ZEMLIN TABLEAU |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 2.7 sec. / 電子線照射量: 50 e/Å2 / 検出モード: COUNTING / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 7982 |
| 電子光学装置 | エネルギーフィルター名称: GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
| 画像スキャン | サンプリングサイズ: 5 µm / 横: 5760 / 縦: 4092 / 動画フレーム数/画像: 10 / 利用したフレーム数/画像: 1-10 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.18.2_3874: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 450473 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C3 (3回回転対称) | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 29183 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| EM volume selection | 手法: Manual Picked / Num. of tomograms: 156 / Num. of volumes extracted: 4104 / Reference model: Reference Free | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 6ZP0 Accession code: 6ZP0 / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj






