+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5kc2 | ||||||
|---|---|---|---|---|---|---|---|
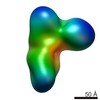
| タイトル | Negative stain structure of Vps15/Vps34 complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | ENDOCYTOSIS / autophagy / phosphatidylinositol 3-kinase (PtdIns3K) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Synthesis of PIPs at the early endosome membrane / Synthesis of PIPs at the late endosome membrane / RHO GTPases Activate NADPH Oxidases / nucleus-vacuole junction / Synthesis of PIPs at the Golgi membrane / vacuole-isolation membrane contact site / vacuole inheritance / phosphatidylinositol 3-kinase complex, class III, type II / phosphatidylinositol 3-kinase complex, class III, type I / Macroautophagy ...Synthesis of PIPs at the early endosome membrane / Synthesis of PIPs at the late endosome membrane / RHO GTPases Activate NADPH Oxidases / nucleus-vacuole junction / Synthesis of PIPs at the Golgi membrane / vacuole-isolation membrane contact site / vacuole inheritance / phosphatidylinositol 3-kinase complex, class III, type II / phosphatidylinositol 3-kinase complex, class III, type I / Macroautophagy / protein retention in Golgi apparatus / pexophagy / phagophore assembly site membrane / protein targeting to vacuole / late endosome to vacuole transport / fungal-type vacuole membrane / phagophore assembly site / phosphatidylinositol 3-kinase / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity / phosphatidylinositol-mediated signaling / phosphatidylinositol phosphate biosynthetic process / autophagosome assembly / ubiquitin binding / macroautophagy / positive regulation of transcription elongation by RNA polymerase II / autophagy / endocytosis / peroxisome / protein transport / late endosome / endosome membrane / non-specific serine/threonine protein kinase / protein kinase activity / endosome / phosphorylation / Golgi membrane / protein serine kinase activity / protein serine/threonine kinase activity / mitochondrion / ATP binding / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / ネガティブ染色法 / 解像度: 28 Å | ||||||
 データ登録者 データ登録者 | Kirsten, M.L. / Zhang, L. / Ohashi, Y. / Perisic, O. / Williams, R.L. / Sachse, C. | ||||||
 引用 引用 |  ジャーナル: Autophagy / 年: 2016 ジャーナル: Autophagy / 年: 2016タイトル: Characterization of Atg38 and NRBF2, a fifth subunit of the autophagic Vps34/PIK3C3 complex. 著者: Yohei Ohashi / Nicolas Soler / Miguel García Ortegón / Lufei Zhang / Marie L Kirsten / Olga Perisic / Glenn R Masson / John E Burke / Arjen J Jakobi / Apostolos A Apostolakis / Christopher ...著者: Yohei Ohashi / Nicolas Soler / Miguel García Ortegón / Lufei Zhang / Marie L Kirsten / Olga Perisic / Glenn R Masson / John E Burke / Arjen J Jakobi / Apostolos A Apostolakis / Christopher M Johnson / Maki Ohashi / Nicholas T Ktistakis / Carsten Sachse / Roger L Williams /   要旨: The phosphatidylinositol 3-kinase Vps34 is part of several protein complexes. The structural organization of heterotetrameric complexes is starting to emerge, but little is known about organization ...The phosphatidylinositol 3-kinase Vps34 is part of several protein complexes. The structural organization of heterotetrameric complexes is starting to emerge, but little is known about organization of additional accessory subunits that interact with these assemblies. Combining hydrogen-deuterium exchange mass spectrometry (HDX-MS), X-ray crystallography and electron microscopy (EM), we have characterized Atg38 and its human ortholog NRBF2, accessory components of complex I consisting of Vps15-Vps34-Vps30/Atg6-Atg14 (yeast) and PIK3R4/VPS15-PIK3C3/VPS34-BECN1/Beclin 1-ATG14 (human). HDX-MS shows that Atg38 binds the Vps30-Atg14 subcomplex of complex I, using mainly its N-terminal MIT domain and bridges the coiled-coil I regions of Atg14 and Vps30 in the base of complex I. The Atg38 C-terminal domain is important for localization to the phagophore assembly site (PAS) and homodimerization. Our 2.2 Å resolution crystal structure of the Atg38 C-terminal homodimerization domain shows 2 segments of α-helices assembling into a mushroom-like asymmetric homodimer with a 4-helix cap and a parallel coiled-coil stalk. One Atg38 homodimer engages a single complex I. This is in sharp contrast to human NRBF2, which also forms a homodimer, but this homodimer can bridge 2 complex I assemblies. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5kc2.cif.gz 5kc2.cif.gz | 324.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5kc2.ent.gz pdb5kc2.ent.gz | 218 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5kc2.json.gz 5kc2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5kc2_validation.pdf.gz 5kc2_validation.pdf.gz | 621.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5kc2_full_validation.pdf.gz 5kc2_full_validation.pdf.gz | 622.8 KB | 表示 | |
| XML形式データ |  5kc2_validation.xml.gz 5kc2_validation.xml.gz | 41.7 KB | 表示 | |
| CIF形式データ |  5kc2_validation.cif.gz 5kc2_validation.cif.gz | 68.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kc/5kc2 https://data.pdbj.org/pub/pdb/validation_reports/kc/5kc2 ftp://data.pdbj.org/pub/pdb/validation_reports/kc/5kc2 ftp://data.pdbj.org/pub/pdb/validation_reports/kc/5kc2 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 101047.180 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: VPS34, END12, PEP15, VPL7, VPT29, YLR240W, L9672.10 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 166550.000 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: VPS15, GRD8, VAC4, VPL19, YBR097W, YBR0825 / 発現宿主:  参照: UniProt: P22219, non-specific serine/threonine protein kinase |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Vps15/34 / タイプ: COMPLEX 詳細: Subcomplex of Vps34 phosphatidylinositol 3-kinase (PtdIns3K) complex I (yeast) Entity ID: all / 由来: RECOMBINANT | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) | 生物種:  | ||||||||||||||||||||
| 由来(組換発現) | 生物種:  | ||||||||||||||||||||
| 緩衝液 | pH: 8.8 | ||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||
| 試料 | 濃度: 5 mg/ml / 包埋: NO / シャドウイング: NO / 染色: YES / 凍結: NO | ||||||||||||||||||||
| 染色 | タイプ: NEGATIVE / 詳細: droplet technique / 染色剤: Uranyl acetate | ||||||||||||||||||||
| 試料支持 |
|
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI POLARA 300 |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 1000 nm |
| 試料ホルダ | 凍結剤: NITROGEN / 試料ホルダーモデル: OTHER |
| 撮影 | 平均露光時間: 1 sec. / 電子線照射量: 20 e/Å2 フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 撮影したグリッド数: 2 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING ONLY | ||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 14172 | ||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 28 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 14172 / アルゴリズム: BACK PROJECTION / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




