+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8886 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of F-actin complexed with the beta-III-spectrin actin-binding domain | ||||||||||||
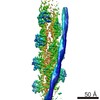
 マップデータ マップデータ | Cryo-EM structure of F-actin complexed with the beta-III-spectrin actin-binding domain | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | actin binding protein / filament / STRUCTURAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cerebellar Purkinje cell layer morphogenesis / structural constituent of synapse / postsynaptic spectrin-associated cytoskeleton / structural constituent of postsynapse / regulation of postsynaptic specialization assembly / spectrin / positive regulation of norepinephrine uptake / cellular response to cytochalasin B / regulation of transepithelial transport / paranodal junction ...cerebellar Purkinje cell layer morphogenesis / structural constituent of synapse / postsynaptic spectrin-associated cytoskeleton / structural constituent of postsynapse / regulation of postsynaptic specialization assembly / spectrin / positive regulation of norepinephrine uptake / cellular response to cytochalasin B / regulation of transepithelial transport / paranodal junction / morphogenesis of a polarized epithelium / bBAF complex / postsynaptic actin cytoskeleton organization / npBAF complex / protein localization to adherens junction / nBAF complex / postsynaptic actin cytoskeleton / brahma complex / Tat protein binding / structural constituent of postsynaptic actin cytoskeleton / GBAF complex / regulation of G0 to G1 transition / Formation of annular gap junctions / dense body / Gap junction degradation / Cell-extracellular matrix interactions / Folding of actin by CCT/TriC / apical protein localization / regulation of double-strand break repair / actin filament capping / regulation of nucleotide-excision repair / adherens junction assembly / RSC-type complex / Prefoldin mediated transfer of substrate to CCT/TriC / RHOF GTPase cycle / Adherens junctions interactions / tight junction / parallel fiber to Purkinje cell synapse / regulation of mitotic metaphase/anaphase transition / Interaction between L1 and Ankyrins / Sensory processing of sound by outer hair cells of the cochlea / Sensory processing of sound by inner hair cells of the cochlea / SWI/SNF complex / regulation of norepinephrine uptake / positive regulation of double-strand break repair / positive regulation of T cell differentiation / regulation of synaptic vesicle endocytosis / apical junction complex / establishment or maintenance of cell polarity / cortical actin cytoskeleton / regulation of cyclin-dependent protein serine/threonine kinase activity / adult behavior / cortical cytoskeleton / maintenance of blood-brain barrier / NuA4 histone acetyltransferase complex / positive regulation of stem cell population maintenance / nitric-oxide synthase binding / Regulation of MITF-M-dependent genes involved in pigmentation / regulation of G1/S transition of mitotic cell cycle / Recycling pathway of L1 / brush border / kinesin binding / negative regulation of cell differentiation / calyx of Held / positive regulation of double-strand break repair via homologous recombination / EPH-ephrin mediated repulsion of cells / positive regulation of myoblast differentiation / regulation of protein localization to plasma membrane / RHO GTPases Activate WASPs and WAVEs / RHO GTPases activate IQGAPs / COPI-mediated anterograde transport / vesicle-mediated transport / synapse assembly / substantia nigra development / MHC class II antigen presentation / EPHB-mediated forward signaling / NCAM signaling for neurite out-growth / axonogenesis / Regulation of endogenous retroelements by Piwi-interacting RNAs (piRNAs) / platelet aggregation / negative regulation of protein binding / cell projection / cell motility / Translocation of SLC2A4 (GLUT4) to the plasma membrane / actin filament / positive regulation of cell differentiation / RHO GTPases Activate Formins / adherens junction / FCGR3A-mediated phagocytosis / regulation of transmembrane transporter activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / DNA Damage Recognition in GG-NER / tau protein binding / multicellular organism growth / B-WICH complex positively regulates rRNA expression / Schaffer collateral - CA1 synapse / phospholipid binding / structural constituent of cytoskeleton 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 7.0 Å | ||||||||||||
 データ登録者 データ登録者 | Wang F / Orlova A | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Structural basis for high-affinity actin binding revealed by a β-III-spectrin SCA5 missense mutation. 著者: Adam W Avery / Michael E Fealey / Fengbin Wang / Albina Orlova / Andrew R Thompson / David D Thomas / Thomas S Hays / Edward H Egelman /  要旨: Spinocerebellar ataxia type 5 (SCA5) is a neurodegenerative disease caused by mutations in the cytoskeletal protein β-III-spectrin. Previously, a SCA5 mutation resulting in a leucine-to-proline ...Spinocerebellar ataxia type 5 (SCA5) is a neurodegenerative disease caused by mutations in the cytoskeletal protein β-III-spectrin. Previously, a SCA5 mutation resulting in a leucine-to-proline substitution (L253P) in the actin-binding domain (ABD) was shown to cause a 1000-fold increase in actin-binding affinity. However, the structural basis for this increase is unknown. Here, we report a 6.9 Å cryo-EM structure of F-actin complexed with the L253P ABD. This structure, along with co-sedimentation and pulsed-EPR measurements, demonstrates that high-affinity binding caused by the CH2-localized mutation is due to opening of the two CH domains. This enables CH1 to bind actin aided by an unstructured N-terminal region that becomes α-helical upon binding. This helix is required for association with actin as truncation eliminates binding. Collectively, these results shed light on the mechanism by which β-III-spectrin, and likely similar actin-binding proteins, interact with actin, and how this mechanism can be perturbed to cause disease. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8886.map.gz emd_8886.map.gz | 24.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8886-v30.xml emd-8886-v30.xml emd-8886.xml emd-8886.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8886.png emd_8886.png | 180.3 KB | ||
| Filedesc metadata |  emd-8886.cif.gz emd-8886.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8886 http://ftp.pdbj.org/pub/emdb/structures/EMD-8886 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8886 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8886 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8886_validation.pdf.gz emd_8886_validation.pdf.gz | 467.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8886_full_validation.pdf.gz emd_8886_full_validation.pdf.gz | 467.5 KB | 表示 | |
| XML形式データ |  emd_8886_validation.xml.gz emd_8886_validation.xml.gz | 6 KB | 表示 | |
| CIF形式データ |  emd_8886_validation.cif.gz emd_8886_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8886 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8886 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8886 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8886 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8886.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8886.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of F-actin complexed with the beta-III-spectrin actin-binding domain | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : F-actin complexed with the spectrin actin-binding domain
| 全体 | 名称: F-actin complexed with the spectrin actin-binding domain |
|---|---|
| 要素 |
|
-超分子 #1: F-actin complexed with the spectrin actin-binding domain
| 超分子 | 名称: F-actin complexed with the spectrin actin-binding domain タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Actin, cytoplasmic 1
| 分子 | 名称: Actin, cytoplasmic 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 41.78266 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDDDIAALVV DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IEHGIVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP LNPKANREKM TQIMFETFNT PAMYVAIQAV LSLYASGRTT GIVMDSGDGV T HTVPIYEG ...文字列: MDDDIAALVV DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IEHGIVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP LNPKANREKM TQIMFETFNT PAMYVAIQAV LSLYASGRTT GIVMDSGDGV T HTVPIYEG YALPHAILRL DLAGRDLTDY LMKILTERGY SFTTTAEREI VRDIKEKLCY VALDFEQEMA TAASSSSLEK SY ELPDGQV ITIGNERFRC PEALFQPSFL GMESCGIHET TFNSIMKCDV DIRKDLYANT VLSGGTTMYP GIADRMQKEI TAL APSTMK IKIIAPPERK YSVWIGGSIL ASLSTFQQMW ISKQEYDESG PSIVHRKCF UniProtKB: Actin, cytoplasmic 1 |
-分子 #2: Spectrin beta chain, non-erythrocytic 2
| 分子 | 名称: Spectrin beta chain, non-erythrocytic 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.857141 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSTLSPTDF DSLEIQGQYS DINNRWDLPD SDWDNDSSSA RLFERSRIKA LADEREAVQK KTFTKWVNSH LARVTCRVGD LYSDLRDGR NLLRLLEVLS GEILPKPTKG RMRIHCLENV DKALQFLKEQ KVHLENMGSH DIVDGNHRLT LGLVWTIILR F QIQDISVE ...文字列: MSSTLSPTDF DSLEIQGQYS DINNRWDLPD SDWDNDSSSA RLFERSRIKA LADEREAVQK KTFTKWVNSH LARVTCRVGD LYSDLRDGR NLLRLLEVLS GEILPKPTKG RMRIHCLENV DKALQFLKEQ KVHLENMGSH DIVDGNHRLT LGLVWTIILR F QIQDISVE TEDNKEKKSA KDALLLWCQM KTAGYPNVNV HNFTTSWRDG LAFNAIVHKH RPDLLDFESL KKCNAHYNLQ NA FNLAEKE LGLTKPLDPE DVNVDQPDEK SIITYVATYY HYFSKMK UniProtKB: Spectrin beta chain, non-erythrocytic 2 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: negative stain |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 平均露光時間: 3.0 sec. / 平均電子線量: 20.0 e/Å2 詳細: Images were stored containing seven parts, where each part represented a set of frames corresponding to a dose of ~20 electrons per Angstrom^2. The full dose image stack was used for the ...詳細: Images were stored containing seven parts, where each part represented a set of frames corresponding to a dose of ~20 electrons per Angstrom^2. The full dose image stack was used for the estimation of the CTF as well as for boxing filaments. Only the first two parts were used for the reconstruction (~5 electrons per Angstrom^2). |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 27.25 Å 想定した対称性 - らせんパラメータ - ΔΦ: -166.87 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 7.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER / 詳細: model-map FSC 0.38 cut-off / 使用した粒子像数: 12443 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER / 詳細: low resolution pure actin filament map |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: SPIDER |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6anu: |
 ムービー
ムービー コントローラー
コントローラー





































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















