+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8756 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast microtubule stabilized with epothilone | ||||||||||||||||||||||||
 マップデータ マップデータ | Yeast microtubule stabilized with epothilone | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | Cytoskeleton / tubulin / HYDROLASE | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear migration by microtubule mediated pushing forces / nuclear division / mitotic spindle elongation / nuclear migration along microtubule / homologous chromosome segregation / Platelet degranulation / positive regulation of intracellular protein transport / tubulin complex / mitotic sister chromatid segregation / microtubule-based process ...nuclear migration by microtubule mediated pushing forces / nuclear division / mitotic spindle elongation / nuclear migration along microtubule / homologous chromosome segregation / Platelet degranulation / positive regulation of intracellular protein transport / tubulin complex / mitotic sister chromatid segregation / microtubule-based process / mitotic spindle assembly / cytoplasmic microtubule organization / cytoskeleton organization / nuclear periphery / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / structural constituent of cytoskeleton / microtubule cytoskeleton organization / spindle / mitotic cell cycle / microtubule / hydrolase activity / response to antibiotic / GTPase activity / GTP binding / nucleus / metal ion binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   | ||||||||||||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Howes SC / Geyer EA | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2017 ジャーナル: J Cell Biol / 年: 2017タイトル: Structural differences between yeast and mammalian microtubules revealed by cryo-EM. 著者: Stuart C Howes / Elisabeth A Geyer / Benjamin LaFrance / Rui Zhang / Elizabeth H Kellogg / Stefan Westermann / Luke M Rice / Eva Nogales /   要旨: Microtubules are polymers of αβ-tubulin heterodimers essential for all eukaryotes. Despite sequence conservation, there are significant structural differences between microtubules assembled in ...Microtubules are polymers of αβ-tubulin heterodimers essential for all eukaryotes. Despite sequence conservation, there are significant structural differences between microtubules assembled in vitro from mammalian or budding yeast tubulin. Yeast MTs were not observed to undergo compaction at the interdimer interface as seen for mammalian microtubules upon GTP hydrolysis. Lack of compaction might reflect slower GTP hydrolysis or a different degree of allosteric coupling in the lattice. The microtubule plus end-tracking protein Bim1 binds yeast microtubules both between αβ-tubulin heterodimers, as seen for other organisms, and within tubulin dimers, but binds mammalian tubulin only at interdimer contacts. At the concentrations used in cryo-electron microscopy, Bim1 causes the compaction of yeast microtubules and induces their rapid disassembly. Our studies demonstrate structural differences between yeast and mammalian microtubules that likely underlie their differing polymerization dynamics. These differences may reflect adaptations to the demands of different cell size or range of physiological growth temperatures. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8756.map.gz emd_8756.map.gz | 482.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8756-v30.xml emd-8756-v30.xml emd-8756.xml emd-8756.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8756.png emd_8756.png | 120.8 KB | ||
| Filedesc metadata |  emd-8756.cif.gz emd-8756.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8756 http://ftp.pdbj.org/pub/emdb/structures/EMD-8756 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8756 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8756 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8756_validation.pdf.gz emd_8756_validation.pdf.gz | 573.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8756_full_validation.pdf.gz emd_8756_full_validation.pdf.gz | 572.8 KB | 表示 | |
| XML形式データ |  emd_8756_validation.xml.gz emd_8756_validation.xml.gz | 8.3 KB | 表示 | |
| CIF形式データ |  emd_8756_validation.cif.gz emd_8756_validation.cif.gz | 9.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8756 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8756 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8756 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8756 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8756.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8756.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Yeast microtubule stabilized with epothilone | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Microtubule lattice stabilized with epothilone
| 全体 | 名称: Microtubule lattice stabilized with epothilone |
|---|---|
| 要素 |
|
-超分子 #1: Microtubule lattice stabilized with epothilone
| 超分子 | 名称: Microtubule lattice stabilized with epothilone / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Tubulin alpha-1 chain
| 分子 | 名称: Tubulin alpha-1 chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 49.853867 KDa |
| 配列 | 文字列: MREVISINVG QAGCQIGNAC WELYSLEHGI KPDGHLEDGL SKPKGGEEGF STFFHETGYG KFVPRAIYVD LEPNVIDEVR NGPYKDLFH PEQLISGKED AANNYARGHY TVGREILGDV LDRIRKLADQ CDGLQGFLFT HSLGGGTGSG LGSLLLEELS A EYGKKSKL ...文字列: MREVISINVG QAGCQIGNAC WELYSLEHGI KPDGHLEDGL SKPKGGEEGF STFFHETGYG KFVPRAIYVD LEPNVIDEVR NGPYKDLFH PEQLISGKED AANNYARGHY TVGREILGDV LDRIRKLADQ CDGLQGFLFT HSLGGGTGSG LGSLLLEELS A EYGKKSKL EFAVYPAPQV STSVVEPYNT VLTTHTTLEH ADCTFMVDNE AIYDMCKRNL DIPRPSFANL NNLIAQVVSS VT ASLRFDG SLNVDLNEFQ TNLVPYPRIH FPLVSYSPVL SKSKAFHESN SVSEITNACF EPGNQMVKCD PRDGKYMATC LLY RGDVVT RDVQRAVEQV KNKKTVQLVD WCPTGFKIGI CYEPPTATPN SQLATVDRAV CMLSNTTSIA EAWKRIDRKF DLMY AKRAF VHWYVGEGME EGEFTEARED LAALERDYIE VGADSYAEEE EF UniProtKB: Tubulin alpha-1 chain |
-分子 #2: Tubulin beta chain
| 分子 | 名称: Tubulin beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 50.967457 KDa |
| 配列 | 文字列: MREIIHISTG QCGNQIGAAF WETICGEHGL DFNGTYHGHD DIQKERLNVY FNEASSGKWV PRSINVDLEP GTIDAVRNSA IGNLFRPDN YIFGQSSAGN VWAKGHYTEG AELVDSVMDV IRREAEGCDS LQGFQITHSL GGGTGSGMGT LLISKIREEF P DRMMATFS ...文字列: MREIIHISTG QCGNQIGAAF WETICGEHGL DFNGTYHGHD DIQKERLNVY FNEASSGKWV PRSINVDLEP GTIDAVRNSA IGNLFRPDN YIFGQSSAGN VWAKGHYTEG AELVDSVMDV IRREAEGCDS LQGFQITHSL GGGTGSGMGT LLISKIREEF P DRMMATFS VLPSPKTSDT VVEPYNATLS VHQLVEHSDE TFCIDNEALY DICQRTLKLN QPSYGDLNNL VSSVMSGVTT SL RYPGQLN SDLRKLAVNL VPFPRLHFFM VGYAPLTAIG SQSFRSLTVP ELTQQMFDAK NMMAAADPRN GRYLTVAAFF RGK VSVKEV EDEMHKVQSK NSDYFVEWIP NNVQTAVCSV APQGLDMAAT FIANSTSIQE LFKRVGDQFS AMFKRKAFLH WYTS EGMDE LEFSEAESNM NDLVSEYQQY QEATVEDDEE VDENGDFGAP QNQDEPITEN FE UniProtKB: Tubulin beta chain |
-分子 #3: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #6: EPOTHILONE A
| 分子 | 名称: EPOTHILONE A / タイプ: ligand / ID: 6 / コピー数: 1 / 式: EP |
|---|---|
| 分子量 | 理論値: 493.656 Da |
| Chemical component information | 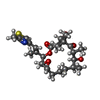 ChemComp-EP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.9 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 303 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 28.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 10.43 Å 想定した対称性 - らせんパラメータ - ΔΦ: -29.87 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 4.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIGN (ver. 9.11) / 使用した粒子像数: 27829 |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー


















