+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Active SV40 LTAg complex with DNA (3D variability component_001, frame_019). | |||||||||
 マップデータ マップデータ | Volume series map from continuous heterogeneity analysis postprocessed by EMReady. Frame_019 (of frames _000 thru _019) along principal component_001 (of components _000, 001, & _002). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ superfamily Helicase Substrate translocation DNA unwinding / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host JAK-STAT cascade via inhibition of JAK1 activity / bidirectional double-stranded viral DNA replication / viral DNA genome replication / DNA 3'-5' helicase / symbiont-mediated perturbation of host cell cycle G1/S transition checkpoint / DNA replication origin binding / helicase activity / single-stranded DNA binding / double-stranded DNA binding / symbiont-mediated perturbation of host ubiquitin-like protein modification ...symbiont-mediated suppression of host JAK-STAT cascade via inhibition of JAK1 activity / bidirectional double-stranded viral DNA replication / viral DNA genome replication / DNA 3'-5' helicase / symbiont-mediated perturbation of host cell cycle G1/S transition checkpoint / DNA replication origin binding / helicase activity / single-stranded DNA binding / double-stranded DNA binding / symbiont-mediated perturbation of host ubiquitin-like protein modification / DNA replication / symbiont-mediated suppression of host innate immune response / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / ATP hydrolysis activity / zinc ion binding / ATP binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Betapolyomavirus macacae (ウイルス) / synthetic construct (人工物) Betapolyomavirus macacae (ウイルス) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Shahid T | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2025 ジャーナル: Nature / 年: 2025タイトル: Structural dynamics of DNA unwinding by a replicative helicase. 著者: Taha Shahid / Ammar U Danazumi / Muhammad Tehseen / Lubna Alhudhali / Alice R Clark / Christos G Savva / Samir M Hamdan / Alfredo De Biasio /   要旨: Hexameric helicases are nucleotide-driven molecular machines that unwind DNA to initiate replication across all domains of life. Despite decades of intensive study, several critical aspects of their ...Hexameric helicases are nucleotide-driven molecular machines that unwind DNA to initiate replication across all domains of life. Despite decades of intensive study, several critical aspects of their function remain unresolved: the site and mechanism of DNA strand separation, the mechanics of unwinding propagation, and the dynamic relationship between nucleotide hydrolysis and DNA movement. Here, using cryo-electron microscopy (cryo-EM), we show that the simian virus 40 large tumour antigen (LTag) helicase assembles in the form of head-to-head hexamers at replication origins, melting DNA at two symmetrically positioned sites to establish bidirectional replication forks. Through continuous heterogeneity analysis, we characterize the conformational landscape of LTag on forked DNA under catalytic conditions, demonstrating coordinated motions that drive DNA translocation and unwinding. We show that the helicase pulls the tracking strand through DNA-binding loops lining the central channel, while directing the non-tracking strand out of the rear, in a cyclic process. ATP hydrolysis functions as an 'entropy switch', removing blocks to translocation rather than directly powering DNA movement. Our structures show the allosteric couplings between nucleotide turnover and subunit motions that enable DNA unwinding while maintaining dedicated exit paths for the separated strands. These findings provide a comprehensive model for replication fork establishment and progression that extends from viral to eukaryotic systems. More broadly, they introduce fundamental principles of the mechanism by which ATP-dependent enzymes achieve efficient mechanical work through entropy-driven allostery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50261.map.gz emd_50261.map.gz | 31.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50261-v30.xml emd-50261-v30.xml emd-50261.xml emd-50261.xml | 25.8 KB 25.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_50261.png emd_50261.png | 141 KB | ||
| Filedesc metadata |  emd-50261.cif.gz emd-50261.cif.gz | 7.3 KB | ||
| その他 |  emd_50261_additional_1.map.gz emd_50261_additional_1.map.gz emd_50261_half_map_1.map.gz emd_50261_half_map_1.map.gz emd_50261_half_map_2.map.gz emd_50261_half_map_2.map.gz | 7.4 MB 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50261 http://ftp.pdbj.org/pub/emdb/structures/EMD-50261 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50261 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50261 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50261_validation.pdf.gz emd_50261_validation.pdf.gz | 896.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50261_full_validation.pdf.gz emd_50261_full_validation.pdf.gz | 895.9 KB | 表示 | |
| XML形式データ |  emd_50261_validation.xml.gz emd_50261_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_50261_validation.cif.gz emd_50261_validation.cif.gz | 13.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50261 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50261 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50261 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50261 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9f9wMC  9evhC  9evpC  9exdC  9f3tC  9f3uC  9f5iC  9f73C  9f74C  9f75C  9f7nC  9f9nC  9f9oC  9f9xC  9fa1C  9fa2C  9fb0C  9fb4C  9fb5C  9fb6C  9kaeC  9kakC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50261.map.gz / 形式: CCP4 / 大きさ: 36.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50261.map.gz / 形式: CCP4 / 大きさ: 36.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Volume series map from continuous heterogeneity analysis postprocessed by EMReady. Frame_019 (of frames _000 thru _019) along principal component_001 (of components _000, 001, & _002). | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unpostprocessed volume series map from continuous heterogeneity analysis...
| ファイル | emd_50261_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unpostprocessed volume series map from continuous heterogeneity analysis (5 A lowpass filter). Frame_019 (of frames _000 thru _019) along principal component_001 (of components _000, 001, & _002). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
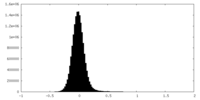
| 密度ヒストグラム |
-ハーフマップ: Unfiltered and unmasked gold-standard half-map (consensus refinement)....
| ファイル | emd_50261_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered and unmasked gold-standard half-map (consensus refinement). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered and unmasked gold-standard half-map (consensus refinement)....
| ファイル | emd_50261_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered and unmasked gold-standard half-map (consensus refinement). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SV40 large T antigen complex with DNA in presence of ATP and Mg2+.
| 全体 | 名称: SV40 large T antigen complex with DNA in presence of ATP and Mg2+. |
|---|---|
| 要素 |
|
-超分子 #1: SV40 large T antigen complex with DNA in presence of ATP and Mg2+.
| 超分子 | 名称: SV40 large T antigen complex with DNA in presence of ATP and Mg2+. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-超分子 #2: Large T antigen
| 超分子 | 名称: Large T antigen / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Betapolyomavirus macacae (ウイルス) Betapolyomavirus macacae (ウイルス) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Large T antigen
| 分子 | 名称: Large T antigen / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Betapolyomavirus macacae (ウイルス) Betapolyomavirus macacae (ウイルス) |
| 分子量 | 理論値: 41.812664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KQVSWKLVTE YAMETKCDDV LLLLGMYLEF QYSFEMCLKC IKKEQPSHYK YHEKHYANAA IFADSKNQKT ICQQAVDTVL AKKRVDSLQ LTREQMLTNR FNDLLDRMDI MFGSTGSADI EEWMAGVAWL HCLLPKMDSV VYDFLKCMVY NIPKKRYWLF K GPIDSGKT ...文字列: KQVSWKLVTE YAMETKCDDV LLLLGMYLEF QYSFEMCLKC IKKEQPSHYK YHEKHYANAA IFADSKNQKT ICQQAVDTVL AKKRVDSLQ LTREQMLTNR FNDLLDRMDI MFGSTGSADI EEWMAGVAWL HCLLPKMDSV VYDFLKCMVY NIPKKRYWLF K GPIDSGKT TLAAALLELC GGKALNVNLP LDRLNFELGV AIDQFLVVFE DVKGTGGESR DLPSGQGINN LDNLRDYLDG SV KVNLEKK HLNKRTQIFP PGIVTMNEYS VPKTLQARFV KQIDFRPKDY LKHCLERSEF LLEKRIIQSG IALLLMLIWY RPV AEFAQS IQSRIVEWKE RLDKEFSLSV YQKMKFNVAM GIGVLD UniProtKB: Large T antigen |
-分子 #2: Chains: S
| 分子 | 名称: Chains: S / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 2.388585 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 5 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM HEPES (pH 7.5), 1 mM ATP, 3 mM MgCl2, 1 mM DTT and 50 mM NaCl. |
| グリッド | モデル: UltrAuFoil / 材質: GOLD / 支持フィルム - 材質: GRAPHENE OXIDE / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 300 sec. / 詳細: 40 mA. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 8460 / 平均露光時間: 2.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER | ||||||
| 得られたモデル |  PDB-9f9w: |
 ムービー
ムービー コントローラー
コントローラー











































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)