+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of neutralizing antibodies CBH-7 and HC84.26 in complex with Hepatitis C virus envelope glycoprotein E2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Immune system / vaccine target / E2 glycoprotein / HCV / ternary complex / ANTIVIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell lipid droplet / clathrin-dependent endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / virion membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Hepacivirus C (ウイルス) Hepacivirus C (ウイルス) | |||||||||
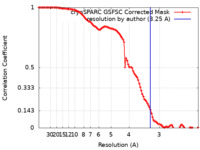
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.25 Å | |||||||||
 データ登録者 データ登録者 | Shahid S / Jiang L / Liu Y / Hasan SS / Mariuzza RA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2025 ジャーナル: Commun Biol / 年: 2025タイトル: Cryo-EM structures of HCV E2 glycoprotein bound to neutralizing and non-neutralizing antibodies determined using bivalent Fabs as fiducial markers. 著者: Salman Shahid / Sharanbasappa S Karade / S Saif Hasan / Rui Yin / Liqun Jiang / Yanxin Liu / Nathaniel Felbinger / Liudmila Kulakova / Eric A Toth / Zhen-Yong Keck / Steven K H Foung / Thomas ...著者: Salman Shahid / Sharanbasappa S Karade / S Saif Hasan / Rui Yin / Liqun Jiang / Yanxin Liu / Nathaniel Felbinger / Liudmila Kulakova / Eric A Toth / Zhen-Yong Keck / Steven K H Foung / Thomas R Fuerst / Brian G Pierce / Roy A Mariuzza /  要旨: Global elimination of hepatitis C virus (HCV) will require an effective cross-genotype vaccine. The HCV E2 envelope glycoprotein is the main target of neutralizing antibodies but also contains ...Global elimination of hepatitis C virus (HCV) will require an effective cross-genotype vaccine. The HCV E2 envelope glycoprotein is the main target of neutralizing antibodies but also contains epitopes that elicit non-neutralizing antibodies which may provide protection through Fc effector functions rather than direct neutralization. We determined cryo-EM structures of a broadly neutralizing antibody, a moderately neutralizing antibody, and a non-neutralizing antibody bound to E2 to resolutions of 3.8, 3.3, and 3.7 Å, respectively. Whereas the broadly neutralizing antibody targeted the front layer of E2 and the non-neutralizing antibody targeted the back layer, the moderately neutralizing antibody straddled both front and back layers, and thereby defined a new neutralizing epitope on E2. The small size of complexes between conventional (monovalent) Fabs and E2 (~110 kDa) presented a challenge for cryo-EM. Accordingly, we engineered bivalent versions of E2-specific Fabs that doubled the size of Fab-E2 complexes and conferred highly identifiable shapes to the complexes that facilitated particle selection and orientation for image processing. This study validates bivalent Fabs as new fiducial markers for cryo-EM analysis of small proteins such as HCV E2 and identifies a new target epitope for vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41275.map.gz emd_41275.map.gz | 64.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41275-v30.xml emd-41275-v30.xml emd-41275.xml emd-41275.xml | 23.8 KB 23.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41275_fsc.xml emd_41275_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41275.png emd_41275.png | 161.9 KB | ||
| Filedesc metadata |  emd-41275.cif.gz emd-41275.cif.gz | 7.4 KB | ||
| その他 |  emd_41275_half_map_1.map.gz emd_41275_half_map_1.map.gz emd_41275_half_map_2.map.gz emd_41275_half_map_2.map.gz | 115.9 MB 115.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41275 http://ftp.pdbj.org/pub/emdb/structures/EMD-41275 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41275 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41275 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41275_validation.pdf.gz emd_41275_validation.pdf.gz | 910.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41275_full_validation.pdf.gz emd_41275_full_validation.pdf.gz | 909.7 KB | 表示 | |
| XML形式データ |  emd_41275_validation.xml.gz emd_41275_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_41275_validation.cif.gz emd_41275_validation.cif.gz | 24.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41275 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41275 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41275 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41275 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8thzMC  8tfeC  8tgvC  8tgzC  8txqC  8tzyC  8u9yC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41275.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41275.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_41275_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
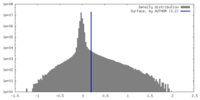
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41275_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
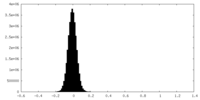
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of CBH7-E2-HC84.26
| 全体 | 名称: Ternary complex of CBH7-E2-HC84.26 |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of CBH7-E2-HC84.26
| 超分子 | 名称: Ternary complex of CBH7-E2-HC84.26 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 340 KDa |
-分子 #1: CBH-7 Heavy chain
| 分子 | 名称: CBH-7 Heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.417719 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDWTWRFLFV VAAATVQSQV QLVQSGAEVR KPGSSVKISC KASGGTFNNY ALSWVRQAPG QGLDWMGEIT PIFGTEKYAQ KFQGRVTIT ADESTNTLYM DLSSLRSEDS AVYYCARRGY IYGSPFDYWG QGTLVTVSSA STKGPSVFPL APSSKSTSGG T AALGCLVK ...文字列: MDWTWRFLFV VAAATVQSQV QLVQSGAEVR KPGSSVKISC KASGGTFNNY ALSWVRQAPG QGLDWMGEIT PIFGTEKYAQ KFQGRVTIT ADESTNTLYM DLSSLRSEDS AVYYCARRGY IYGSPFDYWG QGTLVTVSSA STKGPSVFPL APSSKSTSGG T AALGCLVK DYFPEPVTVS WNSGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKRVEP KS CDKTAGW SHPQFEK |
-分子 #2: CBH-7 Light chain
| 分子 | 名称: CBH-7 Light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.477658 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDMRVPAQLL GLLLLWLRAR CDVLMTQSPS SLSASVGDRV TISCRASQSI SSFLNWYQQK PGKAPKLLIS AASSLSSGVP SRFSGSGSG TSFTLTISSL QPEDVATYYC QQSYSFLLTF GGGTNVEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN F YPREAKVQ ...文字列: MDMRVPAQLL GLLLLWLRAR CDVLMTQSPS SLSASVGDRV TISCRASQSI SSFLNWYQQK PGKAPKLLIS AASSLSSGVP SRFSGSGSG TSFTLTISSL QPEDVATYYC QQSYSFLLTF GGGTNVEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN F YPREAKVQ WKVDNALQSG NSQESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #3: envelope glycoprotein E2
| 分子 | 名称: envelope glycoprotein E2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hepacivirus C (ウイルス) Hepacivirus C (ウイルス) |
| 分子量 | 理論値: 32.28726 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METDTLLLWV LLLWVPGSTG DSTHVTGGTA SHTTRHFASL FSSGASQRVQ LINTNGSWHI NRTALNCNDS LHTGFLAALF YTHKFNASG CPERMAHCRP IDEFAQGWGP ITYAEGHGSD QRPYCWHYAP RQCGTIPASQ VCGPVYCFTP SPVVVGTTDR F GAPTYTWG ...文字列: METDTLLLWV LLLWVPGSTG DSTHVTGGTA SHTTRHFASL FSSGASQRVQ LINTNGSWHI NRTALNCNDS LHTGFLAALF YTHKFNASG CPERMAHCRP IDEFAQGWGP ITYAEGHGSD QRPYCWHYAP RQCGTIPASQ VCGPVYCFTP SPVVVGTTDR F GAPTYTWG ENETDVLILN NTRPPQGNWF GCTWMNSTGF TKTCGGPPCN IGGVGNNTLT CPTDCFRKHP EATYTKCGSG PW LTPRCLV DYPYRLWHYP CTVNFTIFKV RMYVGGVEHR LNAACNIGHH HHHH UniProtKB: Genome polyprotein |
-分子 #4: HC84.26 Heavy chain
| 分子 | 名称: HC84.26 Heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.356758 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METDTLLLWV LLLWVPGSTG DQVQPVQSGA EVKKPGSSVK VSCEASGGTH SNYVITWVRQ APGQGLEWMG GFIPDFRTAM YAQGFQGRV TITADESTSL AYMELTNLRS EDTAVYYCAR GPLSRGYYDY WGPGTLVTVS SASTKGPSVF PLAPSSKSTS G GTAALGCL ...文字列: METDTLLLWV LLLWVPGSTG DQVQPVQSGA EVKKPGSSVK VSCEASGGTH SNYVITWVRQ APGQGLEWMG GFIPDFRTAM YAQGFQGRV TITADESTSL AYMELTNLRS EDTAVYYCAR GPLSRGYYDY WGPGTLVTVS SASTKGPSVF PLAPSSKSTS G GTAALGCL VKDYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKRV EP KSCDKTA GWSHPQFEKT AATGACTCGA G |
-分子 #5: HC84.26 Light chain
| 分子 | 名称: HC84.26 Light chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.35215 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METDTLLLWV LLLWVPGSTG DSYVLTQPPS VSVAPGQTAS ITCSGDKLGD KYVSWYQQRP GQSPVLVLYQ DSKRPSGIPE RFSGSNSGN TATLTISGTQ AMDEADYYCQ AWDSSALVFG GGTKLTVLRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF Y PREAKVQW ...文字列: METDTLLLWV LLLWVPGSTG DSYVLTQPPS VSVAPGQTAS ITCSGDKLGD KYVSWYQQRP GQSPVLVLYQ DSKRPSGIPE RFSGSNSGN TATLTISGTQ AMDEADYYCQ AWDSSALVFG GGTKLTVLRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF Y PREAKVQW KVDNALQSGN SQESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20 mM Tris-HCl pH 8.0 100 mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 7058 / 平均露光時間: 3.6 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Cross-Correlation | ||||||
| 得られたモデル |  PDB-8thz: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)