+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-32328 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of GmALMT12/QUAC1 anion channel | ||||||||||||
 マップデータ マップデータ | Half map 2 for GmALMT12/QUAC1 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Symmetrical dimer / T-shaped pore / twisted two-layer architecture / Malate-modulation / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 | malate transport / Aluminum-activated malate transporter / Aluminium activated malate transporter / plant-type vacuole membrane / monoatomic ion transmembrane transport / membrane => GO:0016020 / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Qin L / Tang LH | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Cryo-EM structure and electrophysiological characterization of ALMT from reveal a previously uncharacterized class of anion channels. 著者: Li Qin / Ling-Hui Tang / Jia-Shu Xu / Xian-Hui Zhang / Yun Zhu / Chun-Rui Zhang / Mei-Hua Wang / Xue-Lei Liu / Fei Li / Fei Sun / Min Su / Yujia Zhai / Yu-Hang Chen /   要旨: Aluminum-activated malate transporters (ALMTs) form an anion channel family that plays essential roles in diverse functions in plants. ALMT12, also named QUAC1 (quick anion channel 1), regulates ...Aluminum-activated malate transporters (ALMTs) form an anion channel family that plays essential roles in diverse functions in plants. ALMT12, also named QUAC1 (quick anion channel 1), regulates stomatal closure in response to environmental stimuli. However, the molecular basis of ALMT12/QUAC1 activity remains elusive. Here, we describe the cryo-EM structure of ALMT12/QUAC1 from at 3.5-Å resolution. ALMT12/QUAC1 is a symmetrical dimer, forming a single electropositive T-shaped pore across the membrane. The transmembrane and cytoplasmic domains are assembled into a twisted two-layer architecture, with their associated dimeric interfaces nearly perpendicular. ALMT12/QUAC1-mediated currents display rapid kinetics of activation/deactivation and a bell-shaped voltage dependency, reminiscent of the rapid (R)-type anion currents. Our structural and functional analyses reveal a domain-twisting mechanism for malate-mediated activation. Together, our study uncovers the molecular basis for a previously uncharacterized class of anion channels and provides insights into the gating and modulation of the ALMT12/QUAC1 anion channel. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32328.map.gz emd_32328.map.gz | 21 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32328-v30.xml emd-32328-v30.xml emd-32328.xml emd-32328.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32328.png emd_32328.png | 69.9 KB | ||
| Filedesc metadata |  emd-32328.cif.gz emd-32328.cif.gz | 6 KB | ||
| その他 |  emd_32328_additional_1.map.gz emd_32328_additional_1.map.gz emd_32328_half_map_1.map.gz emd_32328_half_map_1.map.gz emd_32328_half_map_2.map.gz emd_32328_half_map_2.map.gz | 40.3 MB 39.8 MB 39.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32328 http://ftp.pdbj.org/pub/emdb/structures/EMD-32328 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32328 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32328 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32328_validation.pdf.gz emd_32328_validation.pdf.gz | 732 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32328_full_validation.pdf.gz emd_32328_full_validation.pdf.gz | 731.5 KB | 表示 | |
| XML形式データ |  emd_32328_validation.xml.gz emd_32328_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_32328_validation.cif.gz emd_32328_validation.cif.gz | 13.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32328 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32328 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32328 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32328 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32328.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32328.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 for GmALMT12/QUAC1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Sharpen map for GmALMT12/QUAC1
| ファイル | emd_32328_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpen map for GmALMT12/QUAC1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
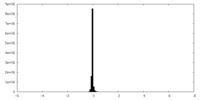
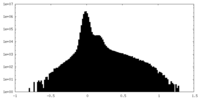
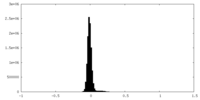
| 密度ヒストグラム |
-ハーフマップ: Reconstructed Density map for GmALMT12/QUAC1
| ファイル | emd_32328_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstructed Density map for GmALMT12/QUAC1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
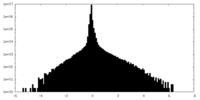
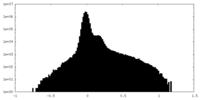
| 密度ヒストグラム |
-ハーフマップ: Half map 1 for GmALMT12/QUAC1
| ファイル | emd_32328_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 for GmALMT12/QUAC1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
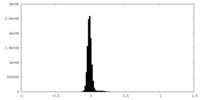
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GmALMT12/QUAC1
| 全体 | 名称: GmALMT12/QUAC1 |
|---|---|
| 要素 |
|
-超分子 #1: GmALMT12/QUAC1
| 超分子 | 名称: GmALMT12/QUAC1 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: GmALMT12/QUAC1
| 分子 | 名称: GmALMT12/QUAC1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 60.321969 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVPKVYAGQE MAMVENENCI MNGKWKKRVH VFGERVMRFP NKAWQTTWKV GREDPRRLIH AFKVGLSLTL ASLLYLLEPL FKGIGQSAI WAVMTVVVVL EFTAGATLCK GLNRGLGTLL AGLLAFLVGY IANASDRVSQ AIIIGAAVFF IGALATYMRF I PYIKKNYD ...文字列: MVPKVYAGQE MAMVENENCI MNGKWKKRVH VFGERVMRFP NKAWQTTWKV GREDPRRLIH AFKVGLSLTL ASLLYLLEPL FKGIGQSAI WAVMTVVVVL EFTAGATLCK GLNRGLGTLL AGLLAFLVGY IANASDRVSQ AIIIGAAVFF IGALATYMRF I PYIKKNYD YGLVIFLLTF NLITVSSYRL ENVLKIAHDR VYTIAIGCAV CLLMSLLVFP NWSGEDLHNS TVYKLEGLAK SI EACVNEY FYGEIEGSGY MKLSEDPIYK GYKAVLDSKS IDETLALHAS WEPRHSRYCH RFPWQQYVKV GAVLRQFGYT VVA LHGCLR TEIQTPRSVR AMFKDPCIRL AAEVSKVLIE LSNSIRNRRH CSPEILSDHL HEALQDLNTA IKSQPRLFLG PKHR HNQAT NMLKIAAAQV GQERHGKTSL SSVKTDSSAL LEWKTKRVSA EQTKESERKS LRPQLSKIAI TSLEFSEALP FAAFA SLLV ETVAKLDLVI EEVEELGRLA CFKEFIPGDE FVVTCQEPRV DVSQNHLPSH GVD UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 6189 / 平均露光時間: 8.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7w6k: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X