[English] 日本語
 Yorodumi
Yorodumi- EMDB-31432: Cryo-EM structure of the minimal protein-only RNase P from Aquife... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-31432 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
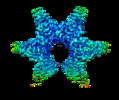
| Title | Cryo-EM structure of the minimal protein-only RNase P from Aquifex aeolicus reveals structural insight into precursor tRNA recognition and catalysis | ||||||||||||
 Map data Map data | |||||||||||||
 Sample Sample |
| ||||||||||||
| Function / homology | RNA-free ribonuclease P / PINc domain ribonuclease / ribonuclease P / ribonuclease P activity / tRNA 5'-leader removal / PIN-like domain superfamily / RNA-free ribonuclease P Function and homology information Function and homology information | ||||||||||||
| Biological species |   Aquifex aeolicus (bacteria) Aquifex aeolicus (bacteria) | ||||||||||||
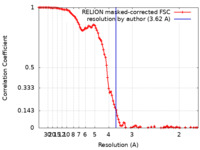
| Method | single particle reconstruction / cryo EM / Resolution: 3.62 Å | ||||||||||||
 Authors Authors | Teramoto T / Koyasu T / Adachi N / Kawasaki M / Moriya T / Numata T / Senda T / Kakuta Y | ||||||||||||
| Funding support |  Japan, 3 items Japan, 3 items
| ||||||||||||
 Citation Citation |  Journal: J Biol Chem / Year: 2021 Journal: J Biol Chem / Year: 2021Title: Minimal protein-only RNase P structure reveals insights into tRNA precursor recognition and catalysis. Authors: Takamasa Teramoto / Takeshi Koyasu / Naruhiko Adachi / Masato Kawasaki / Toshio Moriya / Tomoyuki Numata / Toshiya Senda / Yoshimitsu Kakuta /  Abstract: Ribonuclease P (RNase P) is an endoribonuclease that catalyzes the processing of the 5' leader sequence of precursor tRNA (pre-tRNA). Ribonucleoprotein RNase P and protein-only RNase P (PRORP) in ...Ribonuclease P (RNase P) is an endoribonuclease that catalyzes the processing of the 5' leader sequence of precursor tRNA (pre-tRNA). Ribonucleoprotein RNase P and protein-only RNase P (PRORP) in eukaryotes have been extensively studied, but the mechanism by which a prokaryotic nuclease recognizes and cleaves pre-tRNA is unclear. To gain insights into this mechanism, we studied homologs of Aquifex RNase P (HARPs), thought to be enzymes of approximately 23 kDa comprising only this nuclease domain. We determined the cryo-EM structure of Aq880, the first identified HARP enzyme. The structure unexpectedly revealed that Aq880 consists of both the nuclease and protruding helical (PrH) domains. Aq880 monomers assemble into a dimer via the PrH domain. Six dimers form a dodecamer with a left-handed one-turn superhelical structure. The structure also revealed that the active site of Aq880 is analogous to that of eukaryotic PRORPs. The pre-tRNA docking model demonstrated that 5' processing of pre-tRNAs is achieved by two adjacent dimers within the dodecamer. One dimer is responsible for catalysis, and the PrH domains of the other dimer are responsible for pre-tRNA elbow recognition. Our study suggests that HARPs measure an invariant distance from the pre-tRNA elbow to cleave the 5' leader sequence, which is analogous to the mechanism of eukaryotic PRORPs and the ribonucleoprotein RNase P. Collectively, these findings shed light on how different types of RNase P enzymes utilize the same pre-tRNA processing. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_31432.map.gz emd_31432.map.gz | 93.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-31432-v30.xml emd-31432-v30.xml emd-31432.xml emd-31432.xml | 20.8 KB 20.8 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_31432_fsc.xml emd_31432_fsc.xml | 10.7 KB | Display |  FSC data file FSC data file |
| Images |  emd_31432.png emd_31432.png | 110.1 KB | ||
| Masks |  emd_31432_msk_1.map emd_31432_msk_1.map | 103 MB |  Mask map Mask map | |
| Others |  emd_31432_additional_1.map.gz emd_31432_additional_1.map.gz emd_31432_half_map_1.map.gz emd_31432_half_map_1.map.gz emd_31432_half_map_2.map.gz emd_31432_half_map_2.map.gz | 78.2 MB 79.4 MB 79.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31432 http://ftp.pdbj.org/pub/emdb/structures/EMD-31432 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31432 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31432 | HTTPS FTP |
-Related structure data
| Related structure data |  7f3eMC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | |
| EM raw data |  EMPIAR-11072 (Title: Minimal protein-only RNase P structure reveals insights into tRNA precursor recognition and catalysis EMPIAR-11072 (Title: Minimal protein-only RNase P structure reveals insights into tRNA precursor recognition and catalysisData size: 2.0 TB Data #1: Minimal protein-only RNase P structure reveals insights into tRNA precursor recognition and catalysis [micrographs - multiframe]) |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_31432.map.gz / Format: CCP4 / Size: 103 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_31432.map.gz / Format: CCP4 / Size: 103 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Mask #1
| File |  emd_31432_msk_1.map emd_31432_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Additional map: #1
| File | emd_31432_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_31432_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_31432_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Aq880 dodecamer
| Entire | Name: Aq880 dodecamer |
|---|---|
| Components |
|
-Supramolecule #1: Aq880 dodecamer
| Supramolecule | Name: Aq880 dodecamer / type: complex / ID: 1 / Parent: 0 / Macromolecule list: all / Details: homo dodecamer |
|---|---|
| Source (natural) | Organism:   Aquifex aeolicus (bacteria) Aquifex aeolicus (bacteria) |
| Recombinant expression | Organism:  |
| Molecular weight | Theoretical: 270 KDa |
-Macromolecule #1: Aq880
| Macromolecule | Name: Aq880 / type: protein_or_peptide / ID: 1 / Details: GGATS is derived from plasmid / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:   Aquifex aeolicus (bacteria) Aquifex aeolicus (bacteria) |
| Recombinant expression | Organism:  |
| Sequence | String: GGATSMDVFV LDTSVFTNPE IYRTFEEDQR GAMETFIHLA LNSRAEFYMP TSVYTEMRKI MDVGELWAEF EMVVKIRSPR RFQLTVPADF LYEFIEELRY RINKGLRIAE EHTREASGCE DVGKLIARLR EKYREALRQG ILDSKEDVDV LLLAYELDGV LVSADEGLRT ...String: GGATSMDVFV LDTSVFTNPE IYRTFEEDQR GAMETFIHLA LNSRAEFYMP TSVYTEMRKI MDVGELWAEF EMVVKIRSPR RFQLTVPADF LYEFIEELRY RINKGLRIAE EHTREASGCE DVGKLIARLR EKYREALRQG ILDSKEDVDV LLLAYELDGV LVSADEGLRT WADKIGIKLI DPKNFKNILE SLVRHRF |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 5.0 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 8 Component:
| ||||||||
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Mesh: 300 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Atmosphere: AIR / Details: The grid was washed by acetone prior to use. | ||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 291 K / Instrument: FEI VITROBOT MARK IV / Details: Blotting time was 5 seconds (blot force 20). | ||||||||
| Details | This sample was mono-disperse. |
- Electron microscopy
Electron microscopy
| Microscope | TFS TALOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON III (4k x 4k) / Detector mode: COUNTING / Number grids imaged: 1 / Number real images: 2370 / Average exposure time: 48.62 sec. / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 200 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.0 µm / Nominal magnification: 120000 |
| Sample stage | Cooling holder cryogen: NITROGEN |
 Movie
Movie Controller
Controller












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)