+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31013 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of human Kv4.2-DPP6S complex | |||||||||
 マップデータ マップデータ | map E | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Kv4.2-KChIP2 channel complex / A-type (transient outward) potassium channel activity / Phase 1 - inactivation of fast Na+ channels / voltage-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / membrane repolarization / Voltage gated Potassium channels / regulation of potassium ion transmembrane transport / postsynaptic specialization membrane / anchoring junction / action potential ...Kv4.2-KChIP2 channel complex / A-type (transient outward) potassium channel activity / Phase 1 - inactivation of fast Na+ channels / voltage-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / membrane repolarization / Voltage gated Potassium channels / regulation of potassium ion transmembrane transport / postsynaptic specialization membrane / anchoring junction / action potential / regulation of heart contraction / neuronal cell body membrane / voltage-gated potassium channel activity / plasma membrane raft / locomotor rhythm / GABA-ergic synapse / potassium channel regulator activity / neuronal action potential / potassium ion transmembrane transport / voltage-gated potassium channel complex / sensory perception of pain / serine-type peptidase activity / muscle contraction / protein localization to plasma membrane / protein homooligomerization / cellular response to hypoxia / chemical synaptic transmission / postsynaptic membrane / perikaryon / dendritic spine / neuronal cell body / glutamatergic synapse / proteolysis / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Kise Y / Nureki O | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structural basis of gating modulation of Kv4 channel complexes. 著者: Yoshiaki Kise / Go Kasuya / Hiroyuki H Okamoto / Daichi Yamanouchi / Kan Kobayashi / Tsukasa Kusakizako / Tomohiro Nishizawa / Koichi Nakajo / Osamu Nureki /  要旨: Modulation of voltage-gated potassium (Kv) channels by auxiliary subunits is central to the physiological function of channels in the brain and heart. Native Kv4 tetrameric channels form ...Modulation of voltage-gated potassium (Kv) channels by auxiliary subunits is central to the physiological function of channels in the brain and heart. Native Kv4 tetrameric channels form macromolecular ternary complexes with two auxiliary β-subunits-intracellular Kv channel-interacting proteins (KChIPs) and transmembrane dipeptidyl peptidase-related proteins (DPPs)-to evoke rapidly activating and inactivating A-type currents, which prevent the backpropagation of action potentials. However, the modulatory mechanisms of Kv4 channel complexes remain largely unknown. Here we report cryo-electron microscopy structures of the Kv4.2-DPP6S-KChIP1 dodecamer complex, the Kv4.2-KChIP1 and Kv4.2-DPP6S octamer complexes, and Kv4.2 alone. The structure of the Kv4.2-KChIP1 complex reveals that the intracellular N terminus of Kv4.2 interacts with its C terminus that extends from the S6 gating helix of the neighbouring Kv4.2 subunit. KChIP1 captures both the N and the C terminus of Kv4.2. In consequence, KChIP1 would prevent N-type inactivation and stabilize the S6 conformation to modulate gating of the S6 helices within the tetramer. By contrast, unlike the reported auxiliary subunits of voltage-gated channel complexes, DPP6S interacts with the S1 and S2 helices of the Kv4.2 voltage-sensing domain, which suggests that DPP6S stabilizes the conformation of the S1-S2 helices. DPP6S may therefore accelerate the voltage-dependent movement of the S4 helices. KChIP1 and DPP6S do not directly interact with each other in the Kv4.2-KChIP1-DPP6S ternary complex. Thus, our data suggest that two distinct modes of modulation contribute in an additive manner to evoke A-type currents from the native Kv4 macromolecular complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31013.map.gz emd_31013.map.gz | 20 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31013-v30.xml emd-31013-v30.xml emd-31013.xml emd-31013.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31013_fsc.xml emd_31013_fsc.xml | 11.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31013.png emd_31013.png | 74 KB | ||
| マスクデータ |  emd_31013_msk_1.map emd_31013_msk_1.map | 139.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_31013_half_map_1.map.gz emd_31013_half_map_1.map.gz emd_31013_half_map_2.map.gz emd_31013_half_map_2.map.gz | 108.1 MB 108.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31013 http://ftp.pdbj.org/pub/emdb/structures/EMD-31013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31013 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31013_validation.pdf.gz emd_31013_validation.pdf.gz | 648.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31013_full_validation.pdf.gz emd_31013_full_validation.pdf.gz | 648.2 KB | 表示 | |
| XML形式データ |  emd_31013_validation.xml.gz emd_31013_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_31013_validation.cif.gz emd_31013_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31013 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31013 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31013 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31013 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7e8bMC  7e7zC  7e83C  7e84C  7e87C  7e89C  7e8eC  7e8gC  7e8hC  7f0jC  7f3fC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31013.map.gz / 形式: CCP4 / 大きさ: 139.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31013.map.gz / 形式: CCP4 / 大きさ: 139.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map E | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_31013_msk_1.map emd_31013_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_31013_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_31013_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human Kv4.2-DPP6S complex
| 全体 | 名称: human Kv4.2-DPP6S complex |
|---|---|
| 要素 |
|
-超分子 #1: human Kv4.2-DPP6S complex
| 超分子 | 名称: human Kv4.2-DPP6S complex / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Dipeptidyl aminopeptidase-like protein 6
| 分子 | 名称: Dipeptidyl aminopeptidase-like protein 6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 86.392797 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: WKGIAIALLV ILVICSLIVT SVILLTPAED NSLSQKKKVT VEDLFSEDFK IHDPEAKWIS DTEFIYREQK GTVRLWNVET NTSTVLIEG KKIESLRAIR YEISPDREYA LFSYNVEPIY QHSYTGYYVL SKIPHGDPQS LDPPEVSNAK LQYAGWGPKG Q QLIFIFEN ...文字列: WKGIAIALLV ILVICSLIVT SVILLTPAED NSLSQKKKVT VEDLFSEDFK IHDPEAKWIS DTEFIYREQK GTVRLWNVET NTSTVLIEG KKIESLRAIR YEISPDREYA LFSYNVEPIY QHSYTGYYVL SKIPHGDPQS LDPPEVSNAK LQYAGWGPKG Q QLIFIFEN NIYYCAHVGK QAIRVVSTGK EGVIYNGLSD WLYEEEILKT HIAHWWSPDG TRLAYAAIND SRVPIMELPT YT GSIYPTV KPYHYPKAGS ENPSISLHVI GLNGPTHDLE MMPPDDPRMR EYYITMVKWA TSTKVAVTWL NRAQNVSILT LCD ATTGVC TKKHEDESEA WLHRQNEEPV FSKDGRKFFF IRAIPQGGRG KFYHITVSSS QPNSSNDNIQ SITSGDWDVT KILA YDEKG NKIYFLSTED LPRRRQLYSA NTVGNFNRQC LSCDLVENCT YFSASFSHSM DFFLLKCEGP GVPMVTVHNT TDKKK MFDL ETNEHVKKAI NDRQMPKVEY RDIEIDDYNL PMQILKPATF TDTTHYPLLL VVDGTPGSQS VAEKFEVSWE TVMVSS HGA VVVKCDGRGS GFQGTKLLHE VRRRLGLLEE KDQMEAVRTM LKEQYIDRTR VAVFGKDYGG YLSTYILPAK GENQGQT FT CGSALSPITD FKLYASAFSE RYLGLHGLDN RAYEMTKVAH RVSALEEQQF LIIHPTADEK IHFQHTAELI TQLIRGKA N YSLQIYPDES HYFTSSSLKQ HLYRSIINFF VECFRI |
-分子 #2: Potassium voltage-gated channel subfamily D member 2
| 分子 | 名称: Potassium voltage-gated channel subfamily D member 2 タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.297133 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DALIVLNVSG TRFQTWQDTL ERYPDTLLGS SERDFFYHPE TQQYFFDRDP DIFRHILNFY RTGKLHYPRH ECISAYDEEL AFFGLIPEI IGDCCYEEYK DRRRENAERL QDDADTDTAG ESALPTMTAR QRVWRAFENP HTSTMALVFY YVTGFFIAVS V IANVVETV ...文字列: DALIVLNVSG TRFQTWQDTL ERYPDTLLGS SERDFFYHPE TQQYFFDRDP DIFRHILNFY RTGKLHYPRH ECISAYDEEL AFFGLIPEI IGDCCYEEYK DRRRENAERL QDDADTDTAG ESALPTMTAR QRVWRAFENP HTSTMALVFY YVTGFFIAVS V IANVVETV PCGSSPGHIK ELPCGERYAV AFFCLDTACV MIFTVEYLLR LAAAPSRYRF VRSVMSIIDV VAILPYYIGL VM TDNEDVS GAFVTLRVFR VFRIFKFSRH SQGLRILGYT LKSCASELGF LLFSLTMAII IFATVMFYAE KGSSASKFTS IPA AFWYTI VTMTTLGYGD MVPKTIAGKI FGSICSLSGV LVIALPVPVI VSNFSRIYHQ NQRADKRRAQ KKARLARIRA AK |
-分子 #3: Potassium voltage-gated channel subfamily D member 2
| 分子 | 名称: Potassium voltage-gated channel subfamily D member 2 タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.167953 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DALIVLNVSG TRFQTWQDTL ERYPDTLLGS SERDFFYHPE TQQYFFDRDP DIFRHILNFY RTGKLHYPRH ECISAYDEEL AFFGLIPEI IGDCCYEEYK DRRRENAERL QDDADTDTAG ESALPTMTAR QRVWRAFENP HTSTMALVFY YVTGFFIAVS V IANVVETV ...文字列: DALIVLNVSG TRFQTWQDTL ERYPDTLLGS SERDFFYHPE TQQYFFDRDP DIFRHILNFY RTGKLHYPRH ECISAYDEEL AFFGLIPEI IGDCCYEEYK DRRRENAERL QDDADTDTAG ESALPTMTAR QRVWRAFENP HTSTMALVFY YVTGFFIAVS V IANVVETV PCGSSPGHIK ELPCGERYAV AFFCLDTACV MIFTVEYLLR LAAAPSRYRF VRSVMSIIDV VAILPYYIGL VM TDNEDVS GAFVTLRVFR VFRIFKFSRH SQGLRILGYT LKSCASELGF LLFSLTMAII IFATVMFYAE KGSSASKFTS IPA AFWYTI VTMTTLGYGD MVPKTIAGKI FGSICSLSGV LVIALPVPVI VSNFSRIYHQ NQRADKRRAQ KKARLARIRA A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 91974 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| FSC曲線 (解像度の算出) | 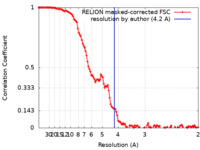 |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-7e8b: |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X