+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22422 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of an endocytic receptor | |||||||||
 マップデータ マップデータ | Final map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endocytosis / signaling receptor activity / carbohydrate binding / inflammatory response / immune response / external side of plasma membrane / extracellular exosome / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
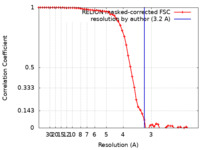
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Gully BS / Rossjohn J / Berry R | |||||||||
| 資金援助 |  オーストラリア, 2件 オーストラリア, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2021 ジャーナル: J Biol Chem / 年: 2021タイトル: The cryo-EM structure of the endocytic receptor DEC-205. 著者: Benjamin S Gully / Hariprasad Venugopal / Alex J Fulcher / Zhihui Fu / Jessica Li / Felix A Deuss / Carmen Llerena / William R Heath / Mireille H Lahoud / Irina Caminschi / Jamie Rossjohn / Richard Berry /   要旨: DEC-205 (CD205), a member of the macrophage mannose receptor protein family, is the prototypic endocytic receptor of dendritic cells, whose ligands include phosphorothioated cytosine-guanosine ...DEC-205 (CD205), a member of the macrophage mannose receptor protein family, is the prototypic endocytic receptor of dendritic cells, whose ligands include phosphorothioated cytosine-guanosine oligonucleotides, a motif often seen in bacterial or viral DNA. However, despite growing biological and clinical significance, little is known about the structural arrangement of this receptor or any of its family members. Here, we describe the 3.2 Å cryo-EM structure of human DEC-205, thereby illuminating the structure of the mannose receptor protein family. The DEC-205 monomer forms a compact structure comprising two intercalated rings of C-type lectin-like domains, where the N-terminal cysteine-rich and fibronectin domains reside at the central intersection. We establish a pH-dependent oligomerization pathway forming tetrameric DEC-205 using solution-based techniques and ultimately solved the 4.9 Å cryo-EM structure of the DEC-205 tetramer to identify the unfurling of the second lectin ring which enables tetramer formation. Furthermore, we suggest the relevance of this oligomerization pathway within a cellular setting, whereby cytosine-guanosine binding appeared to disrupt this cell-surface oligomer. Accordingly, we provide insight into the structure and oligomeric assembly of the DEC-205 receptor. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22422.map.gz emd_22422.map.gz | 2.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22422-v30.xml emd-22422-v30.xml emd-22422.xml emd-22422.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22422_fsc.xml emd_22422_fsc.xml | 5.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22422.png emd_22422.png | 151.6 KB | ||
| マスクデータ |  emd_22422_msk_1.map emd_22422_msk_1.map | 15.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22422_half_map_1.map.gz emd_22422_half_map_1.map.gz emd_22422_half_map_2.map.gz emd_22422_half_map_2.map.gz | 11.9 MB 11.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22422 http://ftp.pdbj.org/pub/emdb/structures/EMD-22422 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22422 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22422 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22422_validation.pdf.gz emd_22422_validation.pdf.gz | 582.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22422_full_validation.pdf.gz emd_22422_full_validation.pdf.gz | 582.2 KB | 表示 | |
| XML形式データ |  emd_22422_validation.xml.gz emd_22422_validation.xml.gz | 11.3 KB | 表示 | |
| CIF形式データ |  emd_22422_validation.cif.gz emd_22422_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22422 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22422 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22422 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22422 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22422.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22422.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22422_msk_1.map emd_22422_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
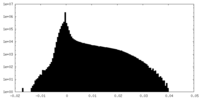
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_22422_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_22422_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lymphocyte antigen 75, DEC205, CD205
| 全体 | 名称: Lymphocyte antigen 75, DEC205, CD205 |
|---|---|
| 要素 |
|
-超分子 #1: Lymphocyte antigen 75, DEC205, CD205
| 超分子 | 名称: Lymphocyte antigen 75, DEC205, CD205 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 実験値: 200 KDa |
-分子 #1: Lymphocyte antigen 75
| 分子 | 名称: Lymphocyte antigen 75 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 195.23025 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RAANDPFTIV HGNTGKCIKP VYGWIVADDC DETEDKLWKW VSQHRLFHLH SQKCLGLDIT KSVNELRMFS CDSSAMLWWK CEHHSLYGA ARYRLALKDG HGTAISNASD VWKKGGSEES LCDQPYHEIY TRDGNSYGRP CEFPFLIDGT WHHDCILDED H SGPWCATT ...文字列: RAANDPFTIV HGNTGKCIKP VYGWIVADDC DETEDKLWKW VSQHRLFHLH SQKCLGLDIT KSVNELRMFS CDSSAMLWWK CEHHSLYGA ARYRLALKDG HGTAISNASD VWKKGGSEES LCDQPYHEIY TRDGNSYGRP CEFPFLIDGT WHHDCILDED H SGPWCATT LNYEYDRKWG ICLKPENGCE DNWEKNEQFG SCYQFNTQTA LSWKEAYVSC QNQGADLLSI NSAAELTYLK EK EGIAKIF WIGLNQLYSA RGWEWSDHKP LNFLNWDPDR PSAPTIGGSS CARMDAESGL WQSFSCEAQL PYVCRKPLNN TVE LTDVWT YSDTRCDAGW LPNNGFCYLL VNESNSWDKA HAKCKAFSSD LISIHSLADV EVVVTKLHNE DIKEEVWIGL KNIN IPTLF QWSDGTEVTL TYWDENEPNV PYNKTPNCVS YLGELGQWKV QSCEEKLKYV CKRKGEKLND ASSDKMCPPD EGWKR HGET CYKIYEDEVP FGTNCNLTIT SRFEQEYLND LMKKYDKSLR KYFWTGLRDV DSCGEYNWAT VGGRRRAVTF SNWNFL EPA SPGGCVAMST GKSVGKWEVK DCRSFKALSI CKKMSGPLGP EEASPKPDDP CPEGWQSFPA SLSCYKVFHA ERIVRKR NW EEAERFCQAL GAHLSSFSHV DEIKEFLHFL TDQFSGQHWL WIGLNKRSPD LQGSWQWSDR TPVSTIIMPN EFQQDYDI R DCAAVKVFHR PWRRGWHFYD DREFIYLRPF ACDTKLEWVC QIPKGRTPKT PDWYNPDRAG IHGPPLIIEG SEYWFVADL HLNYEEAVLY CASNHSFLAT ITSFVGLKAI KNKIANISGD GQKWWIRISE WPIDDHFTYS RYPWHRFPVT FGEECLYMSA KTWLIDLGK PTDCSTKLPF ICEKYNVSSL EKYSPDSAAK VQCSEQWIPF QNKCFLKIKP VSLTFSQASD TCHSYGGTLP S VLSQIEQD FITSLLPDME ATLWIGLRWT AYEKINKWTD NRELTYSNFH PLLVSGRLRI PENFFEEESR YHCALILNLQ KS PFTGTWN FTSCSERHFV SLCQKYSEVK SRQTLQNASE TVKYLNNLYK IIPKTLTWHS AKRECLKSNM QLVSITDPYQ QAF LSVQAL LHNSSLWIGL FSQDDELNFG WSDGKRLHFS RWAETNGQLE DCVVLDTDGF WKTVDCNDNQ PGAICYYSGN ETEK EVKPV DSVKCPSPVL NTPWIPFQNC CYNFIITKNR HMATTQDEVH TKCQKLNPKS HILSIRDEKE NNFVLEQLLY FNYMA SWVM LGITYRNKSL MWFDKTPLSY THWRAGRPTI KNEKFLAGLS TDGFWDIQTF KVIEEAVYFH QHSILACKIE MVDYKE EYN TTLPQFMPYE DGIYSVIQKK VTWYEALNMC SQSGGHLASV HNQNGQLFLE DIVKRDGFPL WVGLSSHDGS ESSFEWS DG STFDYIPWKG QTSPGNCVLL DPKGTWKHEK CNSVKDGAIC YKPTKSKKLS RLTYSSRCPA AKENGSRWIQ YKGHCYKS D QALHSFSEAK KLCSKHDHSA TIVSIKDEDE NKFVSRLMRE NNNITMRVWL GLSQHSVDQS WSWLDGSEVT FVKWENKSK SGVGRCSMLI ASNETWKKVE CEHGFGRVVC KVPLGPDYTA IAIIVATLSI LVLMGGLIWF LFQRHRLHLA GFSSVRYAQG VNEDEIMLP SFHD |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: 20 mM Tris-HCl at pH 8.0, 150 mM NaCl buffer | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK II | ||||||
| 詳細 | 3 microlitres of sample in 20 mM Tris-HCl at pH 8.0, 150 mM NaCl buffer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 63.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X