+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21559 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle cryoEM structure of V. cholerae Type IV competence pilus secretin PilQ | |||||||||
 マップデータ マップデータ | Type IV competence pilus secretin PilQ Relion PostProcess map (postprocess.mrc) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | secretion system / outer membrane protein / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pilus assembly / protein secretion by the type II secretion system / protein secretion / cell outer membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
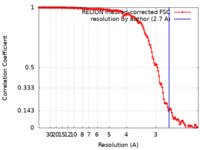
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Weaver SJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: CryoEM structure of the type IVa pilus secretin required for natural competence in Vibrio cholerae. 著者: Sara J Weaver / Davi R Ortega / Matthew H Sazinsky / Triana N Dalia / Ankur B Dalia / Grant J Jensen /  要旨: Natural transformation is the process by which bacteria take up genetic material from their environment and integrate it into their genome by homologous recombination. It represents one mode of ...Natural transformation is the process by which bacteria take up genetic material from their environment and integrate it into their genome by homologous recombination. It represents one mode of horizontal gene transfer and contributes to the spread of traits like antibiotic resistance. In Vibrio cholerae, a type IVa pilus (T4aP) is thought to facilitate natural transformation by extending from the cell surface, binding to exogenous DNA, and retracting to thread this DNA through the outer membrane secretin, PilQ. Here, we use a functional tagged allele of VcPilQ purified from native V. cholerae cells to determine the cryoEM structure of the VcPilQ secretin in amphipol to ~2.7 Å. We use bioinformatics to examine the domain architecture and gene neighborhood of T4aP secretins in Proteobacteria in comparison with VcPilQ. This structure highlights differences in the architecture of the T4aP secretin from the type II and type III secretion system secretins. Based on our cryoEM structure, we design a series of mutants to reversibly regulate VcPilQ gate dynamics. These experiments support the idea of VcPilQ as a potential druggable target and provide insight into the channel that DNA likely traverses to promote the spread of antibiotic resistance via horizontal gene transfer by natural transformation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21559.map.gz emd_21559.map.gz | 228.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21559-v30.xml emd-21559-v30.xml emd-21559.xml emd-21559.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21559_fsc.xml emd_21559_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21559.png emd_21559.png | 226.3 KB | ||
| マスクデータ |  emd_21559_msk_1.map emd_21559_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21559.cif.gz emd-21559.cif.gz | 6.4 KB | ||
| その他 |  emd_21559_additional_1.map.gz emd_21559_additional_1.map.gz emd_21559_half_map_1.map.gz emd_21559_half_map_1.map.gz emd_21559_half_map_2.map.gz emd_21559_half_map_2.map.gz | 192 MB 192.5 MB 192.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21559 http://ftp.pdbj.org/pub/emdb/structures/EMD-21559 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21559 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21559 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21559_validation.pdf.gz emd_21559_validation.pdf.gz | 854.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21559_full_validation.pdf.gz emd_21559_full_validation.pdf.gz | 854 KB | 表示 | |
| XML形式データ |  emd_21559_validation.xml.gz emd_21559_validation.xml.gz | 21.8 KB | 表示 | |
| CIF形式データ |  emd_21559_validation.cif.gz emd_21559_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21559 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21559 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21559 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21559 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21559.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21559.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type IV competence pilus secretin PilQ Relion PostProcess map (postprocess.mrc) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.104 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21559_msk_1.map emd_21559_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
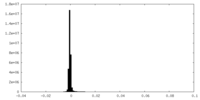
| 密度ヒストグラム |
-追加マップ: Type IV competence pilus secretin PilQ Relion Refine3D...
| ファイル | emd_21559_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type IV competence pilus secretin PilQ Relion Refine3D output map (run_class001.mrc) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
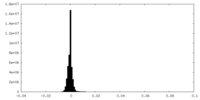
| 密度ヒストグラム |
-ハーフマップ: Type IV competence pilus secretin PilQ Relion Refine3D half map 1
| ファイル | emd_21559_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type IV competence pilus secretin PilQ Relion Refine3D half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
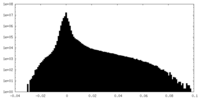
| 密度ヒストグラム |
-ハーフマップ: Type IV competence pilus secretin PilQ Relion Refine3D half map 2
| ファイル | emd_21559_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type IV competence pilus secretin PilQ Relion Refine3D half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
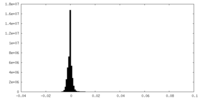
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vibrio cholerae Type IV competence pilus secretin PilQ
| 全体 | 名称: Vibrio cholerae Type IV competence pilus secretin PilQ |
|---|---|
| 要素 |
|
-超分子 #1: Vibrio cholerae Type IV competence pilus secretin PilQ
| 超分子 | 名称: Vibrio cholerae Type IV competence pilus secretin PilQ タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 826 KDa |
-分子 #1: Type IV pilus secretin PilQ family protein
| 分子 | 名称: Type IV pilus secretin PilQ family protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 62.459188 KDa |
| 配列 | 文字列: MRNGLKTYVA QTWLTLWVGL ALCASSTVFS AEFATTNQLE NIDFRVNKEK AAVLIVELAS PSAVVDVQKV QEGLNIELLK TDVADDKLY LLDVKDFSTP VESVEVFRKA PSTQLVVTVD GEFQHDYTLK GKYLEVVISK LKADEKPKPK SVLEKEGKLI S INFQDIPV ...文字列: MRNGLKTYVA QTWLTLWVGL ALCASSTVFS AEFATTNQLE NIDFRVNKEK AAVLIVELAS PSAVVDVQKV QEGLNIELLK TDVADDKLY LLDVKDFSTP VESVEVFRKA PSTQLVVTVD GEFQHDYTLK GKYLEVVISK LKADEKPKPK SVLEKEGKLI S INFQDIPV RNVLQLIADY NGFNLVVSDS VVGNLTLRLD GVPWQQVLDI ILQVKGLDKR VDGNVILIAP KEELDLREKQ AL EKARLAE ELGDLKSEII KINFAKASDI AAMIGGEGNV NMLSERGSIS IDERTNSLLI RELPDNIAVI REIIESLDIP VKQ VQIEAR IVTVKEGNLE ELGVRWGVMS TNGSHSVGGS IESNLWQKGL LADDEFPVDE FLNVNLASTS ANASSIAFQV AKLG SGTLL DLELSALQNE SKAEIISSPR LITTNKQPAY IEQGTEIPYL ESSSSGASTV AFKKAVLSLK VTPQITPDNR LVLDL SVTQ DRRGETVKTG TGEAVSIDTQ RIGTQVLVNN GETVVLGGIF QHSINNSVDK VPLLGDLPVL GALFRRTYEQ MGKSEL LIF VTPKVVIQ UniProtKB: Type IV pilus secretin PilQ family protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 詳細: Pelco EasiGlow, 20 mA, 60 seconds | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293.15 K / 装置: FEI VITROBOT MARK IV / 詳細: blot force -6, blot time 4 s. | |||||||||
| 詳細 | The protein was purified using Ni NTA agarose beads (Anatrace, SUPER-NINTA25) and concentrated to ~1 mg/mL. PilQ was then exchanged into Amphipol A8-35 (0.585 mg for a 3:1 ratio, Anatrace, A835). BioBeads were used to remove excess DDM and the sample was concentrated to ~0.8 mg/mL |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 3808 / 平均露光時間: 3.7 sec. / 平均電子線量: 1.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 30.0 µm / 最小 デフォーカス(公称値): 10.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6w6m: |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X