+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20813 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of full-length, fully glycosylated, non-modified HIV-1 gp160 bound to PG16 Fab at a nominal resolution of 4.6 Angstrom | |||||||||
 マップデータ マップデータ | HIV-1 gp160 bound to PG16 Fab, recommended contour level (absolute as displayed in Chimera): 0.01-0.03 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 / ENV / gp160 / PG16 / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
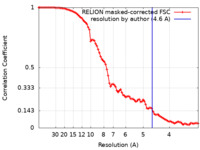
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Pan J / Chen B | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2020 ジャーナル: J Mol Biol / 年: 2020タイトル: Cryo-EM Structure of Full-length HIV-1 Env Bound With the Fab of Antibody PG16. 著者: Junhua Pan / Hanqin Peng / Bing Chen / Stephen C Harrison /  要旨: The HIV-1 envelope protein (Env) is the target of neutralizing antibodies and the template for vaccine immunogen design. The dynamic conformational equilibrium of trimeric Env influences its ...The HIV-1 envelope protein (Env) is the target of neutralizing antibodies and the template for vaccine immunogen design. The dynamic conformational equilibrium of trimeric Env influences its antigenicity and potential immunogenicity. Antibodies that bind at the trimer apex stabilize a "closed" conformation characteristic of the most difficult to neutralize isolates. A goal of vaccine development is therefore to mimic the closed conformation in a designed immunogen. A disulfide-stabilized, trimeric Env ectodomain-the "SOSIP" construct-has many of the relevant properties; it is also particularly suitable for structure determination. Some single-molecule studies have, however, suggested that the SOSIP trimer is not a good representation of Env on the surface of a virion or an infected cell. We isolated Env (fully cleaved to gp120 and gp41) from the surface of expressing cells using tagged, apex-binding Fab PG16 and determined the structure of the PG16-Env complex by cryo-EM to an overall resolution of 4.6 Å. Placing the only purification tag on the Fab ensured that the isolated Env was continuously stabilized in its closed, native conformation. The Env structure in this complex corresponds closely to the SOSIP structures determined by both x-ray crystallography and cryo-EM. Although the membrane-interacting elements are not resolved in our reconstruction, we can make inferences about the connection between ectodomain and membrane-proximal external region (MPER) by reference to the published cryo-tomography structure of an Env "spike" and the NMR structure of the MPER-transmembrane segment. We discuss these results in view of the conflicting interpretations in the literature. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20813.map.gz emd_20813.map.gz | 25.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20813-v30.xml emd-20813-v30.xml emd-20813.xml emd-20813.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20813_fsc.xml emd_20813_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20813.png emd_20813.png | 94.5 KB | ||
| Filedesc metadata |  emd-20813.cif.gz emd-20813.cif.gz | 7.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20813 http://ftp.pdbj.org/pub/emdb/structures/EMD-20813 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20813 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20813 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20813_validation.pdf.gz emd_20813_validation.pdf.gz | 548.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20813_full_validation.pdf.gz emd_20813_full_validation.pdf.gz | 547.8 KB | 表示 | |
| XML形式データ |  emd_20813_validation.xml.gz emd_20813_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_20813_validation.cif.gz emd_20813_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20813 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20813 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20813 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20813 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20813.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20813.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HIV-1 gp160 bound to PG16 Fab, recommended contour level (absolute as displayed in Chimera): 0.01-0.03 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.64 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 GP160 trimer:PG16 Fab complex
| 全体 | 名称: HIV-1 GP160 trimer:PG16 Fab complex |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 GP160 trimer:PG16 Fab complex
| 超分子 | 名称: HIV-1 GP160 trimer:PG16 Fab complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: recombinant protein complex of mature HIV-1 gp160 (cleaved into the receptor/coreceptor binding domain gp120 and the fusogen domain gp41) and antibody PG16 Fab fragment |
|---|---|
| 分子量 | 理論値: 530 KDa |
-超分子 #2: HIV envelope glycoprotein GP160 (mature)
| 超分子 | 名称: HIV envelope glycoprotein GP160 (mature) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 詳細: cleaved form consisting of receptor binding domain GP120 and fusogenic domain GP41 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: Antibody PG16 Fab
| 超分子 | 名称: Antibody PG16 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: envelope glycoprotein gp120,envelope glycoprotein gp120
| 分子 | 名称: envelope glycoprotein gp120,envelope glycoprotein gp120 タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 57.173941 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRVRGIQRNC QHLWRWGTLI LGMLMICSAA ENLWVTVYYG VPVWKDAETT LFCASDAKAY DTEVHNVWAT HACVPTDPNP QEIYMENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPCV QLTPLCVTLD CSYNITNNIT NSITNSSVNM REEIKNCSFN M TTELRDKN ...文字列: MRVRGIQRNC QHLWRWGTLI LGMLMICSAA ENLWVTVYYG VPVWKDAETT LFCASDAKAY DTEVHNVWAT HACVPTDPNP QEIYMENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPCV QLTPLCVTLD CSYNITNNIT NSITNSSVNM REEIKNCSFN M TTELRDKN RKVYSLFYKL DVVQINNGNN SSNLYRLINC NTSALTQACP KVTFEPIPIR YCAPAGYAIL KCNDKEFNGT GL CKNVSTV QCTHGIRPVV STQLLLNGSL AEGKVMIRSE NITNNVKNII VQLNETVTIN CTRPNNNTRK SVRIGPGQTF YAT GDIIGD IRQAHCNVSG SQWNRALHQV VGQLREYWNT TIIFKNSSGG DLEITTHSFN CGGEFFYCNT SGLFNSNWTH NDTA SMKPN DTITLPCRIK QIINMWQRVG QAIYAPPIQG VIRCESNITG LILTRDGGGN INESQIFRPG GGDMRDNWRS ELYKY KVVR IEPLGVAPTK AKRRVVEREK R UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 40.404629 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVVELGAVFI GFLGTAGSTM GAASITLTVQ VRKLLSGIVQ QQSNLLRAIE AQQHLLKLTV WGIKQLQARV LAVERYLRDQ QLLGIWGCS GKLICTTNVP WNSSWSNKSE REIWENMTWL QWDKEISNYT HIIYELIEES QKQQEKNEQE LLELDKWANL W NWFDISNW ...文字列: AVVELGAVFI GFLGTAGSTM GAASITLTVQ VRKLLSGIVQ QQSNLLRAIE AQQHLLKLTV WGIKQLQARV LAVERYLRDQ QLLGIWGCS GKLICTTNVP WNSSWSNKSE REIWENMTWL QWDKEISNYT HIIYELIEES QKQQEKNEQE LLELDKWANL W NWFDISNW LWYIKIFIMI VGGLIGLRIV FAVLSVINRV RQGYSPLSFQ TLTPNPRDPD RPGRIEGEGG EQDRGRSIRL VS GFLALAW DDLRNLCLSS YHQLRDFILI VARTVELLGH SSLKGLRLGW EGLKYLGNLL LYWGRELKTS AINLFDTIAI VVA GWTDRV IEVGQRLGRA ILNIPRRIRQ GLERALL UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: antibody PG16 Fab heavy chain
| 分子 | 名称: antibody PG16 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31.952703 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEFGLSWVFL VALLRGVQCQ EQLVESGGGV VQPGGSLRLS CLASGFTFHK YGMHWVRQAP GKGLEWVALI SDDGMRKYHS DSMWGRVTI SRDNSKNTLY LQFSSLKVED TAMFFCAREA GGPIWHDDVK YYDFNDGYYN YHYMDVWGKG TTVTVSSAST K GPSVFPLA ...文字列: MEFGLSWVFL VALLRGVQCQ EQLVESGGGV VQPGGSLRLS CLASGFTFHK YGMHWVRQAP GKGLEWVALI SDDGMRKYHS DSMWGRVTI SRDNSKNTLY LQFSSLKVED TAMFFCAREA GGPIWHDDVK YYDFNDGYYN YHYMDVWGKG TTVTVSSAST K GPSVFPLA PSSKSTSGGT AALGCLVKDY FPEPVTVSWN SGALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CN VNHKPSN TKVDKRVEPK SCDKASGGGS AWSHPQFEKG GGSGGGSGGS SAWSHPQFEK |
-分子 #4: antibody PG16 Fab light chain
| 分子 | 名称: antibody PG16 Fab light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.424229 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAWALLLLTL LTQGTGSWAQ SALTQPASVS GSPGQTITIS CNGTSSDVGG FDSVSWYQQS PGKAPKVMVF DVSHRPSGIS NRFSGSKSG NTASLTISGL HIEDEGDYFC SSLTDRSHRI FGGGTKVTVL GQPKAAPSVT LFPPSSEELQ ANKATLVCLI S DFYPGAVT ...文字列: MAWALLLLTL LTQGTGSWAQ SALTQPASVS GSPGQTITIS CNGTSSDVGG FDSVSWYQQS PGKAPKVMVF DVSHRPSGIS NRFSGSKSG NTASLTISGL HIEDEGDYFC SSLTDRSHRI FGGGTKVTVL GQPKAAPSVT LFPPSSEELQ ANKATLVCLI S DFYPGAVT VAWKADSSPV KAGVETTTPS KQSNNKYAAS SYLSLTPEQW KSHKSYSCQV THEGSTVEKT VAPTECSGGS GG HHHHHHH HHH |
-分子 #12: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 12 / コピー数: 44 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK I / 詳細: 4.2 second blot before plunging. | ||||||||||||
| 詳細 | This sample was monodisperse (stoichiometry of 3:1 gp160:Fab). |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 2 / 実像数: 8138 / 平均露光時間: 0.125 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最小 デフォーカス(補正後): 0.8 µm / 倍率(補正後): 30488 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 300 |
|---|---|
| 得られたモデル |  PDB-6ulc: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)