+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20301 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EBOV GPdMuc (Makona) in complex with rEBOV-520 and rEBOV-548 Fabs | |||||||||
 マップデータ マップデータ | EBOVGPdMuc (Makona) in complex with rEBOV-520 and rEBOV-548 Fabs | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
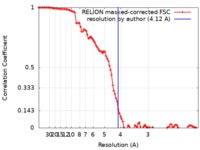
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.12 Å | |||||||||
 データ登録者 データ登録者 | Ward AB / Murin CD / Alkutkar T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2020 ジャーナル: Immunity / 年: 2020タイトル: Analysis of a Therapeutic Antibody Cocktail Reveals Determinants for Cooperative and Broad Ebolavirus Neutralization. 著者: Pavlo Gilchuk / Charles D Murin / Jacob C Milligan / Robert W Cross / Chad E Mire / Philipp A Ilinykh / Kai Huang / Natalia Kuzmina / Pilar X Altman / Sean Hui / Bronwyn M Gunn / Aubrey L ...著者: Pavlo Gilchuk / Charles D Murin / Jacob C Milligan / Robert W Cross / Chad E Mire / Philipp A Ilinykh / Kai Huang / Natalia Kuzmina / Pilar X Altman / Sean Hui / Bronwyn M Gunn / Aubrey L Bryan / Edgar Davidson / Benjamin J Doranz / Hannah L Turner / Tanwee Alkutkar / Robin Flinko / Chiara Orlandi / Robert Carnahan / Rachel Nargi / Robin G Bombardi / Megan E Vodzak / Sheng Li / Adaora Okoli / Morris Ibeawuchi / Benjamin Ohiaeri / George K Lewis / Galit Alter / Alexander Bukreyev / Erica Ollmann Saphire / Thomas W Geisbert / Andrew B Ward / James E Crowe /  要旨: Structural principles underlying the composition of protective antiviral monoclonal antibody (mAb) cocktails are poorly defined. Here, we exploited antibody cooperativity to develop a therapeutic mAb ...Structural principles underlying the composition of protective antiviral monoclonal antibody (mAb) cocktails are poorly defined. Here, we exploited antibody cooperativity to develop a therapeutic mAb cocktail against Ebola virus. We systematically analyzed the antibody repertoire in human survivors and identified a pair of potently neutralizing mAbs that cooperatively bound to the ebolavirus glycoprotein (GP). High-resolution structures revealed that in a two-antibody cocktail, molecular mimicry was a major feature of mAb-GP interactions. Broadly neutralizing mAb rEBOV-520 targeted a conserved epitope on the GP base region. mAb rEBOV-548 bound to a glycan cap epitope, possessed neutralizing and Fc-mediated effector function activities, and potentiated neutralization by rEBOV-520. Remodeling of the glycan cap structures by the cocktail enabled enhanced GP binding and virus neutralization. The cocktail demonstrated resistance to virus escape and protected non-human primates (NHPs) against Ebola virus disease. These data illuminate structural principles of antibody cooperativity with implications for development of antiviral immunotherapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20301.map.gz emd_20301.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20301-v30.xml emd-20301-v30.xml emd-20301.xml emd-20301.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20301_fsc.xml emd_20301_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20301.png emd_20301.png | 143.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20301 http://ftp.pdbj.org/pub/emdb/structures/EMD-20301 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20301 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20301 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20301_validation.pdf.gz emd_20301_validation.pdf.gz | 361.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20301_full_validation.pdf.gz emd_20301_full_validation.pdf.gz | 361.4 KB | 表示 | |
| XML形式データ |  emd_20301_validation.xml.gz emd_20301_validation.xml.gz | 11.6 KB | 表示 | |
| CIF形式データ |  emd_20301_validation.cif.gz emd_20301_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20301 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20301 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20301 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20301 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20301.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20301.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EBOVGPdMuc (Makona) in complex with rEBOV-520 and rEBOV-548 Fabs | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : EBOV GPdMuc (Makona) in complex with rEBOV-520 and rEBOV-548 Fabs
+超分子 #1: EBOV GPdMuc (Makona) in complex with rEBOV-520 and rEBOV-548 Fabs
+超分子 #2: Ebola Virus (Makona) GP
+超分子 #3: rEBOV-548 Fab
+超分子 #4: rEBOV-520 Fab
+分子 #1: Virion spike glycoprotein
+分子 #2: rEBOV-548 Fab heavy chain
+分子 #3: rEBOV-548 Fab light chain
+分子 #4: Virion spike glycoprotein,Virion spike glycoprotein,Ebola Virus (...
+分子 #5: rEBOV-520 Fab light chain
+分子 #6: rEBOV-520 Fab heavy chain
+分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
詳細: Buffers were made fresh from 10X stocks. Detergent was made fresh in TBS as a 6X stock and added immediately prior to vitificaiton. | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Sample was blotted on both sides of the grid. Sample equilibrated in the chamber for 10s before blotting for 5s.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2832 / 平均露光時間: 9.5 sec. / 平均電子線量: 51.85 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー