+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20260 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV Env 16055 NFL TD 2CC+ in complex with antibody 1C2 fragment antigen binding | |||||||||||||||
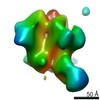
 マップデータ マップデータ | sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | HIV-1 / neutralizing antibody / rabbit antibody / VIRAL PROTEIN / VIRAL PROTEIN-immune system complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.94 Å | |||||||||||||||
 データ登録者 データ登録者 | Ozorowski G / Torres JL | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2019 ジャーナル: Immunity / 年: 2019タイトル: Vaccination with Glycan-Modified HIV NFL Envelope Trimer-Liposomes Elicits Broadly Neutralizing Antibodies to Multiple Sites of Vulnerability. 著者: Viktoriya Dubrovskaya / Karen Tran / Gabriel Ozorowski / Javier Guenaga / Richard Wilson / Shridhar Bale / Christopher A Cottrell / Hannah L Turner / Gemma Seabright / Sijy O'Dell / Jonathan ...著者: Viktoriya Dubrovskaya / Karen Tran / Gabriel Ozorowski / Javier Guenaga / Richard Wilson / Shridhar Bale / Christopher A Cottrell / Hannah L Turner / Gemma Seabright / Sijy O'Dell / Jonathan L Torres / Lifei Yang / Yu Feng / Daniel P Leaman / Néstor Vázquez Bernat / Tyler Liban / Mark Louder / Krisha McKee / Robert T Bailer / Arlette Movsesyan / Nicole A Doria-Rose / Marie Pancera / Gunilla B Karlsson Hedestam / Michael B Zwick / Max Crispin / John R Mascola / Andrew B Ward / Richard T Wyatt /    要旨: The elicitation of broadly neutralizing antibodies (bNAbs) against the HIV-1 envelope glycoprotein (Env) trimer remains a major vaccine challenge. Most cross-conserved protein determinants are ...The elicitation of broadly neutralizing antibodies (bNAbs) against the HIV-1 envelope glycoprotein (Env) trimer remains a major vaccine challenge. Most cross-conserved protein determinants are occluded by self-N-glycan shielding, limiting B cell recognition of the underlying polypeptide surface. The exceptions to the contiguous glycan shield include the conserved receptor CD4 binding site (CD4bs) and glycoprotein (gp)41 elements proximal to the furin cleavage site. Accordingly, we performed heterologous trimer-liposome prime:boosting in rabbits to drive B cells specific for cross-conserved sites. To preferentially expose the CD4bs to B cells, we eliminated proximal N-glycans while maintaining the native-like state of the cleavage-independent NFL trimers, followed by gradual N-glycan restoration coupled with heterologous boosting. This approach successfully elicited CD4bs-directed, cross-neutralizing Abs, including one targeting a unique glycan-protein epitope and a bNAb (87% breadth) directed to the gp120:gp41 interface, both resolved by high-resolution cryoelectron microscopy. This study provides proof-of-principle immunogenicity toward eliciting bNAbs by vaccination. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20260.map.gz emd_20260.map.gz | 85.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20260-v30.xml emd-20260-v30.xml emd-20260.xml emd-20260.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20260_fsc.xml emd_20260_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20260.png emd_20260.png | 182.1 KB | ||
| マスクデータ |  emd_20260_msk_1.map emd_20260_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-20260.cif.gz emd-20260.cif.gz | 7.4 KB | ||
| その他 |  emd_20260_half_map_1.map.gz emd_20260_half_map_1.map.gz emd_20260_half_map_2.map.gz emd_20260_half_map_2.map.gz | 71.3 MB 71.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20260 http://ftp.pdbj.org/pub/emdb/structures/EMD-20260 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20260 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20260 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20260_validation.pdf.gz emd_20260_validation.pdf.gz | 958.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20260_full_validation.pdf.gz emd_20260_full_validation.pdf.gz | 957.9 KB | 表示 | |
| XML形式データ |  emd_20260_validation.xml.gz emd_20260_validation.xml.gz | 17.3 KB | 表示 | |
| CIF形式データ |  emd_20260_validation.cif.gz emd_20260_validation.cif.gz | 22.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20260 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20260 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20260 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20260 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20260.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20260.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20260_msk_1.map emd_20260_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_20260_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_20260_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV Env 16055 NFL TD 2CC+ in complex with antibody 1C2 fragment a...
| 全体 | 名称: HIV Env 16055 NFL TD 2CC+ in complex with antibody 1C2 fragment antigen binding |
|---|---|
| 要素 |
|
-超分子 #1: HIV Env 16055 NFL TD 2CC+ in complex with antibody 1C2 fragment a...
| 超分子 | 名称: HIV Env 16055 NFL TD 2CC+ in complex with antibody 1C2 fragment antigen binding タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 570 KDa |
-分子 #1: HIV Env 16055 NFL TD 2CC+
| 分子 | 名称: HIV Env 16055 NFL TD 2CC+ / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 75.630539 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPMGSLQPLA TLYLLGMLVA SVLANGNLWV TVYYGVPVWK DAETTLFCAS DAKAYEKEKH NVWATHACVP TDPNPQEMVL ENVTENFNM WKNDMVEQMH TDVISLWDQS LKPCVKLTPL CVTLECRQVN TTNATSSVNV TNGEEIKNCS FNATTELRDK K QKVYALFY ...文字列: MPMGSLQPLA TLYLLGMLVA SVLANGNLWV TVYYGVPVWK DAETTLFCAS DAKAYEKEKH NVWATHACVP TDPNPQEMVL ENVTENFNM WKNDMVEQMH TDVISLWDQS LKPCVKLTPL CVTLECRQVN TTNATSSVNV TNGEEIKNCS FNATTELRDK K QKVYALFY RLDIVPLEEE RKGNSSKYRL INCNTSACTQ ACPKVTFDPI PIHYCAPAGY AILKCNNKTF NGTGPCNNVS TV QCTHGIK PVVSTQLLLN GSLAEGEIII RSENLTNNVK TIIVHLNESV EIVCTRPNNY TRKSIRIGPG QTFYATGDII GNI RQAYCN ISKDDWIRTL QRVGKKLAEH FPRRIINFTS PAGGDLEITT HSFNCRGEFF YCNTSSLFNS TYNPNDTNSN SSSS NSSLD ITIPCRIKQI INMWQRVGQC MYAPPIEGNI TCKSNITGLL LVRDGGVESN ETEIFRPGGG DMRNNWRSEL YKYKV VEIK PLGIAPTRCK RRVVEGGGGS GGGGSDDDDK AVGLGAVRRG FLGAAGSTMG AASITLTVQA RQLLSGIVQQ QSNLLK APE AQQHLLQLGV WGIKQLQTRV LAIERYLKDQ QLLGIWGCSG KLICTTAVPW NSSWSNKSHD EIWGNMTWMQ WDREIGN YT NTIYRLLEDS QNQQEQNEKD LLACDGGGGS HHHHHHHH |
-分子 #2: Rabbit antibody 1C2 heavy chain fragment antigen binding
| 分子 | 名称: Rabbit antibody 1C2 heavy chain fragment antigen binding タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 27.476072 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MYRMQLLSCI ALSLALVTNS QCQSLEESGG DLVKPGASLT LTCTASGFSF GWNDYMSWVR QAPGKGLEWI GCIYAGSTRS TYYANWAKG RLTISKTSST AVTLQMTSLT AADTATYFCA RGAVTYDGLG GAYLKHFNLW GPGTLVTVSS GQPKAPSVFP L APCCGDTP ...文字列: MYRMQLLSCI ALSLALVTNS QCQSLEESGG DLVKPGASLT LTCTASGFSF GWNDYMSWVR QAPGKGLEWI GCIYAGSTRS TYYANWAKG RLTISKTSST AVTLQMTSLT AADTATYFCA RGAVTYDGLG GAYLKHFNLW GPGTLVTVSS GQPKAPSVFP L APCCGDTP SSTVTLGCLV KGYLPEPVTV TWNSGTLTNG VRTFPSVRQS SGLYSLSSVV SVTSSSQPVT CNVAHPATNT KV DKTVAPS TCSKHHHHHH HH |
-分子 #3: Rabbit antibody 1C2 kappa chain
| 分子 | 名称: Rabbit antibody 1C2 kappa chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.041023 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MYRMQLLSCI ALSLALVTNS AIKMTQTPSS VSAAVGGTVT VNCRASEDIE SYLAWYQQKP GQPPKLLIYD TSKLASGVPS RFKGSGSGT QFALTISGVQ CDDAATYYCL YGYISSDRID FGFGGGTELV VKGDPVAPSV LIFPPAADQV ATGTVTIVCV A NKYFPDVT ...文字列: MYRMQLLSCI ALSLALVTNS AIKMTQTPSS VSAAVGGTVT VNCRASEDIE SYLAWYQQKP GQPPKLLIYD TSKLASGVPS RFKGSGSGT QFALTISGVQ CDDAATYYCL YGYISSDRID FGFGGGTELV VKGDPVAPSV LIFPPAADQV ATGTVTIVCV A NKYFPDVT VTWEVDGTTQ TTGIENSKTP QNSADCTYNL SSTLTLTSTQ YNSHKEYTCK VTQGTTSVVQ SFNRGDC |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: DDM added to sample shortly (< 5 minutes) before vitrification | ||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 1690 / 平均露光時間: 12.5 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






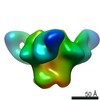
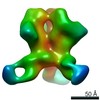
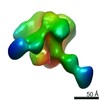

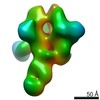
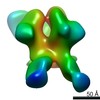
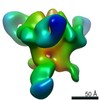
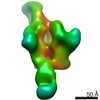
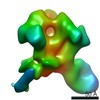













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)















































