+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of coxsackievirus B5 capsid (mutant CVB5F.cas.genogroupB) - A particle | |||||||||
 マップデータ マップデータ | Main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Enterovirus / coxsackievirus / thermostable / mutant / VIRUS | |||||||||
| 生物種 |  Coxsackievirus B5 (コクサッキーウイルス) Coxsackievirus B5 (コクサッキーウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Kumar K / Antanasijevic A | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Environ Sci Technol / 年: 2024 ジャーナル: Environ Sci Technol / 年: 2024タイトル: Influence of Amino Acid Substitutions in Capsid Proteins of Coxsackievirus B5 on Free Chlorine and Thermal Inactivation. 著者: Shotaro Torii / Jérôme Gouttenoire / Kiruthika Kumar / Aleksandar Antanasijevic / Tamar Kohn /  要旨: The sensitivity of enteroviruses to disinfectants varies among genetically similar variants and coincides with amino acid changes in capsid proteins, although the effect of individual substitutions ...The sensitivity of enteroviruses to disinfectants varies among genetically similar variants and coincides with amino acid changes in capsid proteins, although the effect of individual substitutions remains unknown. Here, we employed reverse genetics to investigate how amino acid substitutions in coxsackievirus B5 (CVB5) capsid proteins affect the virus' sensitivity to free chlorine and heat treatment. Of ten amino acid changes observed in CVB5 variants with free chlorine resistance, none significantly reduced the chlorine sensitivity, indicating a minor role of the capsid composition in chlorine sensitivity of CVB5. Conversely, a subset of these amino acid changes located at the C-terminal region of viral protein 1 led to reduced heat sensitivity. Cryo-electron microscopy revealed that these changes affect the assembly of intermediate viral states (altered and empty particles), suggesting that the mechanism for reduced heat sensitivity could be related to improved molecular packing of CVB5, resulting in greater stability or altered dynamics of virus uncoating during infection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18943.map.gz emd_18943.map.gz | 398.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18943-v30.xml emd-18943-v30.xml emd-18943.xml emd-18943.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18943_fsc.xml emd_18943_fsc.xml | 15.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18943.png emd_18943.png | 237.7 KB | ||
| マスクデータ |  emd_18943_msk_1.map emd_18943_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18943.cif.gz emd-18943.cif.gz | 6.7 KB | ||
| その他 |  emd_18943_half_map_1.map.gz emd_18943_half_map_1.map.gz emd_18943_half_map_2.map.gz emd_18943_half_map_2.map.gz | 391.9 MB 391.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18943 http://ftp.pdbj.org/pub/emdb/structures/EMD-18943 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18943 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18943 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18943_validation.pdf.gz emd_18943_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18943_full_validation.pdf.gz emd_18943_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_18943_validation.xml.gz emd_18943_validation.xml.gz | 25 KB | 表示 | |
| CIF形式データ |  emd_18943_validation.cif.gz emd_18943_validation.cif.gz | 32.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18943 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18943 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18943 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18943 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18943.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18943.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.926 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18943_msk_1.map emd_18943_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
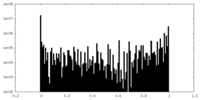
| 密度ヒストグラム |
-ハーフマップ: Half-Map A
| ファイル | emd_18943_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
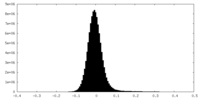
| 密度ヒストグラム |
-ハーフマップ: Half-Map B
| ファイル | emd_18943_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Coxsackievirus B5
| 全体 | 名称:  Coxsackievirus B5 (コクサッキーウイルス) Coxsackievirus B5 (コクサッキーウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Coxsackievirus B5
| 超分子 | 名称: Coxsackievirus B5 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Particle A state of the CVB5 virion capsid / NCBI-ID: 12074 / 生物種: Coxsackievirus B5 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid VP1-4 / 直径: 300.0 Å |
-分子 #1: Coxsackievirus B5 (mutant CVB5F.cas.genogroupB) in particle A sta...
| 分子 | 名称: Coxsackievirus B5 (mutant CVB5F.cas.genogroupB) in particle A state - VP1 タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Coxsackievirus B5 (コクサッキーウイルス) Coxsackievirus B5 (コクサッキーウイルス) |
| 分子量 | 理論値: 93.795359 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGAQVSTQKT GAHETGLNAS GNSIIHYTNM NYYKDSASNS ANRQEFAQDP GKFTEPVKDI MIKSMPALNS PSAEECGYSD RVRSITLGN STITTQECAN VVVGYGTWPT YLRDEEATAE DQPTQPDVAT CRFYTLESVM WQQSSPGWWW KFPDALSNMG L FGQNMQYH ...文字列: MGAQVSTQKT GAHETGLNAS GNSIIHYTNM NYYKDSASNS ANRQEFAQDP GKFTEPVKDI MIKSMPALNS PSAEECGYSD RVRSITLGN STITTQECAN VVVGYGTWPT YLRDEEATAE DQPTQPDVAT CRFYTLESVM WQQSSPGWWW KFPDALSNMG L FGQNMQYH YLGRAGYTLH VQCNASKFHQ GCLLVVCVPE AEMGCATIAN KPDQKSLSNG ETANVFDSQN TSGQTAVQAN VI NAGMGIG VGNLTIFPHQ WINLRTNNSA TIVMPYVNSV PMDNMFRHNN FTLMIIPFAP LSYSTGATTY VPITVTVAPM CAE YNGLRL AGRQGLPTML TPGSNQFLTS DDFQSPSAMP QFDVTPEMAI PGQVNNLMEI AEVDSVVPVN NTEGKVMSIE AYQI PVQSN STNGSQVFGF PLIPGASSVL NRTLLGEILN YYTHWSGSIK LTFMFCGSAM ATGKFLLAYS PPGAGAPTTR KEAML GTHV IWDVGLQSSC VLCIPWISQT HYRYVVMDEY TAGGYITCWY QTNIVVPADT QSDCKILCFV SACNDFSVRM LKDTPF IKQ DSFFQGPPGE AIERAIARVA DTISSGPVNS ESIPALTAAE TGHTSQVVPA DTMQTRHVKN YHSRSESTVE NFLCRSA CV YYTTYKNHGT DGDNFAQWVI NTRQVAQLRR KLEMFTYARF DLELTFVITS SQEQSTIKGQ DSPVLTHQIM YVPPGGPV P TKINSYSWQT STNPSVFWTE GSAPPRISIP FISIGNAYSM FYDGWARFDK QGTYGINTLN NMGTLYMRHV NDGSPGPIV STVRIYFKPK HVKTWVPRPP RLCQYQKAGN VNFEPSGVTE GRTEITAMQT T |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: TBS | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV 詳細: 3ul of sample applied. Blotting time varied. Blotting force = 0. Total blots = 1.. | |||||||||
| 詳細 | Inactivated by formaldehyde. Purified using a combination of sucrose gradient and size-exclusion chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 2 / 実像数: 6653 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model / 詳細: Asymmetric unit used for fitting and interpretation |
|---|---|
| 詳細 | Initial fitting was performed in Chimera and model refinement was performed in Coot and Rosetta. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8r5y: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)