+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Neck of phage 812 virion (C6) | |||||||||
 マップデータ マップデータ | Neck of phage 812 virion (C6) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | phage / neck / portal / connector / VIRUS | |||||||||
| 機能・相同性 | Non-cytoplasmic protein / Non-cytoplasmic protein / Neck protein / Capsid protein / Baseplate hub assembly protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Staphylococcus phage 812 (ファージ) Staphylococcus phage 812 (ファージ) | |||||||||
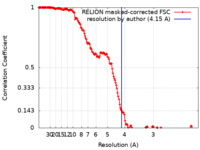
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.15 Å | |||||||||
 データ登録者 データ登録者 | Cienikova Z / Novacek J / Fuzik T / Benesik M / Plevka P | |||||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Genome anchoring, retention, and release by neck proteins of Herelleviridae phage 812 著者: Cienikova Z / Novacek J / Siborova M / Popelarova B / Fuzik T / Benesik M / Bardy P / Plevka P | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18462.map.gz emd_18462.map.gz | 34.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18462-v30.xml emd-18462-v30.xml emd-18462.xml emd-18462.xml | 23.5 KB 23.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18462_fsc.xml emd_18462_fsc.xml | 17 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18462.png emd_18462.png | 87.3 KB | ||
| マスクデータ |  emd_18462_msk_1.map emd_18462_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18462.cif.gz emd-18462.cif.gz | 7.1 KB | ||
| その他 |  emd_18462_half_map_1.map.gz emd_18462_half_map_1.map.gz emd_18462_half_map_2.map.gz emd_18462_half_map_2.map.gz | 330.4 MB 330.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18462 http://ftp.pdbj.org/pub/emdb/structures/EMD-18462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18462 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18462_validation.pdf.gz emd_18462_validation.pdf.gz | 835 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18462_full_validation.pdf.gz emd_18462_full_validation.pdf.gz | 834.6 KB | 表示 | |
| XML形式データ |  emd_18462_validation.xml.gz emd_18462_validation.xml.gz | 24.8 KB | 表示 | |
| CIF形式データ |  emd_18462_validation.cif.gz emd_18462_validation.cif.gz | 33.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18462 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18462 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qkhMC  8q01C  8q1iC  8q7dC  8qekC  8qemC  8qgrC  8qjeC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18462.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18462.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Neck of phage 812 virion (C6) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
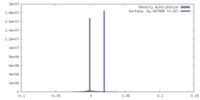
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18462_msk_1.map emd_18462_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
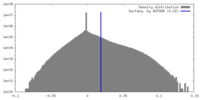
| 密度ヒストグラム |
-ハーフマップ: half-map 2
| ファイル | emd_18462_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map 1
| ファイル | emd_18462_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
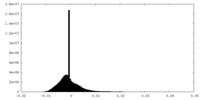
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Staphylococcus phage 812
| 全体 | 名称:  Staphylococcus phage 812 (ファージ) Staphylococcus phage 812 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Staphylococcus phage 812
| 超分子 | 名称: Staphylococcus phage 812 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 / 詳細: Purified phage virion / NCBI-ID: 307898 / 生物種: Staphylococcus phage 812 / Sci species strain: K1-420 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 1100.0 Å |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 33.757332 KDa |
| 配列 | 文字列: MEKPYMIGAN SNPNVINKST TYTTTTQADE QDKPKYTTRL EFDTIDMIRF INDRGIKVLW EEAYFCPCLN PDTGHPRVDC PRCHGKGIA YLPPKETIMA IQSQEKGTNQ LDIGILDTGT AIGTTQLEKR ISYRDRFTVP EVLMPQQMIY FVNKDRIKKG I PLYYDVKE ...文字列: MEKPYMIGAN SNPNVINKST TYTTTTQADE QDKPKYTTRL EFDTIDMIRF INDRGIKVLW EEAYFCPCLN PDTGHPRVDC PRCHGKGIA YLPPKETIMA IQSQEKGTNQ LDIGILDTGT AIGTTQLEKR ISYRDRFTVP EVLMPQQMIY FVNKDRIKKG I PLYYDVKE ITYIATQDGT VYEEDYEIKN NRLYLNEKYE NHTVTLKILM TLRYVVSDIL KESRYQYTKF NQPKSKFENL PQ KLLLKRE DVIVLQDPYK VNDGIEEDLE IQVDDPKASA SNPSNLGGFF GGAFK UniProtKB: Capsid protein |
-分子 #2: Baseplate hub assembly protein
| 分子 | 名称: Baseplate hub assembly protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 31.79968 KDa |
| 配列 | 文字列: MAITSVDSYL LSEIKPRLNT VLENCYIIDE VLKDFDYQTR ESFKEAFCGK NAQHEVTVGF NFPKFKNNYE AHYLIQLGQG QETKNSLGS IQSSYFEATG DTLVESSTAI REDDKLVFTV SKPIGELIKV EDIEFAKYDN LQVEGNKVSF KYQTNEDYEN Y NANIIFTE ...文字列: MAITSVDSYL LSEIKPRLNT VLENCYIIDE VLKDFDYQTR ESFKEAFCGK NAQHEVTVGF NFPKFKNNYE AHYLIQLGQG QETKNSLGS IQSSYFEATG DTLVESSTAI REDDKLVFTV SKPIGELIKV EDIEFAKYDN LQVEGNKVSF KYQTNEDYEN Y NANIIFTE KKNDSKGLVK GFTVEEQVTV VGLSFNVDVA RCLDAVLKMI LISMRDSIEE QQTFQLQNLS FGDIAPIIED GD SMIFGRP TIIKYTSSLD LDYTITQDIN KLTFKERKDW K UniProtKB: Baseplate hub assembly protein |
-分子 #3: Putative non-cytoplasmic protein
| 分子 | 名称: Putative non-cytoplasmic protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 10.146707 KDa |
| 配列 | 文字列: MSIEKKEEVI AHNEVVFRSL TQGLYVKEVD IYSDVVSYTK DVDEALAMPN TINFKNSRKY KKLIMNLDLE PLNKIQKVIY ETHLEGL UniProtKB: Non-cytoplasmic protein |
-分子 #4: Non-cytoplasmic protein
| 分子 | 名称: Non-cytoplasmic protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 17.885197 KDa |
| 配列 | 文字列: MADEISLNPI QDAKPIDDIV DIMTYLKNGK VLRVKQDNQG DILVRMSPGK HKFTEVSRDL DKESFYYKRH WVLYNVSVNS LITFDVYLD EEYSETTKVK YPKDTIVEYT REDQEKDVAM IKEILTDNNG NYFYALIGET MLFDENKLNK VKD UniProtKB: Non-cytoplasmic protein |
-分子 #5: Putative neck protein
| 分子 | 名称: Putative neck protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 34.191703 KDa |
| 配列 | 文字列: MVNSMFGGDL DPYEKSLNYE YPYHPSGNPK HIDVSEIDNL TLADYGWSPD AVKAYMFGIV VQNPDTGQPM GDEFYNHILE RAVGKAERA LDISILPDTQ HEMRDYHETE FNSYMFVHAY RKPILQVENL QLQFNGRPIY KYPANWWKVE HLAGHVQLFP T ALMQTGQS ...文字列: MVNSMFGGDL DPYEKSLNYE YPYHPSGNPK HIDVSEIDNL TLADYGWSPD AVKAYMFGIV VQNPDTGQPM GDEFYNHILE RAVGKAERA LDISILPDTQ HEMRDYHETE FNSYMFVHAY RKPILQVENL QLQFNGRPIY KYPANWWKVE HLAGHVQLFP T ALMQTGQS MSYDAVFNGY PQLAGVYPPS GATFAPQMIR LEYVSGMLPR KKAGRNKPWE MPPELEQLVI KYALKEIYQV WG NLIIGAG IANKTLEVDG ITETIGTTQS AMYGGASAQI LQINEDIKEL LDGLRAYFGY NMIGL UniProtKB: Neck protein |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) デジタル化 - サイズ - 横: 4000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / 実像数: 30553 / 平均露光時間: 1.0 sec. / 平均電子線量: 49.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL 当てはまり具合の基準: cross-correlation coefficient | ||||||
| 得られたモデル |  PDB-8qkh: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)