+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Neck of phage 812 after tail contraction (C12) | |||||||||
 マップデータ マップデータ | Neck of phage 812 after tail contraction (C12) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | phage / neck / portal / connector / VIRUS | |||||||||
| 機能・相同性 | Bacteriophage/Gene transfer agent portal protein / Phage portal protein / viral capsid / Portal protein / Neck protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Staphylococcus phage 812 (ファージ) Staphylococcus phage 812 (ファージ) | |||||||||
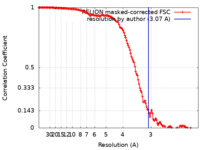
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.07 Å | |||||||||
 データ登録者 データ登録者 | Cienikova Z / Siborova M / Fuzik T / Plevka P | |||||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Genome anchoring, retention, and release by neck proteins of Herelleviridae phage 812 著者: Cienikova Z / Novacek J / Siborova M / Popelarova B / Fuzik T / Benesik M / Bardy P / Plevka P | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18213.map.gz emd_18213.map.gz | 22.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18213-v30.xml emd-18213-v30.xml emd-18213.xml emd-18213.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18213_fsc.xml emd_18213_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18213.png emd_18213.png | 72.5 KB | ||
| マスクデータ |  emd_18213_msk_1.map emd_18213_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18213.cif.gz emd-18213.cif.gz | 7.1 KB | ||
| その他 |  emd_18213_half_map_1.map.gz emd_18213_half_map_1.map.gz emd_18213_half_map_2.map.gz emd_18213_half_map_2.map.gz | 164.9 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18213 http://ftp.pdbj.org/pub/emdb/structures/EMD-18213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18213 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18213_validation.pdf.gz emd_18213_validation.pdf.gz | 898 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18213_full_validation.pdf.gz emd_18213_full_validation.pdf.gz | 897.6 KB | 表示 | |
| XML形式データ |  emd_18213_validation.xml.gz emd_18213_validation.xml.gz | 21.3 KB | 表示 | |
| CIF形式データ |  emd_18213_validation.cif.gz emd_18213_validation.cif.gz | 28.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18213 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18213 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18213 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18213 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q7dMC  8q01C  8q1iC  8qekC  8qemC  8qgrC  8qjeC  8qkhC  8r5gC  8r69C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18213.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18213.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Neck of phage 812 after tail contraction (C12) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.057 Å | ||||||||||||||||||||||||||||||||||||
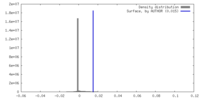
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18213_msk_1.map emd_18213_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
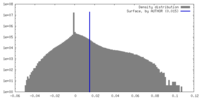
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Staphylococcus phage 812
| 全体 | 名称:  Staphylococcus phage 812 (ファージ) Staphylococcus phage 812 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Staphylococcus phage 812
| 超分子 | 名称: Staphylococcus phage 812 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Purified phage virion was incubated in urea and LTA to induce tail contraction and genome ejection NCBI-ID: 307898 / 生物種: Staphylococcus phage 812 / Sci species strain: K1-420 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 1100.0 Å |
-分子 #1: Putative neck protein
| 分子 | 名称: Putative neck protein / タイプ: protein_or_peptide / ID: 1 / 詳細: Adaptor protein / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 34.191703 KDa |
| 配列 | 文字列: MVNSMFGGDL DPYEKSLNYE YPYHPSGNPK HIDVSEIDNL TLADYGWSPD AVKAYMFGIV VQNPDTGQPM GDEFYNHILE RAVGKAERA LDISILPDTQ HEMRDYHETE FNSYMFVHAY RKPILQVENL QLQFNGRPIY KYPANWWKVE HLAGHVQLFP T ALMQTGQS ...文字列: MVNSMFGGDL DPYEKSLNYE YPYHPSGNPK HIDVSEIDNL TLADYGWSPD AVKAYMFGIV VQNPDTGQPM GDEFYNHILE RAVGKAERA LDISILPDTQ HEMRDYHETE FNSYMFVHAY RKPILQVENL QLQFNGRPIY KYPANWWKVE HLAGHVQLFP T ALMQTGQS MSYDAVFNGY PQLAGVYPPS GATFAPQMIR LEYVSGMLPR KKAGRNKPWE MPPELEQLVI KYALKEIYQV WG NLIIGAG IANKTLEVDG ITETIGTTQS AMYGGASAQI LQINEDIKEL LDGLRAYFGY NMIGL UniProtKB: Neck protein |
-分子 #2: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 64.155684 KDa |
| 配列 | 文字列: MADLFKQFRL GKDYGNNSTI AQVPIDEGLQ ANIKKIEQDN KEYQDLTKSL YGQQQAYAEP FIEMMDTNPE FRDKRSYMKN EHNLHDVLK KFGNNPILNA IILTRSNQVA MYCQPARYSE KGLGFEVRLR DLDAEPGRKE KEEMKRIEDF IVNTGKDKDV D RDSFQTFC ...文字列: MADLFKQFRL GKDYGNNSTI AQVPIDEGLQ ANIKKIEQDN KEYQDLTKSL YGQQQAYAEP FIEMMDTNPE FRDKRSYMKN EHNLHDVLK KFGNNPILNA IILTRSNQVA MYCQPARYSE KGLGFEVRLR DLDAEPGRKE KEEMKRIEDF IVNTGKDKDV D RDSFQTFC KKIVRDTYIY DQVNFEKVFN KNNKTKLEKF IAVDPSTIFY ATDKKGKIIK GGKRFVQVVD KRVVASFTSR EL AMGIRNP RTELSSSGYG LSEVEIAMKE FIAYNNTESF NDRFFSHGGT TRGILQIRSD QQQSQHALEN FKREWKSSLS GIN GSWQIP VVMADDIKFV NMTPTANDMQ FEKWLNYLIN IISALYGIDP AEIGFPNRGG ATGSKGGSTL NEADPGKKQQ QSQN KGLQP LLRFIEDLVN RHIISEYGDK YTFQFVGGDT KSATDKLNIL KLETQIFKTV NEAREEQGKK PIEGGDIILD ASFLQ GTAQ LQQDKQYNDG KQKERLQMMM SLLEGDNDDS EEGQSTDSSN DDKEIGTDAQ IKGDDNVYRT QTSNKGQGRK GEKSSD FKH UniProtKB: Portal protein |
-分子 #3: DNA forward strand (120-MER)
| 分子 | 名称: DNA forward strand (120-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 36.24227 KDa |
| 配列 | 文字列: (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT)(DC) (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT)(DC) (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT) (DC)(DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT) (DC) (DT)(DA)(DC)(DA)(DC) ...文字列: (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT)(DC) (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT)(DC) (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT) (DC)(DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA)(DT) (DC) (DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA) (DT)(DC)(DT)(DA)(DC)(DA)(DC)(DT)(DT)(DA) (DT)(DC) (DT)(DA)(DC)(DA)(DC)(DT)(DT) (DA)(DT)(DC)(DT)(DA)(DC)(DA)(DC)(DT)(DT) (DA)(DT)(DC) (DT)(DA)(DC)(DA)(DC)(DT) (DT)(DA)(DT)(DC)(DT)(DA)(DC)(DA)(DC)(DT) (DT)(DA)(DT)(DC) (DT)(DA)(DC)(DA)(DC) (DT)(DT)(DA)(DT)(DC)(DT)(DA)(DC)(DA)(DC) (DT)(DT)(DA)(DT)(DC) |
-分子 #4: DNA reverse strand (120-MER)
| 分子 | 名称: DNA reverse strand (120-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 812 (ファージ) / 株: K1-420 Staphylococcus phage 812 (ファージ) / 株: K1-420 |
| 分子量 | 理論値: 37.79132 KDa |
| 配列 | 文字列: (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT)(DA) (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT)(DA) (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT) (DA)(DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT) (DA) (DG)(DA)(DT)(DA)(DA) ...文字列: (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT)(DA) (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT)(DA) (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT) (DA)(DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG)(DT) (DA) (DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG) (DT)(DA)(DG)(DA)(DT)(DA)(DA)(DG)(DT)(DG) (DT)(DA) (DG)(DA)(DT)(DA)(DA)(DG)(DT) (DG)(DT)(DA)(DG)(DA)(DT)(DA)(DA)(DG)(DT) (DG)(DT)(DA) (DG)(DA)(DT)(DA)(DA)(DG) (DT)(DG)(DT)(DA)(DG)(DA)(DT)(DA)(DA)(DG) (DT)(DG)(DT)(DA) (DG)(DA)(DT)(DA)(DA) (DG)(DT)(DG)(DT)(DA)(DG)(DA)(DT)(DA)(DA) (DG)(DT)(DG)(DT)(DA) |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 4000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / 撮影したグリッド数: 1 / 実像数: 15371 / 平均露光時間: 7.0 sec. / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL 当てはまり具合の基準: cross-correlation coefficient |
| 得られたモデル |  PDB-8q7d: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)