+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1811 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast 80S ribosome stalled by a stem-loop containing mRNA in complex with Dom34-Hbs1. The dataset is computationally sorted for presence of P-site tRNA and Dom34-Hbs1. | |||||||||
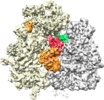
 マップデータ マップデータ | This map represents a cryo-EM reconstruction of yeast 80S ribosome stalled by a stable stem-loop structure in complex with Dom34 and Hbs1. Additionally, it contains a P-site tRNA. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ribosome / stalling / mRNA / P-site tRNA / no-go mRNA decay | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Eukaryotic Translation Elongation / RNA surveillance / Dom34-Hbs1 complex / nuclear-transcribed mRNA catabolic process, no-go decay / nuclear-transcribed mRNA catabolic process, non-stop decay / HSF1 activation / ribosome disassembly / Protein methylation / nonfunctional rRNA decay / positive regulation of translational initiation ...Eukaryotic Translation Elongation / RNA surveillance / Dom34-Hbs1 complex / nuclear-transcribed mRNA catabolic process, no-go decay / nuclear-transcribed mRNA catabolic process, non-stop decay / HSF1 activation / ribosome disassembly / Protein methylation / nonfunctional rRNA decay / positive regulation of translational initiation / translation elongation factor activity / Neutrophil degranulation / RNA endonuclease activity / rescue of stalled ribosome / positive regulation of translation / meiotic cell cycle / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / translation / cell division / GTPase activity / GTP binding / metal ion binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.5 Å | |||||||||
 データ登録者 データ登録者 | Becker T / Armache JP / Anger AM / Jarasch A / Villa E / Sieber H / AbdelMotaal B / Berninghausen O / Mielke T / Beckmann R | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2011 ジャーナル: Nat Struct Mol Biol / 年: 2011タイトル: Structure of the no-go mRNA decay complex Dom34-Hbs1 bound to a stalled 80S ribosome. 著者: Thomas Becker / Jean-Paul Armache / Alexander Jarasch / Andreas M Anger / Elizabeth Villa / Heidemarie Sieber / Basma Abdel Motaal / Thorsten Mielke / Otto Berninghausen / Roland Beckmann /  要旨: No-go decay (NGD) is a mRNA quality-control mechanism in eukaryotic cells that leads to degradation of mRNAs stalled during translational elongation. The key factors triggering NGD are Dom34 and Hbs1. ...No-go decay (NGD) is a mRNA quality-control mechanism in eukaryotic cells that leads to degradation of mRNAs stalled during translational elongation. The key factors triggering NGD are Dom34 and Hbs1. We used cryo-EM to visualize NGD intermediates resulting from binding of the Dom34-Hbs1 complex to stalled ribosomes. At subnanometer resolution, all domains of Dom34 and Hbs1 were identified, allowing the docking of crystal structures and homology models. Moreover, the close structural similarity of Dom34 and Hbs1 to eukaryotic release factors (eRFs) enabled us to propose a model for the ribosome-bound eRF1-eRF3 complex. Collectively, our data provide structural insights into how stalled mRNA is recognized on the ribosome and how the eRF complex can simultaneously recognize stop codons and catalyze peptide release. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1811.map.gz emd_1811.map.gz | 28.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1811-v30.xml emd-1811-v30.xml emd-1811.xml emd-1811.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1811.gif EMD-1811.gif | 122.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1811 http://ftp.pdbj.org/pub/emdb/structures/EMD-1811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1811 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1811_validation.pdf.gz emd_1811_validation.pdf.gz | 393 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1811_full_validation.pdf.gz emd_1811_full_validation.pdf.gz | 392.5 KB | 表示 | |
| XML形式データ |  emd_1811_validation.xml.gz emd_1811_validation.xml.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1811 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1811 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1811 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1811 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1811.map.gz / 形式: CCP4 / 大きさ: 185.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1811.map.gz / 形式: CCP4 / 大きさ: 185.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This map represents a cryo-EM reconstruction of yeast 80S ribosome stalled by a stable stem-loop structure in complex with Dom34 and Hbs1. Additionally, it contains a P-site tRNA. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2375 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Stem-loop stalled yeast 80S ribosome in complex with Dom34-Hbs1 a...
| 全体 | 名称: Stem-loop stalled yeast 80S ribosome in complex with Dom34-Hbs1 and P-site tRNA. |
|---|---|
| 要素 |
|
-超分子 #1000: Stem-loop stalled yeast 80S ribosome in complex with Dom34-Hbs1 a...
| 超分子 | 名称: Stem-loop stalled yeast 80S ribosome in complex with Dom34-Hbs1 and P-site tRNA. タイプ: sample / ID: 1000 詳細: Mammalian Sec61 was added to saturate the hydrophobic signal sequence present in the nascent polypeptide chain. 集合状態: One ribosome / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 3.3 MDa |
-超分子 #1: Saccharomyces cerevisiae 80S ribosome
| 超分子 | 名称: Saccharomyces cerevisiae 80S ribosome / タイプ: complex / ID: 1 / Name.synonym: yeast 80S ribosome 詳細: The mRNA stem-loop structure is not visible in the Cryo-EM reconstruction indicating its flexibility Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 分子量 | 実験値: 3.2 MDa / 理論値: 3.2 MDa |
-分子 #1: Hbs1p
| 分子 | 名称: Hbs1p / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Hbs1p / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 68 KDa / 理論値: 68 KDa |
| 組換発現 | 生物種:  |
-分子 #2: Dom34p
| 分子 | 名称: Dom34p / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Dom34p / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 44 KDa / 理論値: 44 KDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 7 詳細: 20 mM Tris/HCl, pH 7.0, 80 mM NaCl, 97 mM KOAc, 10 mM Mg(OAc)2, 1.5 mM DTT, 0.02 % Nikkol, 1.8 % Glycerol, 0.01 mg/ml Cycloheximide, 500 0.5 mM GDPNP, 0.3 % Digitonin |
| グリッド | 詳細: Quantifoil Grid with 2 nm carbon on top |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot 手法: Blotted for 10 seconds before plunging, used 2 layer of filter paper |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 平均: 84 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100000 times magnification |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - サンプリング間隔: 4.76 µm / 実像数: 78 / 平均電子線量: 25 e/Å2 詳細: Scanned with a Heidelberg PrimeScan drum scanner at 5334 dpi Od range: 1.2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 38000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 3.78 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 39000 |
| 試料ステージ | 試料ホルダー: FEI Polara Cartridge System / 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Mammalian Sec61 complex was added to the sample to saturate the hydrophobic nascent chain |
|---|---|
| CTF補正 | 詳細: CTF correction on the level of 3D volumes (SPIDER TF CTS command) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER 詳細: The dataset was sorted according to presence of Dom34-Hbs1 complex and P-site tRNA. 使用した粒子像数: 38400 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Molecular Dynamics based flexible fitting MDFF |
| 詳細 | Rigid body fitting of individual domains using Coot followed by MDFF |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3izq: |
 ムービー
ムービー コントローラー
コントローラー
































 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)























