+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (CI membrane core) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Plant / Mitochondria / Complex / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plastid outer membrane / TIM22 mitochondrial import inner membrane insertion complex / anther dehiscence / vegetative to reproductive phase transition of meristem / chloroplast outer membrane / P450-containing electron transport chain / 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; デヒドラターゼ類 / NADH dehydrogenase complex / photorespiration / response to abscisic acid ...plastid outer membrane / TIM22 mitochondrial import inner membrane insertion complex / anther dehiscence / vegetative to reproductive phase transition of meristem / chloroplast outer membrane / P450-containing electron transport chain / 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; デヒドラターゼ類 / NADH dehydrogenase complex / photorespiration / response to abscisic acid / protein insertion into mitochondrial inner membrane / embryo development ending in seed dormancy / plant-type vacuole / regulation of reactive oxygen species metabolic process / porin activity / plastid / ubiquinone binding / pore complex / protein homotrimerization / NADH:ubiquinone reductase (H+-translocating) / NADH dehydrogenase activity / respiratory chain complex I / : / mitochondrial electron transport, NADH to ubiquinone / electron transport coupled proton transport / NADH dehydrogenase (ubiquinone) activity / ATP synthesis coupled electron transport / : / monoatomic ion transport / response to salt stress / aerobic respiration / chloroplast / carbonate dehydratase activity / mitochondrial membrane / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / peroxisome / mitochondrial outer membrane / mitochondrial inner membrane / copper ion binding / nucleolus / protein homodimerization activity / mitochondrion / extracellular region / identical protein binding / nucleus / metal ion binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
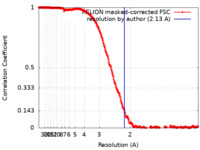
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.13 Å | |||||||||
 データ登録者 データ登録者 | Klusch N / Kuehlbrandt W | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2023 ジャーナル: Nat Plants / 年: 2023タイトル: Cryo-EM structure of the respiratory I + III supercomplex from Arabidopsis thaliana at 2 Å resolution. 著者: Niklas Klusch / Maximilian Dreimann / Jennifer Senkler / Nils Rugen / Werner Kühlbrandt / Hans-Peter Braun /  要旨: Protein complexes of the mitochondrial respiratory chain assemble into respiratory supercomplexes. Here we present the high-resolution electron cryo-microscopy structure of the Arabidopsis ...Protein complexes of the mitochondrial respiratory chain assemble into respiratory supercomplexes. Here we present the high-resolution electron cryo-microscopy structure of the Arabidopsis respiratory supercomplex consisting of complex I and a complex III dimer, with a total of 68 protein subunits and numerous bound cofactors. A complex I-ferredoxin, subunit B14.7 and P9, a newly defined subunit of plant complex I, mediate supercomplex formation. The component complexes stabilize one another, enabling new detailed insights into their structure. We describe (1) an interrupted aqueous passage for proton translocation in the membrane arm of complex I; (2) a new coenzyme A within the carbonic anhydrase module of plant complex I defining a second catalytic centre; and (3) the water structure at the proton exit pathway of complex III with a co-purified ubiquinone in the Q site. We propose that the main role of the plant supercomplex is to stabilize its components in the membrane. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16000.map.gz emd_16000.map.gz | 91.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16000-v30.xml emd-16000-v30.xml emd-16000.xml emd-16000.xml | 44.3 KB 44.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16000_fsc.xml emd_16000_fsc.xml | 26.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16000.png emd_16000.png | 151.6 KB | ||
| Filedesc metadata |  emd-16000.cif.gz emd-16000.cif.gz | 10.6 KB | ||
| その他 |  emd_16000_additional_1.map.gz emd_16000_additional_1.map.gz emd_16000_half_map_1.map.gz emd_16000_half_map_1.map.gz emd_16000_half_map_2.map.gz emd_16000_half_map_2.map.gz | 94.9 MB 1.1 GB 1.1 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16000 http://ftp.pdbj.org/pub/emdb/structures/EMD-16000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16000 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16000_validation.pdf.gz emd_16000_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16000_full_validation.pdf.gz emd_16000_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_16000_validation.xml.gz emd_16000_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_16000_validation.cif.gz emd_16000_validation.cif.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16000 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16000 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8befMC  8bedC  8beeC  8behC  8belC  8bepC  8bpxC  8bq5C  8bq6C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16000.map.gz / 形式: CCP4 / 大きさ: 98.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16000.map.gz / 形式: CCP4 / 大きさ: 98.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.573 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_16000_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
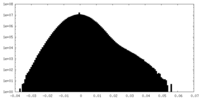
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16000_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16000_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Mitochondrial Arabidopsis thaliana I+III2 supercomplex (CI membra...
+超分子 #1: Mitochondrial Arabidopsis thaliana I+III2 supercomplex (CI membra...
+分子 #1: NADH-ubiquinone oxidoreductase chain 3
+分子 #2: NADH-ubiquinone oxidoreductase chain 1
+分子 #3: NADH-ubiquinone oxidoreductase chain 6
+分子 #4: NADH-ubiquinone oxidoreductase chain 4L
+分子 #5: NADH-ubiquinone oxidoreductase chain 5
+分子 #6: NADH-ubiquinone oxidoreductase chain 4
+分子 #7: NADH-ubiquinone oxidoreductase chain 2
+分子 #8: AT3G07480.1
+分子 #9: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 8-B
+分子 #10: Outer envelope pore protein 16-3, chloroplastic/mitochondrial
+分子 #11: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 13-A
+分子 #12: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 1
+分子 #13: At2g46540/F11C10.23
+分子 #14: Excitatory amino acid transporter
+分子 #15: NADH dehydrogenase [ubiquinone] iron-sulfur protein 5-B
+分子 #16: At4g16450
+分子 #17: P1
+分子 #18: Uncharacterized protein At1g67785
+分子 #19: Uncharacterized protein At2g27730, mitochondrial
+分子 #20: Gamma carbonic anhydrase-like 2, mitochondrial
+分子 #21: Gamma carbonic anhydrase 2, mitochondrial
+分子 #22: Gamma carbonic anhydrase 1, mitochondrial
+分子 #23: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
+分子 #24: Ubiquinone-9
+分子 #25: PHOSPHATIDYLETHANOLAMINE
+分子 #26: 2-{[(4-O-alpha-D-glucopyranosyl-alpha-D-glucopyranosyl)oxy]methyl...
+分子 #27: FE (III) ION
+分子 #28: 2,3-DIMETHOXY-5-METHYL-6-(3,11,15,19-TETRAMETHYL-EICOSA-2,6,10,14...
+分子 #29: (7S)-4-HYDROXY-N,N,N-TRIMETHYL-9-OXO-7-[(PALMITOYLOXY)METHYL]-3,5...
+分子 #30: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE
+分子 #31: CARDIOLIPIN
+分子 #32: ZINC ION
+分子 #33: Butyryl Coenzyme A
+分子 #34: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.18 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: CONTINUOUS / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: GRAPHENE / 支持フィルム - #1 - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 215000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| ソフトウェア | 名称:  Coot (ver. 0.9.5) Coot (ver. 0.9.5) |
|---|---|
| 精密化 | 空間: REAL |
| 得られたモデル |  PDB-8bef: |
 ムービー
ムービー コントローラー
コントローラー




























 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)