+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12918 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Cetacean morbillivirus nucleoprotein-RNA complex | |||||||||
 マップデータ マップデータ | Masked postprocessed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cetacean morbillivirus / nucleocapsid / RNA virus replication / cryo-electron microscopy / helical reconstruction / single particle analysis / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Paramyxovirus nucleocapsid protein / Paramyxovirus nucleocapsid protein / helical viral capsid / viral nucleocapsid / host cell cytoplasm / structural molecule activity / RNA binding / Nucleocapsid 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Morbillivirus sp. (ウイルス) Morbillivirus sp. (ウイルス) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Zinzula L / Beck F | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2021 ジャーナル: J Struct Biol / 年: 2021タイトル: Cryo-EM structure of the cetacean morbillivirus nucleoprotein-RNA complex. 著者: Luca Zinzula / Florian Beck / Sven Klumpe / Stefan Bohn / Günter Pfeifer / Daniel Bollschweiler / István Nagy / Jürgen M Plitzko / Wolfgang Baumeister /  要旨: Cetacean morbillivirus (CeMV) is an emerging and highly infectious paramyxovirus that causes outbreaks in cetaceans and occasionally in pinnipeds, representing a major threat to biodiversity and ...Cetacean morbillivirus (CeMV) is an emerging and highly infectious paramyxovirus that causes outbreaks in cetaceans and occasionally in pinnipeds, representing a major threat to biodiversity and conservation of endangered marine mammal populations in both hemispheres. As for all non-segmented, negative-sense, single-stranded RNA (ssRNA) viruses, the morbilliviral genome is enwrapped by thousands of nucleoprotein (N) protomers. Each bound to six ribonucleotides, N protomers assemble to form a helical ribonucleoprotein (RNP) complex that serves as scaffold for nucleocapsid formation and as template for viral replication and transcription. While the molecular details on RNP complexes elucidated in human measles virus (MeV) served as paradigm model for these processes in all members of the Morbillivirus genus, no structural information has been obtained from other morbilliviruses, nor has any CeMV structure been solved so far. We report the structure of the CeMV RNP complex, reconstituted in vitro upon binding of recombinant CeMV N to poly-adenine ssRNA hexamers and solved to 4.0 Å resolution by cryo-electron microscopy. In spite of the amino acid sequence similarity and consequently similar folding of the N protomer, the CeMV RNP complex exhibits different helical parameters as compared to previously reported MeV orthologs. The CeMV structure reveals exclusive interactions leading to more extensive protomer-RNA and protomer-protomer interfaces. We identified twelve residues, among those varying between CeMV strains, as putatively important for the stabilization of the RNP complex, which highlights the need to study the potential of CeMV N mutations that modulate nucleocapsid assembly to also affect viral phenotype and host adaptation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12918.map.gz emd_12918.map.gz | 9.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12918-v30.xml emd-12918-v30.xml emd-12918.xml emd-12918.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12918_fsc.xml emd_12918_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12918.png emd_12918.png | 162.6 KB | ||
| マスクデータ |  emd_12918_msk_1.map emd_12918_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12918.cif.gz emd-12918.cif.gz | 5.8 KB | ||
| その他 |  emd_12918_additional_1.map.gz emd_12918_additional_1.map.gz emd_12918_additional_2.map.gz emd_12918_additional_2.map.gz emd_12918_half_map_1.map.gz emd_12918_half_map_1.map.gz emd_12918_half_map_2.map.gz emd_12918_half_map_2.map.gz | 56.3 MB 49.5 MB 49.7 MB 49.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12918 http://ftp.pdbj.org/pub/emdb/structures/EMD-12918 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12918 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12918 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12918_validation.pdf.gz emd_12918_validation.pdf.gz | 818.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12918_full_validation.pdf.gz emd_12918_full_validation.pdf.gz | 818 KB | 表示 | |
| XML形式データ |  emd_12918_validation.xml.gz emd_12918_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_12918_validation.cif.gz emd_12918_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12918 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12918 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12918 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12918 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12918.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12918.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked postprocessed map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12918_msk_1.map emd_12918_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
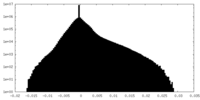
| 密度ヒストグラム |
-追加マップ: DeepEMhancer map
| ファイル | emd_12918_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
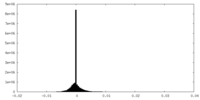
| 密度ヒストグラム |
-追加マップ: Map before postprocessing
| ファイル | emd_12918_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map before postprocessing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_12918_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_12918_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cetacean morbillivirus nucleoprotein-RNA complex
| 全体 | 名称: Cetacean morbillivirus nucleoprotein-RNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Cetacean morbillivirus nucleoprotein-RNA complex
| 超分子 | 名称: Cetacean morbillivirus nucleoprotein-RNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Cetacean morbillivirus nucleoprotein
| 超分子 | 名称: Cetacean morbillivirus nucleoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Morbillivirus sp. (ウイルス) Morbillivirus sp. (ウイルス) |
-超分子 #3: poly-A 6-mer
| 超分子 | 名称: poly-A 6-mer / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Morbillivirus sp. (ウイルス) Morbillivirus sp. (ウイルス) |
-分子 #1: Cetacean morbillivirus nucleoprotein
| 分子 | 名称: Cetacean morbillivirus nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Morbillivirus sp. (ウイルス) Morbillivirus sp. (ウイルス) |
| 分子量 | 理論値: 46.273625 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GMATLLRSLA LFKRNKDRTP LTAGSGGAIR GIKHVIVVPV PGDSSIVTRS RLLDRLVRLA GDPYISGPKL TGVMISILSL FVESPSQLI QRITDDPDVS IRLVEVIQSE KSLSGLTFAS RGANMEDEAD DYFSIQAGEE GDTRGTHWFE NKEIVEIEVQ D PEEFNILL ...文字列: GMATLLRSLA LFKRNKDRTP LTAGSGGAIR GIKHVIVVPV PGDSSIVTRS RLLDRLVRLA GDPYISGPKL TGVMISILSL FVESPSQLI QRITDDPDVS IRLVEVIQSE KSLSGLTFAS RGANMEDEAD DYFSIQAGEE GDTRGTHWFE NKEIVEIEVQ D PEEFNILL ASILAQIWIL LAKAVTAPDT AADSETRRWI KYTQQRRVVG EFRLDKGWLD AVRNRIAEDL SLRRFMVALI LD IKRTPGN KPRIAEMICD IDTYIVEAGL ASFILTIKFG IETMYPALGL HEFSGELTTV ESLMNLYQQM GETAPYMVIL ENS IQNKFS AGSYPLLWSY AMGVGVELEN SMGGLNFGRS YFDPAYFRLG QEMVRRSAGK VSSSLAAELG ITAEDAKLVS EIAA QANDD RVEHHHHHHH H UniProtKB: Nucleocapsid |
-分子 #2: poly-A 6-mer
| 分子 | 名称: poly-A 6-mer / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Morbillivirus sp. (ウイルス) Morbillivirus sp. (ウイルス) |
| 分子量 | 理論値: 1.930277 KDa |
| 配列 | 文字列: AAAAAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3153 / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X