+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11630 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Envelope glycprotein of endogenous retrovirus Y032 (Atlas virus) from the human hookworm Ancylostoma ceylanicum | ||||||||||||
 マップデータ マップデータ | Final EM map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | class II membrane fusion protein / retroviral envelope protein (Env) / lipid binding protein / disulfide bonding / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Ancylostoma ceylanicum (セイロン鉤虫) Ancylostoma ceylanicum (セイロン鉤虫) | ||||||||||||
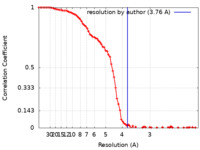
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.76 Å | ||||||||||||
 データ登録者 データ登録者 | Mata CP / Merchant M | ||||||||||||
| 資金援助 |  英国, 英国,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: A bioactive phlebovirus-like envelope protein in a hookworm endogenous virus. 著者: Monique Merchant / Carlos P Mata / Yangci Liu / Haoming Zhai / Anna V Protasio / Yorgo Modis /  要旨: Endogenous viral elements (EVEs), accounting for 15% of our genome, serve as a genetic reservoir from which new genes can emerge. Nematode EVEs are particularly diverse and informative of virus ...Endogenous viral elements (EVEs), accounting for 15% of our genome, serve as a genetic reservoir from which new genes can emerge. Nematode EVEs are particularly diverse and informative of virus evolution. We identify Atlas virus-an intact retrovirus-like EVE in the human hookworm , with an envelope protein genetically related to G-G glycoproteins from the family Phenuiviridae. A cryo-EM structure of Atlas G reveals a class II viral membrane fusion protein fold not previously seen in retroviruses. Atlas G has the structural hallmarks of an active fusogen. Atlas G trimers insert into membranes with endosomal lipid compositions and low pH. When expressed on the plasma membrane, Atlas G has cell-cell fusion activity. With its preserved biological activities, Atlas G has the potential to acquire a cellular function. Our work reveals structural plasticity in reverse-transcribing RNA viruses. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: A bioactive phlebovirus-like envelope protein in a hookworm endogenous virus 著者: Merchant M / Mata CP / Liu Y / Zhai H / Protasio AV / Modis Y | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11630.map.gz emd_11630.map.gz | 39.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11630-v30.xml emd-11630-v30.xml emd-11630.xml emd-11630.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11630_fsc.xml emd_11630_fsc.xml | 9.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11630.png emd_11630.png | 167.6 KB | ||
| マスクデータ |  emd_11630_msk_1.map emd_11630_msk_1.map | 42.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11630.cif.gz emd-11630.cif.gz | 7.4 KB | ||
| その他 |  emd_11630_half_map_1.map.gz emd_11630_half_map_1.map.gz emd_11630_half_map_2.map.gz emd_11630_half_map_2.map.gz | 33 MB 33 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11630 http://ftp.pdbj.org/pub/emdb/structures/EMD-11630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11630 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11630_validation.pdf.gz emd_11630_validation.pdf.gz | 830.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11630_full_validation.pdf.gz emd_11630_full_validation.pdf.gz | 829.9 KB | 表示 | |
| XML形式データ |  emd_11630_validation.xml.gz emd_11630_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_11630_validation.cif.gz emd_11630_validation.cif.gz | 19.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11630 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11630 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11630 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11630 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7a4aMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10266 (タイトル: CryoEM image reconstuction of the envelope protein of endogenous retrovirus Y032 from the human hookworm Ancylostoma ceylanicum EMPIAR-10266 (タイトル: CryoEM image reconstuction of the envelope protein of endogenous retrovirus Y032 from the human hookworm Ancylostoma ceylanicumData size: 5.6 TB Data #1: Unaligned multi-frame micrographs of Env protein endogenous retrovirus AceY032 from Ancylostoma ceylanicum [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11630.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11630.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final EM map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.047 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11630_msk_1.map emd_11630_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_11630_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_11630_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Viral envelope glycoprotein
| 全体 | 名称: Viral envelope glycoprotein |
|---|---|
| 要素 |
|
-超分子 #1: Viral envelope glycoprotein
| 超分子 | 名称: Viral envelope glycoprotein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Envelope glycoprotein of endogenous retrovirus Y032 (Atlas virus) from Ancylostoma ceylanicum |
|---|---|
| 由来(天然) | 生物種:  Ancylostoma ceylanicum (セイロン鉤虫) Ancylostoma ceylanicum (セイロン鉤虫) |
| 分子量 | 理論値: 143 KDa |
-分子 #1: Integrase catalytic domain-containing protein
| 分子 | 名称: Integrase catalytic domain-containing protein / タイプ: protein_or_peptide / ID: 1 詳細: In chains A, B and C, residue Asn414 is covalently modified with an N-linked N-acetyl glucosamine ligand. Chains A, B and C each contain the following 15 disulfide bonds: Cys1-Cys41, Cys14- ...詳細: In chains A, B and C, residue Asn414 is covalently modified with an N-linked N-acetyl glucosamine ligand. Chains A, B and C each contain the following 15 disulfide bonds: Cys1-Cys41, Cys14-Cys23, Cys66-Cys162, Cys87-Cys135, Cys93-Cys142, Cys98-Cys123, Cys127-Cys132, Cys129-Cys138, Cys246-Cys257, Cys264-Cys277, Cys266-Cys275, Cys337-Cys408, Cys347-Cys350, Cys360-Cys382, Cys373-Cys404. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Ancylostoma ceylanicum (セイロン鉤虫) Ancylostoma ceylanicum (セイロン鉤虫) |
| 分子量 | 理論値: 51.51909 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: CSEVISVTST EEVCTIQENK ETCTFNHATT ITLQPLQQQT CLTLNDPEKR PMGMLTVKPD GIKFRCNKKI EFFTRDHQIV SESVHRCHR AGSCHSDECH HVKDTDALPE FSSEANSRPG YTSCSSSCGC ITCDGCFFCE PSCLFHRLYA IPTTPTIYSI F YCPSWELE ...文字列: CSEVISVTST EEVCTIQENK ETCTFNHATT ITLQPLQQQT CLTLNDPEKR PMGMLTVKPD GIKFRCNKKI EFFTRDHQIV SESVHRCHR AGSCHSDECH HVKDTDALPE FSSEANSRPG YTSCSSSCGC ITCDGCFFCE PSCLFHRLYA IPTTPTIYSI F YCPSWELE VDAEISLQRE DETTTSTIRL LPGRTSTWNN IRFSLIGTIV PQLPILSSAF VTNGRQTSIV KPAYAGQLQS NS VGQLQCP NLEAAKQFEC HFSRNLCTCT NALHKVSCTC YDGSVEDHME ALPLPQTSKN FLVFEKDRNI YAKTHVGSAL QLH IVAQDL KITTVKHTSH CQVEASDLSG CYSCTSGASL TLSCKSDNGE VLANMKCNEQ THVIRCTESG FINNILLMFD TSEV AADCT AACPGGIVNF TIKGLLAFVN ERIISQSYSA TDVERNIKGK PIPNPLLGLD STRTGHHHHH H UniProtKB: Integrase catalytic domain-containing protein |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.025 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: 20 mM Tris/HCl (NH12C4O3Cl) 0.1 M NaCl 'sodium chloride' 5 % glycerol (C3H8O3) 0.5 mM TCEP (C9H15O6P) | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.2 K / 装置: FEI VITROBOT MARK IV / 詳細: Grids were blotted for 4 s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: OTHER / 球面収差補正装置: None / 色収差補正装置: None / エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 1 / 実像数: 3027 / 平均露光時間: 8.0 sec. / 平均電子線量: 46.18 e/Å2 / 詳細: Dose rate = 1.28 e- A^-2 per frame |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 75000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.3 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 691-1136 / Chain - Source name: PDB / Chain - Initial model type: experimental model 詳細: A homology model was built from PDB:6EGU using the Swiss-Model server (swissmodel.expasy.org). The model was docked as a rigid body into the density with UCSF Chimera prior to refinement. |
|---|---|
| 詳細 | A homology model was built from PDB:6EGU using the Swiss-Model server (swissmodel.expasy.org). The model was docked as a rigid body into the density with UCSF Chimera prior to refinement. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 82 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-7a4a: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)