+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10789 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of ExbB pentamer from Serratia marcescens by single particle cryo electron microscopy | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein / iron uptake / proton transfer / TonB complex / METAL TRANSPORT | |||||||||
| 機能・相同性 | TonB-system energizer ExbB type-1 / : / MotA/TolQ/ExbB proton channel / MotA/TolQ/ExbB proton channel family / protein import / transmembrane transporter activity / plasma membrane / Biopolymer transport protein ExbB 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Serratia marcescens (霊菌) Serratia marcescens (霊菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Biou V / Delepelaire P | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 | ジャーナル: J Struct Biol / 年: 2012 タイトル: RELION: implementation of a Bayesian approach to cryo-EM structure determination. 著者: Sjors H W Scheres /  要旨: RELION, for REgularized LIkelihood OptimizatioN, is an open-source computer program for the refinement of macromolecular structures by single-particle analysis of electron cryo-microscopy (cryo-EM) ...RELION, for REgularized LIkelihood OptimizatioN, is an open-source computer program for the refinement of macromolecular structures by single-particle analysis of electron cryo-microscopy (cryo-EM) data. Whereas alternative approaches often rely on user expertise for the tuning of parameters, RELION uses a Bayesian approach to infer parameters of a statistical model from the data. This paper describes developments that reduce the computational costs of the underlying maximum a posteriori (MAP) algorithm, as well as statistical considerations that yield new insights into the accuracy with which the relative orientations of individual particles may be determined. A so-called gold-standard Fourier shell correlation (FSC) procedure to prevent overfitting is also described. The resulting implementation yields high-quality reconstructions and reliable resolution estimates with minimal user intervention and at acceptable computational costs. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: Functional and structural characterization of Serratia marcescens ExbB: determinants of the interaction with HasB/TonB 著者: Biou V / Chami M / Coureux PD / Laurent B / Ntsogo Y / Izadi-Pruneyre N / Malosse C / Chamot-Rooke J / Stahlberg H / Delepelaire P | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10789.map.gz emd_10789.map.gz | 13.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10789-v30.xml emd-10789-v30.xml emd-10789.xml emd-10789.xml | 25.1 KB 25.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10789_fsc.xml emd_10789_fsc.xml | 12.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10789.png emd_10789.png | 132.3 KB | ||
| マスクデータ |  emd_10789_msk_1.map emd_10789_msk_1.map | 184 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10789.cif.gz emd-10789.cif.gz | 7.2 KB | ||
| その他 |  emd_10789_additional_1.map.gz emd_10789_additional_1.map.gz emd_10789_half_map_1.map.gz emd_10789_half_map_1.map.gz emd_10789_half_map_2.map.gz emd_10789_half_map_2.map.gz | 3.6 MB 145.7 MB 145.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10789 http://ftp.pdbj.org/pub/emdb/structures/EMD-10789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10789 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10789_validation.pdf.gz emd_10789_validation.pdf.gz | 737.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10789_full_validation.pdf.gz emd_10789_full_validation.pdf.gz | 737.5 KB | 表示 | |
| XML形式データ |  emd_10789_validation.xml.gz emd_10789_validation.xml.gz | 19.9 KB | 表示 | |
| CIF形式データ |  emd_10789_validation.cif.gz emd_10789_validation.cif.gz | 26 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10789 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10789 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10789 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10789 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10789.map.gz / 形式: CCP4 / 大きさ: 184 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10789.map.gz / 形式: CCP4 / 大きさ: 184 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.827 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
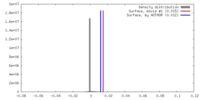
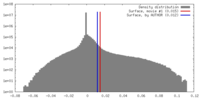
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10789_msk_1.map emd_10789_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_10789_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10789_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10789_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homopentamer of ExbB
| 全体 | 名称: Homopentamer of ExbB |
|---|---|
| 要素 |
|
-超分子 #1: Homopentamer of ExbB
| 超分子 | 名称: Homopentamer of ExbB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: the expressed sequence corresponds to the mature sequence after signal peptide cleavage. |
|---|---|
| 由来(天然) | 生物種:  Serratia marcescens (霊菌) / 株: Db11 Serratia marcescens (霊菌) / 株: Db11 |
| 分子量 | 理論値: 174 KDa |
-分子 #1: Biopolymer transport protein ExbB
| 分子 | 名称: Biopolymer transport protein ExbB / タイプ: protein_or_peptide / ID: 1 / 詳細: a 6-histidine tag is present at the C-terminus / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Serratia marcescens (霊菌) Serratia marcescens (霊菌) |
| 分子量 | 理論値: 30.413621 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: APAANPAVTE SVAPTTAPAP AAAAPESITP VNPAPTIQPP ETRGMDLSIW GMYQHADAVV KAVMIGLVLA SIVTWTILFA KGSELLRAK RRLRREQLAL AEARSLDEAS ELAQNFSPES VSAVLLNDAQ NELELSAESN DNNGIKERTG FRLERRVAAY S RNMGRGNG ...文字列: APAANPAVTE SVAPTTAPAP AAAAPESITP VNPAPTIQPP ETRGMDLSIW GMYQHADAVV KAVMIGLVLA SIVTWTILFA KGSELLRAK RRLRREQLAL AEARSLDEAS ELAQNFSPES VSAVLLNDAQ NELELSAESN DNNGIKERTG FRLERRVAAY S RNMGRGNG FLATIGAISP FVGLFGTVWG IMNSFIGIAH SQTTNLAVVA PGIAEALLAT AMGLVAAIPA VVIYNIFARV IS GHRAQVG DVAAQVLLLQ GRDLDLAATA EAKRSQHAHQ LRAGHHHHHH UniProtKB: Biopolymer transport protein ExbB |
-分子 #2: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
| 分子 | 名称: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-1-[(PALMITOYLOXY)METHYL]ETHYL STEARATE タイプ: ligand / ID: 2 / コピー数: 5 / 式: PGT |
|---|---|
| 分子量 | 理論値: 751.023 Da |
| Chemical component information |  ChemComp-PGT: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: 20mM Tris-HCl pH 8,0 100mM NaCl 0,0015% LMNG | ||||||||||||
| グリッド | モデル: Quantifoil R2/4 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM CPC | ||||||||||||
| 詳細 | the sample was monodisperse as evidenced by gel filtration column |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 83.0 K / 最高: 93.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 1-56 / 撮影したグリッド数: 1 / 実像数: 3122 / 平均露光時間: 7.0 sec. / 平均電子線量: 55.95 e/Å2 詳細: frames were weighted according to electron dose and particle movement during Relion bayesian polishing procedure. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 10-234 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | the ExbB sequence from S. marcescens was modeled by homology from the 5SV0 monomer from E. coli using Phyre software and the pentamer was generated using the 5SV0 symmetry. real space refinement was carried out with rigid body, simulated annealing and morphing steps. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 55.3 / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-6ye4: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)