+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10528 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the chaperonin gp146 from the bacteriophage EL (Pseudomonas aeruginosa) in the apo state | |||||||||
 マップデータ マップデータ | CryoEM map of apo gp146, the chaperonin from bacteriophage EL (Pseudomonas putida) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | molecular chaperone / ATPase / chaperonin / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATP-dependent protein folding chaperone / protein refolding / ATP binding / metal ion binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas phage EL (ファージ) Pseudomonas phage EL (ファージ) | |||||||||
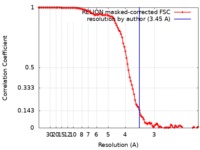
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.45 Å | |||||||||
 データ登録者 データ登録者 | Bracher A / Wang H | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2020 ジャーナル: PLoS One / 年: 2020タイトル: Structure and conformational cycle of a bacteriophage-encoded chaperonin. 著者: Andreas Bracher / Simanta S Paul / Huping Wang / Nadine Wischnewski / F Ulrich Hartl / Manajit Hayer-Hartl /  要旨: Chaperonins are ubiquitous molecular chaperones found in all domains of life. They form ring-shaped complexes that assist in the folding of substrate proteins in an ATP-dependent reaction cycle. Key ...Chaperonins are ubiquitous molecular chaperones found in all domains of life. They form ring-shaped complexes that assist in the folding of substrate proteins in an ATP-dependent reaction cycle. Key to the folding cycle is the transient encapsulation of substrate proteins by the chaperonin. Here we present a structural and functional characterization of the chaperonin gp146 (ɸEL) from the phage EL of Pseudomonas aeruginosa. ɸEL, an evolutionarily distant homolog of bacterial GroEL, is active in ATP hydrolysis and prevents the aggregation of denatured protein in a nucleotide-dependent manner. However, ɸEL failed to refold the encapsulation-dependent model substrate rhodanese and did not interact with E. coli GroES, the lid-shaped co-chaperone of GroEL. ɸEL forms tetradecameric double-ring complexes, which dissociate into single rings in the presence of ATP. Crystal structures of ɸEL (at 3.54 and 4.03 Å) in presence of ATP•BeFx revealed two distinct single-ring conformational states, both with open access to the ring cavity. One state showed uniform ATP-bound subunit conformations (symmetric state), whereas the second combined distinct ATP- and ADP-bound subunit conformations (asymmetric state). Cryo-electron microscopy of apo-ɸEL revealed a double-ring structure composed of rings in the asymmetric state (3.45 Å resolution). We propose that the phage chaperonin undergoes nucleotide-dependent conformational switching between double- and single rings and functions in aggregation prevention without substrate protein encapsulation. Thus, ɸEL may represent an evolutionarily more ancient chaperonin prior to acquisition of the encapsulation mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10528.map.gz emd_10528.map.gz | 12.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10528-v30.xml emd-10528-v30.xml emd-10528.xml emd-10528.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10528_fsc.xml emd_10528_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10528.png emd_10528.png | 91.6 KB | ||
| Filedesc metadata |  emd-10528.cif.gz emd-10528.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10528 http://ftp.pdbj.org/pub/emdb/structures/EMD-10528 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10528 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10528 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10528_validation.pdf.gz emd_10528_validation.pdf.gz | 446.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10528_full_validation.pdf.gz emd_10528_full_validation.pdf.gz | 446 KB | 表示 | |
| XML形式データ |  emd_10528_validation.xml.gz emd_10528_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_10528_validation.cif.gz emd_10528_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10528 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10528 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10528 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10528 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10528.map.gz / 形式: CCP4 / 大きさ: 96.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10528.map.gz / 形式: CCP4 / 大きさ: 96.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of apo gp146, the chaperonin from bacteriophage EL (Pseudomonas putida) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : The chaperonin gp146 from the bacteriophage EL (Pseudomonas aerug...
| 全体 | 名称: The chaperonin gp146 from the bacteriophage EL (Pseudomonas aeruginosa) in the apo state |
|---|---|
| 要素 |
|
-超分子 #1: The chaperonin gp146 from the bacteriophage EL (Pseudomonas aerug...
| 超分子 | 名称: The chaperonin gp146 from the bacteriophage EL (Pseudomonas aeruginosa) in the apo state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage EL (ファージ) Pseudomonas phage EL (ファージ) |
| 分子量 | 理論値: 864 KDa |
-分子 #1: Putative GroEL-like chaperonine protein
| 分子 | 名称: Putative GroEL-like chaperonine protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage EL (ファージ) Pseudomonas phage EL (ファージ) |
| 分子量 | 理論値: 61.760941 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSQTLLVHGK DAQGIIKQVL SEVYDAVTST MGPNGQLVMI KNGVSTKTTK DGVTVARSIR FADEAHELVN RVITEPATKT DEECGDGTT TTIMLTHALY HLFKDFPGFQ HHRNIEDLVE RVIQRLESMA IRVEVDDPRL YQVALTSSNQ DEKLARLVSE L YANNKGSY ...文字列: MSQTLLVHGK DAQGIIKQVL SEVYDAVTST MGPNGQLVMI KNGVSTKTTK DGVTVARSIR FADEAHELVN RVITEPATKT DEECGDGTT TTIMLTHALY HLFKDFPGFQ HHRNIEDLVE RVIQRLESMA IRVEVDDPRL YQVALTSSNQ DEKLARLVSE L YANNKGSY PDIELKEGVN FEDQIEQTTG RTIRMFYANP WFAKGHQGGV TELTGFTAFV IDRRIDKEDT QKLIDGVNHL VK THKQHLA LPILLIARSF EEAANSTLMQ LNAAHPTLVE DGRPWLIPLS TPVGGAIGTS ELQDIAVMLN APMLSDVADL TKL DTHSIN GQHGQLELGG NRSILKSTTP KDEDRIEQHA RGIEELLEGF SLSDKFSVRA RYNERRIRTL RGKLITISVG GETY SEVKE RVDRYEDVVK AIRSALENGI LPGGGVSLVK AVFGTIKEGL EDKDQSAEFA KRYINSGIAN ELMRLSTIQH KLLFK DTAL YKENGSFHFN DDWLNTPTVM NLATGEIGTP EGLGIYDTAY ASITALKGGL QTAKILATTK TLILGEKLSA VKVR UniProtKB: Putative GroEL-like chaperonine protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.125 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot time was 2 sec.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 撮影したグリッド数: 1 / #0 - 実像数: 2519 / #0 - 平均露光時間: 0.11984 sec. / #0 - 平均電子線量: 1.552 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 撮影したグリッド数: 1 / #1 - 実像数: 3063 / #1 - 平均露光時間: 0.12 sec. / #1 - 平均電子線量: 1.55 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Jelly body refinement C2 symmetry NCS restraints |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-6tmv: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)