+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10504 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Multiple Genomic RNA-Coat Protein Contacts Play Vital Roles in the Assembly of Infectious Enterovirus-E | ||||||||||||||||||||||||
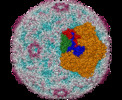
 マップデータ マップデータ | BEV1 I2 cryoEM map at 2.23A resolution | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | BEV1 / enterovirus / picornavirus / RNA / VIRUS | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / nucleoside-triphosphate phosphatase ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / symbiont-mediated suppression of host gene expression / DNA replication / RNA helicase activity / induction by virus of host autophagy / RNA-directed RNA polymerase / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / virus-mediated perturbation of host defense response / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity / proteolysis / RNA binding / ATP binding / membrane / metal ion binding 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Bovine enterovirus (strain VG-5-27) (エンテロウイルス) Bovine enterovirus (strain VG-5-27) (エンテロウイルス) | ||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.23 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Chandler-Bostock R / Mata CP / Bingham R / Dykeman EC / Meng B / Tuthill TJ / Rowlands DJ / Ranson NA / Twarock R / Stockley PG | ||||||||||||||||||||||||
| 資金援助 |  英国, 7件 英国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2020 ジャーナル: PLoS Pathog / 年: 2020タイトル: Assembly of infectious enteroviruses depends on multiple, conserved genomic RNA-coat protein contacts. 著者: Rebecca Chandler-Bostock / Carlos P Mata / Richard J Bingham / Eric C Dykeman / Bo Meng / Tobias J Tuthill / David J Rowlands / Neil A Ranson / Reidun Twarock / Peter G Stockley /  要旨: Picornaviruses are important viral pathogens, but despite extensive study, the assembly process of their infectious virions is still incompletely understood, preventing the development of anti-viral ...Picornaviruses are important viral pathogens, but despite extensive study, the assembly process of their infectious virions is still incompletely understood, preventing the development of anti-viral strategies targeting this essential part of the life cycle. We report the identification, via RNA SELEX and bioinformatics, of multiple RNA sites across the genome of a typical enterovirus, enterovirus-E (EV-E), that each have affinity for the cognate viral capsid protein (CP) capsomer. Many of these sites are evolutionarily conserved across known EV-E variants, suggesting they play essential functional roles. Cryo-electron microscopy was used to reconstruct the EV-E particle at ~2.2 Å resolution, revealing extensive density for the genomic RNA. Relaxing the imposed symmetry within the reconstructed particles reveals multiple RNA-CP contacts, a first for any picornavirus. Conservative mutagenesis of the individual RNA-contacting amino acid side chains in EV-E, many of which are conserved across the enterovirus family including poliovirus, is lethal but does not interfere with replication or translation. Anti-EV-E and anti-poliovirus aptamers share sequence similarities with sites distributed across the poliovirus genome. These data are consistent with the hypothesis that these RNA-CP contacts are RNA Packaging Signals (PSs) that play vital roles in assembly and suggest that the RNA PSs are evolutionarily conserved between pathogens within the family, augmenting the current protein-only assembly paradigm for this family of viruses. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10504.map.gz emd_10504.map.gz | 363.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10504-v30.xml emd-10504-v30.xml emd-10504.xml emd-10504.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10504.png emd_10504.png | 404.9 KB | ||
| Filedesc metadata |  emd-10504.cif.gz emd-10504.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10504 http://ftp.pdbj.org/pub/emdb/structures/EMD-10504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10504 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10504_validation.pdf.gz emd_10504_validation.pdf.gz | 706.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10504_full_validation.pdf.gz emd_10504_full_validation.pdf.gz | 706.1 KB | 表示 | |
| XML形式データ |  emd_10504_validation.xml.gz emd_10504_validation.xml.gz | 7.7 KB | 表示 | |
| CIF形式データ |  emd_10504_validation.cif.gz emd_10504_validation.cif.gz | 8.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10504 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10504 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6thdMC  6thnC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10341 (タイトル: Multiple Genomic RNA-Coat Protein Contacts Play Vital Roles in the Assembly of Infectious Enterovirus-E EMPIAR-10341 (タイトル: Multiple Genomic RNA-Coat Protein Contacts Play Vital Roles in the Assembly of Infectious Enterovirus-EData size: 635.4 Data #1: Motion corrected micrographs of BEV1 [micrographs - single frame] Data #2: Polished particles of BEV1 [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10504.map.gz / 形式: CCP4 / 大きさ: 391 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10504.map.gz / 形式: CCP4 / 大きさ: 391 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BEV1 I2 cryoEM map at 2.23A resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bovine enterovirus (strain VG-5-27)
| 全体 | 名称:  Bovine enterovirus (strain VG-5-27) (エンテロウイルス) Bovine enterovirus (strain VG-5-27) (エンテロウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Bovine enterovirus (strain VG-5-27)
| 超分子 | 名称: Bovine enterovirus (strain VG-5-27) / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 12065 / 生物種: Bovine enterovirus (strain VG-5-27) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| ウイルス殻 | Shell ID: 1 / T番号(三角分割数): 3 |
-分子 #1: Genome polyprotein
| 分子 | 名称: Genome polyprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: picornain 2A |
|---|---|
| 由来(天然) | 生物種:  Bovine enterovirus (strain VG-5-27) (エンテロウイルス) Bovine enterovirus (strain VG-5-27) (エンテロウイルス) |
| 分子量 | 理論値: 31.280955 KDa |
| 配列 | 文字列: NDPGKMLKDA IDKQVAGALV AGTTTSTHSV ATDSTPALQA AETGATSTAR DESMIETRTI VPTHGIHETS VESFFGRSSL VGMPLLATG TSITHWRIDF REFVQLRAKM SWFTYMRFDV EFTIIATSST GQNVTTEQHT TYQVMYVPPG APVPSNQDSF Q WQSGCNPS ...文字列: NDPGKMLKDA IDKQVAGALV AGTTTSTHSV ATDSTPALQA AETGATSTAR DESMIETRTI VPTHGIHETS VESFFGRSSL VGMPLLATG TSITHWRIDF REFVQLRAKM SWFTYMRFDV EFTIIATSST GQNVTTEQHT TYQVMYVPPG APVPSNQDSF Q WQSGCNPS VFADTDGPPA QFSVPFMSSA NAYSTVYDGY ARFMDTDPDR YGILPSNFLG FMYFRTLEDA AHQVRFRIYA KI KHTSCWI PRAPRQAPYK KRYNLVFSGD SDRICSNRAS LTSY UniProtKB: Genome polyprotein |
-分子 #2: Genome polyprotein
| 分子 | 名称: Genome polyprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: picornain 2A |
|---|---|
| 由来(天然) | 生物種:  Bovine enterovirus (strain VG-5-27) (エンテロウイルス) Bovine enterovirus (strain VG-5-27) (エンテロウイルス) |
| 分子量 | 理論値: 27.325604 KDa |
| 配列 | 文字列: SPSAEACGYS DRVAQLTLGN STITTQEAAN ICVAYGCWPA KLSDTDATSV DKPTEPGVSA DRFYTLRSKP WQADSKGWYW KLPDALNNT GMFGQNAQFH YIYRGGWAVH VQCNATKFHQ GTLLVLAIPE HQIATQEQPA FDRTMPGSEG GTFQEPFWLE D GTSLGNSL ...文字列: SPSAEACGYS DRVAQLTLGN STITTQEAAN ICVAYGCWPA KLSDTDATSV DKPTEPGVSA DRFYTLRSKP WQADSKGWYW KLPDALNNT GMFGQNAQFH YIYRGGWAVH VQCNATKFHQ GTLLVLAIPE HQIATQEQPA FDRTMPGSEG GTFQEPFWLE D GTSLGNSL IYPHQWINLR TNNSATLILP YVNAIPMDSA IRHSNWTLAI IPVAPLKYAA ETTPLVPITV TIAPMETEYN GL RRAIASN Q UniProtKB: Genome polyprotein |
-分子 #3: Genome polyprotein
| 分子 | 名称: Genome polyprotein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: picornain 2A |
|---|---|
| 由来(天然) | 生物種:  Bovine enterovirus (strain VG-5-27) (エンテロウイルス) Bovine enterovirus (strain VG-5-27) (エンテロウイルス) |
| 分子量 | 理論値: 26.627221 KDa |
| 配列 | 文字列: GLPTKPGPGS YQFMTTDEDC SPCILPDFQP TPEIFIPGKV NNLLEIAQVE SILEANNREG VEGVERYVIP VSVQDALDAQ IYALRLELG GSGPLSSSLL GTLAKHYTQW SGSVEITCMF TGTFMTTGKV LLAYTPPGGD MPRNREEAML GTHVIWDFGL Q SSITLVIP ...文字列: GLPTKPGPGS YQFMTTDEDC SPCILPDFQP TPEIFIPGKV NNLLEIAQVE SILEANNREG VEGVERYVIP VSVQDALDAQ IYALRLELG GSGPLSSSLL GTLAKHYTQW SGSVEITCMF TGTFMTTGKV LLAYTPPGGD MPRNREEAML GTHVIWDFGL Q SSITLVIP WISASHFRGV SNDDVLNYQY YAAGHVTIWY QTNMVIPPGF PNTAGIIMMI AAQPNFSFRI QKDREDMTQT AI LQ UniProtKB: Genome polyprotein |
-分子 #4: Genome polyprotein
| 分子 | 名称: Genome polyprotein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: picornain 2A |
|---|---|
| 由来(天然) | 生物種:  Bovine enterovirus (strain VG-5-27) (エンテロウイルス) Bovine enterovirus (strain VG-5-27) (エンテロウイルス) |
| 分子量 | 理論値: 5.619105 KDa |
| 配列 | 文字列: GAQGGSTINY NNINYYSHAA SAAQNKQDFT QDPSKFTQPI ADVIKETAVP LK UniProtKB: Genome polyprotein |
-分子 #5: MYRISTIC ACID
| 分子 | 名称: MYRISTIC ACID / タイプ: ligand / ID: 5 / コピー数: 2 / 式: MYR |
|---|---|
| 分子量 | 理論値: 228.371 Da |
| Chemical component information |  ChemComp-MYR: |
-分子 #6: SULFATE ION
| 分子 | 名称: SULFATE ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: SO4 |
|---|---|
| 分子量 | 理論値: 96.063 Da |
| Chemical component information |  ChemComp-SO4: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 97 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素 - 式: PBS / 詳細: PBS |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 8785 / 平均露光時間: 1.0 sec. / 平均電子線量: 49.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー