+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7615 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
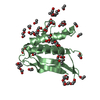
| タイトル | Rabbit muscle aldolase at 4.6 A resolution (17nov02c 1st700 img, 75k particles) | |||||||||
 マップデータ マップデータ | rabbit muscle aldolase at 4.6 A resolution (17nov02c 1st700 img, 75K particles) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
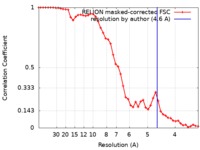
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Kim LK / Rice WJ / Eng ET / Kopylov M / Cheng A / Raczkowski AM / Jordan KD / Bobe D / Potter CS / Carragher B | |||||||||
 引用 引用 |  ジャーナル: Front Mol Biosci / 年: 2018 ジャーナル: Front Mol Biosci / 年: 2018タイトル: Benchmarking cryo-EM Single Particle Analysis Workflow. 著者: Laura Y Kim / William J Rice / Edward T Eng / Mykhailo Kopylov / Anchi Cheng / Ashleigh M Raczkowski / Kelsey D Jordan / Daija Bobe / Clinton S Potter / Bridget Carragher /  要旨: Cryo electron microscopy facilities running multiple instruments and serving users with varying skill levels need a robust and reliable method for benchmarking both the hardware and software ...Cryo electron microscopy facilities running multiple instruments and serving users with varying skill levels need a robust and reliable method for benchmarking both the hardware and software components of their single particle analysis workflow. The workflow is complex, with many bottlenecks existing at the specimen preparation, data collection and image analysis steps; the samples and grid preparation can be of unpredictable quality, there are many different protocols for microscope and camera settings, and there is a myriad of software programs for analysis that can depend on dozens of settings chosen by the user. For this reason, we believe it is important to benchmark the entire workflow, using a standard sample and standard operating procedures, on a regular basis. This provides confidence that all aspects of the pipeline are capable of producing maps to high resolution. Here we describe benchmarking procedures using a test sample, rabbit muscle aldolase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7615.map.gz emd_7615.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7615-v30.xml emd-7615-v30.xml emd-7615.xml emd-7615.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7615_fsc.xml emd_7615_fsc.xml | 4.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7615.png emd_7615.png | 43.2 KB | ||
| その他 |  emd_7615_half_map_1.map.gz emd_7615_half_map_1.map.gz emd_7615_half_map_2.map.gz emd_7615_half_map_2.map.gz | 48.5 MB 48.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7615 http://ftp.pdbj.org/pub/emdb/structures/EMD-7615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7615 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7615_validation.pdf.gz emd_7615_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7615_full_validation.pdf.gz emd_7615_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_7615_validation.xml.gz emd_7615_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7615 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7615 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7528C  7541C  7550C  7551C  7562C  7614C  7616C  7617C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10183 (タイトル: Benchmarking cryo-EM single particle analysis workflow EMPIAR-10183 (タイトル: Benchmarking cryo-EM single particle analysis workflowData size: 345.0 Data #1: aligned micrographs of rabbit muscle aldolase 17nov02c [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7615.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7615.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | rabbit muscle aldolase at 4.6 A resolution (17nov02c 1st700 img, 75K particles) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
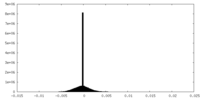
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: rabbit muscle aldolase at 4.6 A resolution (17nov02c...
| ファイル | emd_7615_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | rabbit muscle aldolase at 4.6 A resolution (17nov02c 1st700 img, 75K particles); half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
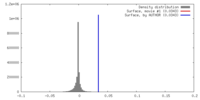
| 密度ヒストグラム |
-ハーフマップ: rabbit muscle aldolase at 4.6 A resolution (17nov02c...
| ファイル | emd_7615_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | rabbit muscle aldolase at 4.6 A resolution (17nov02c 1st700 img, 75K particles); half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
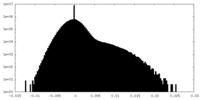
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rabbit muscle aldolase at 4.6A resolution (17nov02c 1st 700 img, ...
| 全体 | 名称: Rabbit muscle aldolase at 4.6A resolution (17nov02c 1st 700 img, 75k particles) |
|---|---|
| 要素 |
|
-超分子 #1: Rabbit muscle aldolase at 4.6A resolution (17nov02c 1st 700 img, ...
| 超分子 | 名称: Rabbit muscle aldolase at 4.6A resolution (17nov02c 1st 700 img, 75k particles) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 150 KDa |
-分子 #1: rabbit muscle aldolase
| 分子 | 名称: rabbit muscle aldolase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO / EC番号: ec: 4.1.12.13 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PHSHPALTPE QKKELSDIAH RIVAPGKGIL AADESTGSIA KRLQSIGTEN TEENRRFYRQ LLLTADDRVN PCIGGVILFH ETLYQKADD GRPFPQVIKS KGGVVGIKVD KGVVPLAGTN GETTTQGLDG LSERCAQYKK DGADFAAWRC VLKIGEHTPS A LAIMENAN ...文字列: PHSHPALTPE QKKELSDIAH RIVAPGKGIL AADESTGSIA KRLQSIGTEN TEENRRFYRQ LLLTADDRVN PCIGGVILFH ETLYQKADD GRPFPQVIKS KGGVVGIKVD KGVVPLAGTN GETTTQGLDG LSERCAQYKK DGADFAAWRC VLKIGEHTPS A LAIMENAN VLARYASICQ QNGIVPIVEP EILPDGDHDL KRCQYVTEKV LAAVYKALSD HHIYLEGTLL KPNMVTPGHA CT QKYSHEE IAMATVTALR RTVPPAVTGV TFLSGGQSEE EASINLNAIN KCPLLKPWAL TFSYGRALQA SALKAWGGKK ENL KAAQEE YVKRALANSL ACQGKYTPSG QAGAAASESL FISNHAY |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)