+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5891 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of MAVSdeltaProTM filament | |||||||||
 マップデータ マップデータ | Cryo-EM map of filaments formed by a truncated MAVS lacking part of the proline-rich region and the C-terminal transmembrane domain | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Innate immunity / helical filament / cryoEM / helical reconstruction | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of IP-10 production / regulation of peroxisome organization / positive regulation of chemokine (C-C motif) ligand 5 production / positive regulation of myeloid dendritic cell cytokine production / CARD domain binding / NF-kB activation through FADD/RIP-1 pathway mediated by caspase-8 and -10 / positive regulation of response to cytokine stimulus / protein localization to mitochondrion / positive regulation of type I interferon-mediated signaling pathway / peroxisomal membrane ...positive regulation of IP-10 production / regulation of peroxisome organization / positive regulation of chemokine (C-C motif) ligand 5 production / positive regulation of myeloid dendritic cell cytokine production / CARD domain binding / NF-kB activation through FADD/RIP-1 pathway mediated by caspase-8 and -10 / positive regulation of response to cytokine stimulus / protein localization to mitochondrion / positive regulation of type I interferon-mediated signaling pathway / peroxisomal membrane / TRAF6 mediated IRF7 activation / negative regulation of viral genome replication / cytoplasmic pattern recognition receptor signaling pathway / type I interferon-mediated signaling pathway / cellular response to exogenous dsRNA / positive regulation of NLRP3 inflammasome complex assembly / TRAF6 mediated NF-kB activation / positive regulation of interferon-alpha production / positive regulation of type I interferon production / ubiquitin ligase complex / positive regulation of defense response to virus by host / signaling adaptor activity / antiviral innate immune response / activation of innate immune response / protein serine/threonine kinase binding / positive regulation of interferon-beta production / positive regulation of interleukin-8 production / Negative regulators of DDX58/IFIH1 signaling / molecular condensate scaffold activity / mitochondrial membrane / DDX58/IFIH1-mediated induction of interferon-alpha/beta / PKR-mediated signaling / Evasion by RSV of host interferon responses / positive regulation of interleukin-6 production / positive regulation of protein import into nucleus / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of tumor necrosis factor production / Ovarian tumor domain proteases / TRAF3-dependent IRF activation pathway / protein-macromolecule adaptor activity / defense response to virus / molecular adaptor activity / DNA-binding transcription factor binding / mitochondrial outer membrane / positive regulation of canonical NF-kappaB signal transduction / intracellular signal transduction / defense response to bacterium / innate immune response / protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / signal transduction / positive regulation of transcription by RNA polymerase II / mitochondrion / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 16.4 Å | |||||||||
 データ登録者 データ登録者 | Xu H / He X / Zheng H / Huang LJ / Hou F / Yu Z / de la Cruz MJ / Borkowski B / Zhang X / Chen ZJ / Jiang Q-X | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2014 ジャーナル: Elife / 年: 2014タイトル: Structural basis for the prion-like MAVS filaments in antiviral innate immunity. 著者: Hui Xu / Xiaojing He / Hui Zheng / Lily J Huang / Fajian Hou / Zhiheng Yu / Michael Jason de la Cruz / Brian Borkowski / Xuewu Zhang / Zhijian J Chen / Qiu-Xing Jiang /  要旨: Mitochondrial antiviral signaling (MAVS) protein is required for innate immune responses against RNA viruses. In virus-infected cells MAVS forms prion-like aggregates to activate antiviral signaling ...Mitochondrial antiviral signaling (MAVS) protein is required for innate immune responses against RNA viruses. In virus-infected cells MAVS forms prion-like aggregates to activate antiviral signaling cascades, but the underlying structural mechanism is unknown. Here we report cryo-electron microscopic structures of the helical filaments formed by both the N-terminal caspase activation and recruitment domain (CARD) of MAVS and a truncated MAVS lacking part of the proline-rich region and the C-terminal transmembrane domain. Both structures are left-handed three-stranded helical filaments, revealing specific interfaces between individual CARD subunits that are dictated by electrostatic interactions between neighboring strands and hydrophobic interactions within each strand. Point mutations at multiple locations of these two interfaces impaired filament formation and antiviral signaling. Super-resolution imaging of virus-infected cells revealed rod-shaped MAVS clusters on mitochondria. These results elucidate the structural mechanism of MAVS polymerization, and explain how an α-helical domain uses distinct chemical interactions to form self-perpetuating filaments. DOI: http://dx.doi.org/10.7554/eLife.01489.001. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5891.map.gz emd_5891.map.gz | 1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5891-v30.xml emd-5891-v30.xml emd-5891.xml emd-5891.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5891.tif emd_5891.tif | 144.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5891 http://ftp.pdbj.org/pub/emdb/structures/EMD-5891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5891 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5891_validation.pdf.gz emd_5891_validation.pdf.gz | 79.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5891_full_validation.pdf.gz emd_5891_full_validation.pdf.gz | 78.9 KB | 表示 | |
| XML形式データ |  emd_5891_validation.xml.gz emd_5891_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5891 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5891 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5891 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5891 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5890C  3j6cC  4o9fC  4o9lC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10014 (タイトル: MAVS CARD and DeltaProTM filaments / Data size: 35.3 / Data #1: CARD set1 micrographs [micrographs - single frame] / Data #2: CARD set2 micrographs [micrographs - single frame] / Data #3: CARD set3 micrographs [micrographs - single frame] / Data #4: CARD set4 micrographs [micrographs - single frame] EMPIAR-10014 (タイトル: MAVS CARD and DeltaProTM filaments / Data size: 35.3 / Data #1: CARD set1 micrographs [micrographs - single frame] / Data #2: CARD set2 micrographs [micrographs - single frame] / Data #3: CARD set3 micrographs [micrographs - single frame] / Data #4: CARD set4 micrographs [micrographs - single frame]Data #5: CARD set1 segments [picked particles - multiframe - unprocessed] Data #6: CARD set2 segments [picked particles - multiframe - unprocessed] Data #7: CARD set3 segments [picked particles - multiframe - unprocessed] Data #8: CARD set4 segments [picked particles - multiframe - unprocessed] Data #9: DeltProTM micrographs [micrographs - single frame] Data #10: DeltaProTM segments [picked particles - multiframe - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5891.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5891.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of filaments formed by a truncated MAVS lacking part of the proline-rich region and the C-terminal transmembrane domain | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.333 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
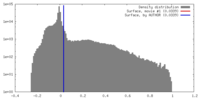
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Filaments formed by a truncated MAVS lacking part of the proline-...
| 全体 | 名称: Filaments formed by a truncated MAVS lacking part of the proline-rich region and the C-terminal transmembrane domain |
|---|---|
| 要素 |
|
-超分子 #1000: Filaments formed by a truncated MAVS lacking part of the proline-...
| 超分子 | 名称: Filaments formed by a truncated MAVS lacking part of the proline-rich region and the C-terminal transmembrane domain タイプ: sample / ID: 1000 / 集合状態: polymer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 1.396 MDa |
-分子 #1: Mitochondrial antiviral-signaling protein (MAVS)
| 分子 | 名称: Mitochondrial antiviral-signaling protein (MAVS) / タイプ: protein_or_peptide / ID: 1 / Name.synonym: IPS1, KIAA1271, VISA / 集合状態: polymer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 42.3 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Mitochondrial antiviral-signaling protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 10mM Tris-HCl, 150mM NaCl, 1mM DTT |
|---|---|
| グリッド | 詳細: Quantifoil grids coated with thin carbon on top |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100,000 x magnification |
| 特殊光学系 | エネルギーフィルター - 名称: JEOL omega filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 35.0 eV |
| 日付 | 2011年11月30日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 61950 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | IHRSR. Due to the smaller size of the dataset, we did not sort the data based on variations in symmetry parameters. |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 16.9 Å 想定した対称性 - らせんパラメータ - ΔΦ: 52.9 ° 想定した対称性 - らせんパラメータ - 軸対称性: C3 (3回回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.4 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Spider, MRC, EMAN, IMAGIC4D |
| CTF補正 | 詳細: Each filament segment |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)