+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3137 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Multiple capsid-stabilizing protein-RNA and protein-protein interactions revealed in a high-resolution structure of an emerging picornavirus causing neonatal sepsis | |||||||||
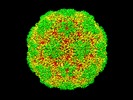
 マップデータ マップデータ | Reconstruction of human parechovirus 3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | picornavirus / parechovirus / human parechovirus 3 / HPeV3 / neonatal sepsis / cryoEM / image processing / single particle anaylsis | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-protein covalent cross-linking / : / : / host cell cytoplasmic vesicle membrane / cytoplasmic vesicle membrane / viral capsid / protein complex oligomerization / monoatomic ion channel activity / RNA helicase activity / symbiont entry into host cell ...RNA-protein covalent cross-linking / : / : / host cell cytoplasmic vesicle membrane / cytoplasmic vesicle membrane / viral capsid / protein complex oligomerization / monoatomic ion channel activity / RNA helicase activity / symbiont entry into host cell / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / structural molecule activity / RNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human parechovirus 3 (ウイルス) Human parechovirus 3 (ウイルス) | |||||||||
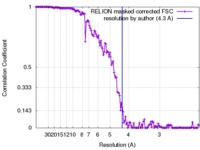
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Shakeel S / Westerhuis BM / Domanska A / Koning RI / Matadeen R / Koster AJ / Bakker AQ / Beaumont T / Wolthers KC / Butcher SJ | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2016 ジャーナル: Nat Commun / 年: 2016タイトル: Multiple capsid-stabilizing interactions revealed in a high-resolution structure of an emerging picornavirus causing neonatal sepsis. 著者: Shabih Shakeel / Brenda M Westerhuis / Ausra Domanska / Roman I Koning / Rishi Matadeen / Abraham J Koster / Arjen Q Bakker / Tim Beaumont / Katja C Wolthers / Sarah J Butcher /   要旨: The poorly studied picornavirus, human parechovirus 3 (HPeV3) causes neonatal sepsis with no therapies available. Our 4.3-Å resolution structure of HPeV3 on its own and at 15 Å resolution in ...The poorly studied picornavirus, human parechovirus 3 (HPeV3) causes neonatal sepsis with no therapies available. Our 4.3-Å resolution structure of HPeV3 on its own and at 15 Å resolution in complex with human monoclonal antibody Fabs demonstrates the expected picornavirus capsid structure with three distinct features. First, 25% of the HPeV3 RNA genome in 60 sites is highly ordered as confirmed by asymmetric reconstruction, and interacts with conserved regions of the capsid proteins VP1 and VP3. Second, the VP0 N terminus stabilizes the capsid inner surface, in contrast to other picornaviruses where on expulsion as VP4, it forms an RNA translocation channel. Last, VP1's hydrophobic pocket, the binding site for the antipicornaviral drug, pleconaril, is blocked and thus inappropriate for antiviral development. Together, these results suggest a direction for development of neutralizing antibodies, antiviral drugs based on targeting the RNA-protein interactions and dissection of virus assembly on the basis of RNA nucleation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3137.map.gz emd_3137.map.gz | 227.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3137-v30.xml emd-3137-v30.xml emd-3137.xml emd-3137.xml | 11.4 KB 11.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3137_fsc.xml emd_3137_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3137.tif emd_3137.tif | 1.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3137 http://ftp.pdbj.org/pub/emdb/structures/EMD-3137 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3137 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3137 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3137_validation.pdf.gz emd_3137_validation.pdf.gz | 313 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3137_full_validation.pdf.gz emd_3137_full_validation.pdf.gz | 312.1 KB | 表示 | |
| XML形式データ |  emd_3137_validation.xml.gz emd_3137_validation.xml.gz | 13.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3137 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3137 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3137 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3137 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5apmMC  3138C  3322C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10033 (タイトル: Multiple capsid-stabilizing protein-RNA and protein-protein interactions revealed in a high-resolution structure of an emerging picornavirus causing neonatal sepsis EMPIAR-10033 (タイトル: Multiple capsid-stabilizing protein-RNA and protein-protein interactions revealed in a high-resolution structure of an emerging picornavirus causing neonatal sepsisData size: 3.3 TB Data #1: Selected aligned movie frames, aligned-averaged micrographs and particle coordinates of human parechovirus 3 (HPeV3) virions [Aligned movie frames (.mrcs extension), aligned-averaged ...Data #1: Selected aligned movie frames, aligned-averaged micrographs and particle coordinates of human parechovirus 3 (HPeV3) virions [Aligned movie frames (.mrcs extension), aligned-averaged micrographs (.mrc extension) and particle coordinates (.box extension)] Data #2: Unaligned movie frames of human parechovirus 3 (HPeV3) virions [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3137.map.gz / 形式: CCP4 / 大きさ: 238.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3137.map.gz / 形式: CCP4 / 大きさ: 238.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of human parechovirus 3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.14 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human parechovirus 3 capsid structure
| 全体 | 名称: Human parechovirus 3 capsid structure |
|---|---|
| 要素 |
|
-超分子 #1000: Human parechovirus 3 capsid structure
| 超分子 | 名称: Human parechovirus 3 capsid structure / タイプ: sample / ID: 1000 詳細: The purified human parechovirus 3 virions were monodisperse. They were formaldehyde fixed. 集合状態: icosahedrally-symmetric virus with 60 copies each of VP0, VP3 and VP1 Number unique components: 3 |
|---|---|
| 分子量 | 実験値: 5 MDa / 理論値: 5 MDa 手法: theoretical weight determination from protein sequences, excluding viral genome. |
-超分子 #1: Human parechovirus 3
| 超分子 | 名称: Human parechovirus 3 / タイプ: virus / ID: 1 / Name.synonym: HPeV3 / NCBI-ID: 195055 / 生物種: Human parechovirus 3 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: HPeV3 |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| Host system | 生物種:  Chlorocebus aethiops (ミドリザル) / 組換細胞: Vero Chlorocebus aethiops (ミドリザル) / 組換細胞: Vero |
| 分子量 | 実験値: 5 MDa / 理論値: 5 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 280 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10 mM Tris-HCL, 150 mM NaCl, 1 mM MgCl2 |
| 染色 | タイプ: NEGATIVE / 詳細: Unstained |
| グリッド | 詳細: Quantifoil holey carbon on copper grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 92 % / チャンバー内温度: 92 K / 装置: LEICA EM GP / 手法: Blot for 2 seconds on one side before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80 K / 最高: 95 K / 平均: 87 K |
| アライメント法 | Legacy - 非点収差: Done as part of Cs corrector routine. |
| 詳細 | Cs corrector was used during imaging. |
| 日付 | 2014年1月21日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 6604 / 平均電子線量: 36 e/Å2 詳細: Total number of images collected were 6604. Total number of images used in the reconstruction were 1028. Every image is the average of seven aligned frames recorded by the direct electron detector. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.34 µm / 最小 デフォーカス(公称値): 0.42 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen cooled. 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー