+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1 complex (ATP-bound) with MoFeP during catalytic N2 reduction | |||||||||||||||
 マップデータ マップデータ | deepEMhancer sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | nitrogenase / FeP / nitrogen fixation / nitrogenase complex / OXIDOREDUCTASE | |||||||||||||||
| 生物種 |  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) | |||||||||||||||
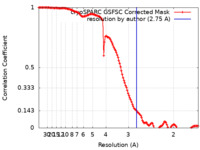
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.75 Å | |||||||||||||||
 データ登録者 データ登録者 | Rutledge HL / Cook B / Tezcan FA / Herzik MA | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Structures of the nitrogenase complex prepared under catalytic turnover conditions. 著者: Hannah L Rutledge / Brian D Cook / Hoang P M Nguyen / Mark A Herzik / F Akif Tezcan /  要旨: The enzyme nitrogenase couples adenosine triphosphate (ATP) hydrolysis to the multielectron reduction of atmospheric dinitrogen into ammonia. Despite extensive research, the mechanistic details of ...The enzyme nitrogenase couples adenosine triphosphate (ATP) hydrolysis to the multielectron reduction of atmospheric dinitrogen into ammonia. Despite extensive research, the mechanistic details of ATP-dependent energy transduction and dinitrogen reduction by nitrogenase are not well understood, requiring new strategies to monitor its structural dynamics during catalytic action. Here, we report cryo-electron microscopy structures of the nitrogenase complex prepared under enzymatic turnover conditions. We observe that asymmetry governs all aspects of the nitrogenase mechanism, including ATP hydrolysis, protein-protein interactions, and catalysis. Conformational changes near the catalytic iron-molybdenum cofactor are correlated with the nucleotide-hydrolysis state of the enzyme. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26759.map.gz emd_26759.map.gz | 180 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26759-v30.xml emd-26759-v30.xml emd-26759.xml emd-26759.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26759_fsc.xml emd_26759_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26759.png emd_26759.png | 55.1 KB | ||
| マスクデータ |  emd_26759_msk_1.map emd_26759_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26759.cif.gz emd-26759.cif.gz | 5.5 KB | ||
| その他 |  emd_26759_additional_1.map.gz emd_26759_additional_1.map.gz emd_26759_half_map_1.map.gz emd_26759_half_map_1.map.gz emd_26759_half_map_2.map.gz emd_26759_half_map_2.map.gz | 108.2 MB 200.2 MB 200.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26759 http://ftp.pdbj.org/pub/emdb/structures/EMD-26759 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26759 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26759 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26759_validation.pdf.gz emd_26759_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26759_full_validation.pdf.gz emd_26759_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_26759_validation.xml.gz emd_26759_validation.xml.gz | 21.6 KB | 表示 | |
| CIF形式データ |  emd_26759_validation.cif.gz emd_26759_validation.cif.gz | 28 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26759 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26759 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26759 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26759 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26759.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26759.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | deepEMhancer sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26759_msk_1.map emd_26759_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
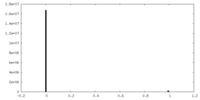
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_26759_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
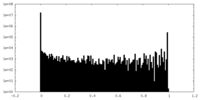
| 密度ヒストグラム |
-ハーフマップ: gold-standard half map A
| ファイル | emd_26759_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | gold-standard half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
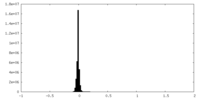
| 密度ヒストグラム |
-ハーフマップ: gold-standard half map B
| ファイル | emd_26759_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | gold-standard half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
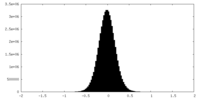
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1...
| 全体 | 名称: Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1 complex (ATP-bound) with MoFeP during catalytic N2 reduction |
|---|---|
| 要素 |
|
-超分子 #1: Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1...
| 超分子 | 名称: Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1 complex (ATP-bound) with MoFeP during catalytic N2 reduction タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Wild-type MoFeP and FeP were purified from the native organism, Azotobacter vinelandii. This map is the locally refined map for the FeP portion of the 1:1 complex. |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) |
| 分子量 | 理論値: 63 KDa |
-分子 #1: Nitrogenase iron protein gamma chain
| 分子 | 名称: Nitrogenase iron protein gamma chain / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) |
| 配列 | 文字列: MAMRQCAIYG KGGIGKSTTT QNLVAALAEM GKKVMIVGCD PKADSTRLIL HSKAQNTIME MAAEAGTVE DLELEDVLKA GYGGVKCVES GGPEPGVGCA GRGVITAINF LEEEGAYEDD L DFVFYDVL GDVVCGGFAM PIRENKAQEI YIVCSGEMMA MYAANNISKG ...文字列: MAMRQCAIYG KGGIGKSTTT QNLVAALAEM GKKVMIVGCD PKADSTRLIL HSKAQNTIME MAAEAGTVE DLELEDVLKA GYGGVKCVES GGPEPGVGCA GRGVITAINF LEEEGAYEDD L DFVFYDVL GDVVCGGFAM PIRENKAQEI YIVCSGEMMA MYAANNISKG IVKYANSGSV RL GGLICNS RNTDREDELI IALANKLGTQ MIHFVPRDNV VQRAEIRRMT VIEYDPKAKQ ADE YRALAR KVVDNKLLVI PNPITMDELE ELLMEFGIME VEDESIVGKT AEEV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Solutions were prepared and filtered immediately prior to the experiment. | |||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING | |||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER / 詳細: Custom manual plunger. Greater than 95% humidity.. | |||||||||
| 詳細 | 3.6 mg/mL FeP Sample also contained 1.44 mg/mL MoFeP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 温度 | 最低: 93.0 K / 最高: 123.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 14903 / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 135000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: EF / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)