+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24546 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli bL17-limitation ribosome assembly intermediate #38 | ||||||||||||
 マップデータ マップデータ | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) sharpened map | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  | ||||||||||||
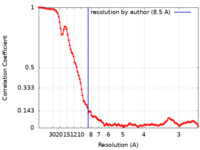
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.5 Å | ||||||||||||
 データ登録者 データ登録者 | Rabuck-Gibbons JN / Lyumkis D / Williamson JR | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Quantitative mining of compositional heterogeneity in cryo-EM datasets of ribosome assembly intermediates. 著者: Jessica N Rabuck-Gibbons / Dmitry Lyumkis / James R Williamson /  要旨: Single-particle cryoelectron microscopy (cryo-EM) offers a unique opportunity to characterize macromolecular structural heterogeneity by virtue of its ability to place distinct particle populations ...Single-particle cryoelectron microscopy (cryo-EM) offers a unique opportunity to characterize macromolecular structural heterogeneity by virtue of its ability to place distinct particle populations into different groups through computational classification. However, there is a dearth of tools for surveying the heterogeneity landscape, quantitatively analyzing heterogeneous particle populations after classification, deciding how many unique classes are represented by the data, and accurately cross-comparing reconstructions. Here, we develop a workflow that contains discovery and analysis modules to quantitatively mine cryo-EM data for sets of structures with maximal diversity. This workflow was applied to a dataset of E. coli 50S ribosome assembly intermediates, which are characterized by significant structural heterogeneity. We identified more detailed branchpoints in the assembly process and characterized the interactions of an assembly factor with immature intermediates. While the tools described here were developed for ribosome assembly, they should be broadly applicable to the analysis of other heterogeneous cryo-EM datasets. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24546.map.gz emd_24546.map.gz | 96.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24546-v30.xml emd-24546-v30.xml emd-24546.xml emd-24546.xml | 27.2 KB 27.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24546_fsc.xml emd_24546_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24546.png emd_24546.png | 84.7 KB | ||
| その他 |  emd_24546_additional_1.map.gz emd_24546_additional_1.map.gz emd_24546_additional_2.map.gz emd_24546_additional_2.map.gz emd_24546_additional_3.map.gz emd_24546_additional_3.map.gz emd_24546_half_map_1.map.gz emd_24546_half_map_1.map.gz emd_24546_half_map_2.map.gz emd_24546_half_map_2.map.gz | 16.2 MB 36 KB 14.5 MB 16.3 MB 16.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24546 http://ftp.pdbj.org/pub/emdb/structures/EMD-24546 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24546 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24546 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24546_validation.pdf.gz emd_24546_validation.pdf.gz | 444 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24546_full_validation.pdf.gz emd_24546_full_validation.pdf.gz | 443.6 KB | 表示 | |
| XML形式データ |  emd_24546_validation.xml.gz emd_24546_validation.xml.gz | 19.9 KB | 表示 | |
| CIF形式データ |  emd_24546_validation.cif.gz emd_24546_validation.cif.gz | 24.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24546 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24546 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24546 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24546 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10841 (タイトル: Raw movies and final particle stack for a dataset of bL17-limited E. coli ribosome assembly intermediates. EMPIAR-10841 (タイトル: Raw movies and final particle stack for a dataset of bL17-limited E. coli ribosome assembly intermediates.Data size: 1.3 TB Data #1: Particle stack file of L17-depleted ribosomal subunits used for 3D classification [picked particles - single frame - processed] Data #2: Movies of L17-depleted ribosomal subunits used for 3D classification [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24546.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24546.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: E. coli bL17-limitation ribosome assembly intermediate #38 (C-class)...
| ファイル | emd_24546_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: E. coli bL17-limitation ribosome assembly intermediate #38 (C-class)...
| ファイル | emd_24546_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) binarized map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: E. coli bL17-limitation ribosome assembly intermediate #38 (C-class)...
| ファイル | emd_24546_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) binned, unmasked map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: E. coli bL17-limitation ribosome assembly intermediate #38 (C-class)...
| ファイル | emd_24546_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) odd halfmap | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: E. coli bL17-limitation ribosome assembly intermediate #38 (C-class)...
| ファイル | emd_24546_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli bL17-limitation ribosome assembly intermediate #38 (C-class) even halfmap | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : bL17-depleted large ribosomal subunit assembly intermediate #38
| 全体 | 名称: bL17-depleted large ribosomal subunit assembly intermediate #38 |
|---|---|
| 要素 |
|
-超分子 #1: bL17-depleted large ribosomal subunit assembly intermediate #38
| 超分子 | 名称: bL17-depleted large ribosomal subunit assembly intermediate #38 タイプ: complex / ID: 1 / 親要素: 0 詳細: bL17-depleted large ribosomal subunit assembly intermediate #38 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.5 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 詳細: 3 ul of this sample was added to 3 plasma cleaned (Gatan, Solarus) 1.2mm hole, 1.3mm spacing holey gold grids (Russo and Passmore, 2014). Grids were manually frozen in liquid ethane.. | ||||||||||||
| 詳細 | depleted ribosome assembly intermediate purified by sucrose gradient |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | In order to account for highly preferred orientation of the specimen, data was acquired using a tilt of -20 degrees. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 833 / 平均露光時間: 10.0 sec. / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)