+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of an ALYREF-exon junction complex hexamer | |||||||||
 マップデータ マップデータ | Locally sharpened ALYREF(55-183)-EJC-RNA map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transcription-exort complex / TREX / splicing / exon junction complex / EJC / RNA export / RNA binding proteins / GENE REGULATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exon-exon junction subcomplex mago-y14 / transcription export complex / negative regulation of selenocysteine incorporation / regulation of nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / C5-methylcytidine-containing RNA reader activity / cellular response to selenite ion / selenocysteine insertion sequence binding / exon-exon junction complex / regulation of translation at postsynapse, modulating synaptic transmission / negative regulation of excitatory postsynaptic potential ...exon-exon junction subcomplex mago-y14 / transcription export complex / negative regulation of selenocysteine incorporation / regulation of nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / C5-methylcytidine-containing RNA reader activity / cellular response to selenite ion / selenocysteine insertion sequence binding / exon-exon junction complex / regulation of translation at postsynapse, modulating synaptic transmission / negative regulation of excitatory postsynaptic potential / Z-decay: degradation of maternal mRNAs by zygotically expressed factors / regulation of mRNA processing / Deadenylation of mRNA / embryonic cranial skeleton morphogenesis / Transport of the SLBP independent Mature mRNA / Transport of the SLBP Dependant Mature mRNA / M-decay: degradation of maternal mRNAs by maternally stored factors / poly(A) binding / mRNA 3'-end processing / Transport of Mature mRNA Derived from an Intronless Transcript / U2-type catalytic step 1 spliceosome / RNA export from nucleus / Transport of Mature mRNA derived from an Intron-Containing Transcript / RNA Polymerase II Transcription Termination / exploration behavior / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / regulation of alternative mRNA splicing, via spliceosome / associative learning / carbohydrate transmembrane transporter activity / mRNA export from nucleus / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / ribonucleoprotein complex binding / cellular response to brain-derived neurotrophic factor stimulus / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / RNA splicing / positive regulation of translation / response to organic cyclic compound / mRNA splicing, via spliceosome / ISG15 antiviral mechanism / Regulation of expression of SLITs and ROBOs / mRNA processing / osteoblast differentiation / RNA stem-loop binding / rRNA processing / regulation of translation / outer membrane-bounded periplasmic space / postsynapse / RNA helicase activity / negative regulation of translation / RNA helicase / nuclear speck / mRNA binding / neuronal cell body / glutamatergic synapse / dendrite / nucleolus / ATP hydrolysis activity / RNA binding / extracellular exosome / nucleoplasm / ATP binding / membrane / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
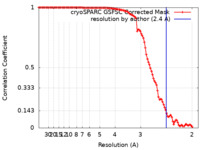
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Pacheco-Fiallos FB / Vorlaender MK / Plaschka C | |||||||||
| 資金援助 | European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: mRNA recognition and packaging by the human transcription-export complex. 著者: Belén Pacheco-Fiallos / Matthias K Vorländer / Daria Riabov-Bassat / Laura Fin / Francis J O'Reilly / Farja I Ayala / Ulla Schellhaas / Juri Rappsilber / Clemens Plaschka /    要旨: Newly made mRNAs are processed and packaged into mature ribonucleoprotein complexes (mRNPs) and are recognized by the essential transcription-export complex (TREX) for nuclear export. However, the ...Newly made mRNAs are processed and packaged into mature ribonucleoprotein complexes (mRNPs) and are recognized by the essential transcription-export complex (TREX) for nuclear export. However, the mechanisms of mRNP recognition and three-dimensional mRNP organization are poorly understood. Here we report cryo-electron microscopy and tomography structures of reconstituted and endogenous human mRNPs bound to the 2-MDa TREX complex. We show that mRNPs are recognized through multivalent interactions between the TREX subunit ALYREF and mRNP-bound exon junction complexes. Exon junction complexes can multimerize through ALYREF, which suggests a mechanism for mRNP organization. Endogenous mRNPs form compact globules that are coated by multiple TREX complexes. These results reveal how TREX may simultaneously recognize, compact and protect mRNAs to promote their packaging for nuclear export. The organization of mRNP globules provides a framework to understand how mRNP architecture facilitates mRNA biogenesis and export. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14803.map.gz emd_14803.map.gz | 9.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14803-v30.xml emd-14803-v30.xml emd-14803.xml emd-14803.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14803_fsc.xml emd_14803_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14803.png emd_14803.png | 170.9 KB | ||
| マスクデータ |  emd_14803_msk_1.map emd_14803_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14803.cif.gz emd-14803.cif.gz | 6.9 KB | ||
| その他 |  emd_14803_additional_1.map.gz emd_14803_additional_1.map.gz emd_14803_additional_2.map.gz emd_14803_additional_2.map.gz emd_14803_half_map_1.map.gz emd_14803_half_map_1.map.gz emd_14803_half_map_2.map.gz emd_14803_half_map_2.map.gz | 117.9 MB 62.1 MB 115.8 MB 115.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14803 http://ftp.pdbj.org/pub/emdb/structures/EMD-14803 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14803 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14803 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14803_validation.pdf.gz emd_14803_validation.pdf.gz | 811.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14803_full_validation.pdf.gz emd_14803_full_validation.pdf.gz | 810.9 KB | 表示 | |
| XML形式データ |  emd_14803_validation.xml.gz emd_14803_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_14803_validation.cif.gz emd_14803_validation.cif.gz | 24.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14803 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14803 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14803 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14803 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7znjMC  7znkC  7znlC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14803.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14803.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally sharpened ALYREF(55-183)-EJC-RNA map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.945 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14803_msk_1.map emd_14803_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
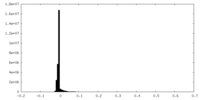
| 密度ヒストグラム |
-追加マップ: Globally sharpened ALYREF(55-183)-EJC-RNA map
| ファイル | emd_14803_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Globally sharpened ALYREF(55-183)-EJC-RNA map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
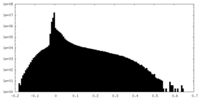
| 密度ヒストグラム |
-追加マップ: Unsharpened ALYREF(55-183)-EJC-RNA map
| ファイル | emd_14803_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened ALYREF(55-183)-EJC-RNA map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ALYREF(55-183)-EJC-RNA half map
| ファイル | emd_14803_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ALYREF(55-183)-EJC-RNA half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
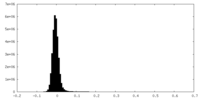
| 密度ヒストグラム |
-ハーフマップ: ALYREF(55-183)-EJC-RNA half map
| ファイル | emd_14803_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ALYREF(55-183)-EJC-RNA half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
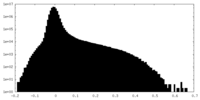
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexameric complex between the TREX subunit ALYREF and the exon ju...
| 全体 | 名称: Hexameric complex between the TREX subunit ALYREF and the exon junction complex |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric complex between the TREX subunit ALYREF and the exon ju...
| 超分子 | 名称: Hexameric complex between the TREX subunit ALYREF and the exon junction complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 詳細: Assembled with truncated MBP-tagged ALYREF (residues 55-183) |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 650 KDa |
-分子 #1: Eukaryotic initiation factor 4A-III, N-terminally processed
| 分子 | 名称: Eukaryotic initiation factor 4A-III, N-terminally processed タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 43.819367 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTKVEFETSE EVDVTPTFDT MGLREDLLRG IYAYGFEKPS AIQQRAIKQI IKGRDVIAQS QSGTGKTATF SISVLQCLDI QVRETQALI LAPTRELAVQ IQKGLLALGD YMNVQCHACI GGTNVGEDIR KLDYGQHVVA GTPGRVFDMI RRRSLRTRAI K MLVLDEAD ...文字列: MTKVEFETSE EVDVTPTFDT MGLREDLLRG IYAYGFEKPS AIQQRAIKQI IKGRDVIAQS QSGTGKTATF SISVLQCLDI QVRETQALI LAPTRELAVQ IQKGLLALGD YMNVQCHACI GGTNVGEDIR KLDYGQHVVA GTPGRVFDMI RRRSLRTRAI K MLVLDEAD EMLNKGFKEQ IYDVYRYLPP ATQVVLISAT LPHEILEMTN KFMTDPIRIL VKRDELTLEG IKQFFVAVER EE WKFDTLC DLYDTLTITQ AVIFCNTKRK VDWLTEKMRE ANFTVSSMHG DMPQKERESI MKEFRSGASR VLISTDVWAR GLD VPQVSL IINYDLPNNR ELYIHRIGRS GRYGRKGVAI NFVKNDDIRI LRDIEQYYST QIDEMP UniProtKB: Eukaryotic initiation factor 4A-III |
-分子 #2: Protein mago nashi homolog
| 分子 | 名称: Protein mago nashi homolog / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 17.189625 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MESDFYLRYY VGHKGKFGHE FLEFEFRPDG KLRYANNSNY KNDVMIRKEA YVHKSVMEEL KRIIDDSEIT KEDDALWPPP DRVGRQELE IVIGDEHISF TTSKIGSLID VNQSKDPEGL RVFYYLVQDL KCLVFSLIGL HFKIKPI UniProtKB: Protein mago nashi homolog |
-分子 #3: RNA-binding protein 8A
| 分子 | 名称: RNA-binding protein 8A / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.370525 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPQRSVEGWI LFVTGVHEEA TEEDIHDKFA EYGEIKNIHL NLDRRTGYLK GYTLVEYETY KEAQAAMEGL NGQDLMGQPI SVDWCFVRG PP UniProtKB: RNA-binding protein 8A |
-分子 #4: Maltose/maltodextrin-binding periplasmic protein,THO complex subunit 4
| 分子 | 名称: Maltose/maltodextrin-binding periplasmic protein,THO complex subunit 4 タイプ: protein_or_peptide / ID: 4 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 57.598855 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQLSHHHHHH HHHHSSGMKI EEGKLVIWIN GDKGYNGLAE VGKKFEKDTG IKVTVEHPDK LEEKFPQVAA TGDGPDIIFW AHDRFGGYA QSGLLAEITP DKAFQDKLYP FTWDAVRYNG KLIAYPIAVE ALSLIYNKDL LPNPPKTWEE IPALDKELKA K GKSALMFN ...文字列: MQLSHHHHHH HHHHSSGMKI EEGKLVIWIN GDKGYNGLAE VGKKFEKDTG IKVTVEHPDK LEEKFPQVAA TGDGPDIIFW AHDRFGGYA QSGLLAEITP DKAFQDKLYP FTWDAVRYNG KLIAYPIAVE ALSLIYNKDL LPNPPKTWEE IPALDKELKA K GKSALMFN LQEPYFTWPL IAADGGYAFK YENGKYDIKD VGVDNAGAKA GLTFLVDLIK NKHMNADTDY SIAEAAFNKG ET AMTINGP WAWSNIDTSK VNYGVTVLPT FKGQPSKPFV GVLSAGINAA SPNKELAKEF LENYLLTDEG LEAVNKDKPL GAV ALKSYE EELAKDPRIA ATMENAQKGE IMPNIPQMSA FWYAVRTAVI NAASGRQTVD EALKDAQTSS GLEVLFQGPG SSGI RNRPA IARGAAGGGG RNRPAPYSRP KQLPDKWQHD LFDSGFGGGA GVETGGKLLV SNLDFGVSDA DIQELFAEFG TLKKA AVHY DRSGRSLGTA DVHFERKADA LKAMKQYNGV PLDGRPMNIQ LVT UniProtKB: Maltose/maltodextrin-binding periplasmic protein, THO complex subunit 4 |
-分子 #5: RNA
| 分子 | 名称: RNA / タイプ: rna / ID: 5 / コピー数: 6 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.792037 KDa |
| 配列 | 文字列: UUUUUU |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 7 / コピー数: 6 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)