+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12680 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure (model_2a) of the RC-dLH complex from Gemmatimonas phototrophica at 2.5 A | ||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthesis, light reaction / photosynthetic electron transport in photosystem II / chlorophyll binding / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / electron transfer activity / iron ion binding / heme binding ...organelle inner membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthesis, light reaction / photosynthetic electron transport in photosystem II / chlorophyll binding / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / electron transfer activity / iron ion binding / heme binding / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Gemmatimonas phototrophica (バクテリア) Gemmatimonas phototrophica (バクテリア) | ||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Qian P / Koblizek M | ||||||||||||||||||||||||
| 資金援助 |  チェコ, チェコ,  英国, European Union, 7件 英国, European Union, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: 2.4-Å structure of the double-ring photosystem. 著者: Pu Qian / Alastair T Gardiner / Ivana Šímová / Katerina Naydenova / Tristan I Croll / Philip J Jackson / Nupur / Miroslav Kloz / Petra Čubáková / Marek Kuzma / Yonghui Zeng / Pablo ...著者: Pu Qian / Alastair T Gardiner / Ivana Šímová / Katerina Naydenova / Tristan I Croll / Philip J Jackson / Nupur / Miroslav Kloz / Petra Čubáková / Marek Kuzma / Yonghui Zeng / Pablo Castro-Hartmann / Bart van Knippenberg / Kenneth N Goldie / David Kaftan / Pavel Hrouzek / Jan Hájek / Jon Agirre / C Alistair Siebert / David Bína / Kasim Sader / Henning Stahlberg / Roman Sobotka / Christopher J Russo / Tomáš Polívka / C Neil Hunter / Michal Koblížek /     要旨: Phototrophic Gemmatimonadetes evolved the ability to use solar energy following horizontal transfer of photosynthesis-related genes from an ancient phototrophic proteobacterium. The electron cryo- ...Phototrophic Gemmatimonadetes evolved the ability to use solar energy following horizontal transfer of photosynthesis-related genes from an ancient phototrophic proteobacterium. The electron cryo-microscopy structure of the photosystem at 2.4 Å reveals a unique, double-ring complex. Two unique membrane-extrinsic polypeptides, RC-S and RC-U, hold the central type 2 reaction center (RC) within an inner 16-subunit light-harvesting 1 (LH1) ring, which is encircled by an outer 24-subunit antenna ring (LHh) that adds light-gathering capacity. Femtosecond kinetics reveal the flow of energy within the RC-dLH complex, from the outer LHh ring to LH1 and then to the RC. This structural and functional study shows that has independently evolved its own compact, robust, and highly effective architecture for harvesting and trapping solar energy. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12680.map.gz emd_12680.map.gz | 18.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12680-v30.xml emd-12680-v30.xml emd-12680.xml emd-12680.xml | 33.8 KB 33.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
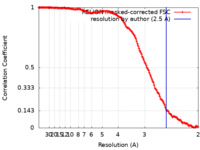
| FSC (解像度算出) |  emd_12680_fsc.xml emd_12680_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12680.png emd_12680.png | 75.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12680 http://ftp.pdbj.org/pub/emdb/structures/EMD-12680 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12680 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12680 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12680_validation.pdf.gz emd_12680_validation.pdf.gz | 345.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12680_full_validation.pdf.gz emd_12680_full_validation.pdf.gz | 344.9 KB | 表示 | |
| XML形式データ |  emd_12680_validation.xml.gz emd_12680_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_12680_validation.cif.gz emd_12680_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12680 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12680 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12680 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12680 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7o0vMC  7o0uC  7o0wC  7o0xC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10681 (タイトル: Movies of a photosynthetic reaction centre - double light-harvesting 1 complex from the bacterium G. phototrophica EMPIAR-10681 (タイトル: Movies of a photosynthetic reaction centre - double light-harvesting 1 complex from the bacterium G. phototrophicaData size: 12.1 TB Data #1: Unaligned multi-frame micrographs of RC-dLH1 complexes on HexAuFoil support [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12680.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12680.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.99946 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : RC-dLH (model_2a)
+超分子 #1: RC-dLH (model_2a)
+分子 #1: LHh-alpha
+分子 #2: LHh-alpha
+分子 #3: Light-harvesting protein B:885 subunit beta
+分子 #4: Light-harvesting protein B:885 subunit beta
+分子 #5: MULTIHEME_CYTC domain-containing protein
+分子 #6: RC-S
+分子 #7: PRCH domain-containing protein
+分子 #8: RC-Hc
+分子 #9: Photosynthetic reaction center L subunit
+分子 #10: RC-M
+分子 #11: LHC domain-containing protein
+分子 #12: LHC domain-containing protein
+分子 #14: BACTERIOCHLOROPHYLL A
+分子 #15: DODECYL-BETA-D-MALTOSIDE
+分子 #16: (2~{E},4~{E},6~{E},10~{E},12~{E},14~{E},16~{E},18~{E},20~{E},22~{...
+分子 #17: HEME C
+分子 #18: (2~{S},3~{S},4~{S},5~{S})-4,5-diacetyloxy-3-oxidanyl-oxane-2-carb...
+分子 #19: 2-acetamido-2-deoxy-alpha-D-glucopyranose
+分子 #20: (19R,22S)-25-amino-22-hydroxy-22-oxido-16-oxo-17,21,23-trioxa-22l...
+分子 #21: (2R,5R,11R,14R)-5,8,11-trihydroxy-5,11-dioxido-17-oxo-2,14-bis(te...
+分子 #22: (1R)-2-{[(S)-{[(2S)-2,3-dihydroxypropyl]oxy}(hydroxy)phosphoryl]o...
+分子 #23: MENAQUINONE 8
+分子 #24: [(2~{S})-3-[(2~{R},3~{R},4~{R},5~{S},6~{R})-6-(hydroxymethyl)-5-[...
+分子 #25: BACTERIOPHEOPHYTIN A
+分子 #26: FE (III) ION
+分子 #27: SPIRILLOXANTHIN
+分子 #28: [(2~{S})-3-[(2~{R},3~{R},4~{S},5~{S},6~{R})-6-(hydroxymethyl)-3,4...
+分子 #29: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.5 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
詳細: The final purified complex is in 20mM Tris.Cl, 0.025 DDM, PH 8.0 buffer. | ||||||
| グリッド | モデル: Homemade / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 30.0 nm / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER / 詳細: Talmon type. | ||||||
| 詳細 | The protein complex was isolated from photosynthetic membrane using detergent beta-DDM. After purification, concentrated protein sample solution was stored in LN before using. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 19865 / 平均露光時間: 1.2 sec. / 平均電子線量: 24.8 e/Å2 詳細: All movies were recorded from a HexAuFoil grid with a special EPU version, which can recognise 300 nm holes on the grid. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.4 µm / 最小 デフォーカス(公称値): -0.8 µm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)