+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12181 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of Icosahedrally averaged native core of Pyruvate Dehydrogenase Complex from Ch. thermophilum | |||||||||
 マップデータ マップデータ | Symmetrically averaged (Icosahedral Symmetry) Pyruvate Dehydrogenase Complex core from Ch. thermophilum. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Dihydrolipoyl / transacetylase / E2 / Pyruvate / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dihydrolipoyllysine-residue acetyltransferase / dihydrolipoyllysine-residue acetyltransferase activity / acetyl-CoA biosynthetic process from pyruvate / : 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.9 Å | |||||||||
 データ登録者 データ登録者 | Skalidis I / Kyrilis FL / Tueting C / Semchonok DA / Kastritis PL | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2021 ジャーナル: Cell Rep / 年: 2021タイトル: Integrative structure of a 10-megadalton eukaryotic pyruvate dehydrogenase complex from native cell extracts. 著者: Fotis L Kyrilis / Dmitry A Semchonok / Ioannis Skalidis / Christian Tüting / Farzad Hamdi / Francis J O'Reilly / Juri Rappsilber / Panagiotis L Kastritis /   要旨: The pyruvate dehydrogenase complex (PDHc) is a giant enzymatic assembly involved in pyruvate oxidation. PDHc components have been characterized in isolation, but the complex's quaternary structure ...The pyruvate dehydrogenase complex (PDHc) is a giant enzymatic assembly involved in pyruvate oxidation. PDHc components have been characterized in isolation, but the complex's quaternary structure has remained elusive due to sheer size, heterogeneity, and plasticity. Here, we identify fully assembled Chaetomium thermophilum α-keto acid dehydrogenase complexes in native cell extracts and characterize their domain arrangements utilizing mass spectrometry, activity assays, crosslinking, electron microscopy (EM), and computational modeling. We report the cryo-EM structure of the PDHc core and observe unique features of the previously unknown native state. The asymmetric reconstruction of the 10-MDa PDHc resolves spatial proximity of its components, agrees with stoichiometric data (60 E2p:12 E3BP:∼20 E1p: ≤ 12 E3), and proposes a minimum reaction path among component enzymes. The PDHc shows the presence of a dynamic pyruvate oxidation compartment, organized by core and peripheral protein species. Our data provide a framework for further understanding PDHc and α-keto acid dehydrogenase complex structure and function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12181.map.gz emd_12181.map.gz | 45.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12181-v30.xml emd-12181-v30.xml emd-12181.xml emd-12181.xml | 30.8 KB 30.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12181_fsc.xml emd_12181_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12181.png emd_12181.png | 64.6 KB | ||
| Filedesc metadata |  emd-12181.cif.gz emd-12181.cif.gz | 6.3 KB | ||
| その他 |  emd_12181_additional_1.map.gz emd_12181_additional_1.map.gz emd_12181_additional_2.map.gz emd_12181_additional_2.map.gz emd_12181_additional_3.map.gz emd_12181_additional_3.map.gz emd_12181_additional_4.map.gz emd_12181_additional_4.map.gz emd_12181_additional_5.map.gz emd_12181_additional_5.map.gz emd_12181_additional_6.map.gz emd_12181_additional_6.map.gz emd_12181_half_map_1.map.gz emd_12181_half_map_1.map.gz emd_12181_half_map_2.map.gz emd_12181_half_map_2.map.gz | 46.2 MB 46.2 MB 46.2 MB 46.2 MB 46.2 MB 46.1 MB 45.9 MB 45.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12181 http://ftp.pdbj.org/pub/emdb/structures/EMD-12181 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12181 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12181 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12181_validation.pdf.gz emd_12181_validation.pdf.gz | 460.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12181_full_validation.pdf.gz emd_12181_full_validation.pdf.gz | 459.9 KB | 表示 | |
| XML形式データ |  emd_12181_validation.xml.gz emd_12181_validation.xml.gz | 14.3 KB | 表示 | |
| CIF形式データ |  emd_12181_validation.cif.gz emd_12181_validation.cif.gz | 20.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12181 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12181 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12181 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12181 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7bgjMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10625 (タイトル: Raw cryo- and negative stain micrographs of a native extract from C. thermophilum used to reconstruct oxo acid dehydrogenase complexes EMPIAR-10625 (タイトル: Raw cryo- and negative stain micrographs of a native extract from C. thermophilum used to reconstruct oxo acid dehydrogenase complexesData size: 5.4 TB Data #1: Unaligned multiframe and summed frame mrc files of the datasets presented in the corresponding Cell Reports manuscript, by Kastritis et al. Please read the README file for more information ...Data #1: Unaligned multiframe and summed frame mrc files of the datasets presented in the corresponding Cell Reports manuscript, by Kastritis et al. Please read the README file for more information [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12181.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12181.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Symmetrically averaged (Icosahedral Symmetry) Pyruvate Dehydrogenase Complex core from Ch. thermophilum. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.1765 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Asymmetrical Pyruvate Dehydrogenase Complex from Ch. thermophilum.
| ファイル | emd_12181_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetrical Pyruvate Dehydrogenase Complex from Ch. thermophilum. | ||||||||||||
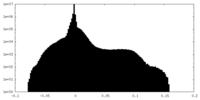
| 投影像・断面図 |
| ||||||||||||
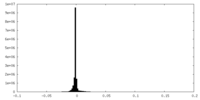
| 密度ヒストグラム |
-追加マップ: Asymmetrical Pyruvate Dehydrogenase Complex from Ch. thermophilum, Half-map...
| ファイル | emd_12181_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetrical Pyruvate Dehydrogenase Complex from Ch. thermophilum, Half-map 1. | ||||||||||||
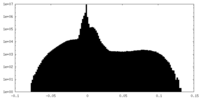
| 投影像・断面図 |
| ||||||||||||
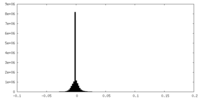
| 密度ヒストグラム |
-追加マップ: Asymmetrical Pyruvate Dehydrogenase Complex from Ch. thermophilum, Half-map...
| ファイル | emd_12181_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetrical Pyruvate Dehydrogenase Complex from Ch. thermophilum, Half-map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Asymmetrical Pyruvate Dehydrogenase Complex core from Ch. thermophilum....
| ファイル | emd_12181_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetrical Pyruvate Dehydrogenase Complex core from Ch. thermophilum. Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Asymmetrical Pyruvate Dehydrogenase Complex core from Ch. thermophilum....
| ファイル | emd_12181_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetrical Pyruvate Dehydrogenase Complex core from Ch. thermophilum. Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Asymmetrical Pyruvate Dehydrogenase Complex core from Ch. thermophilum....
| ファイル | emd_12181_additional_6.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetrical Pyruvate Dehydrogenase Complex core from Ch. thermophilum. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Symmetrically averaged (Icosahedral Symmetry) Pyruvate Dehydrogenase Complex core...
| ファイル | emd_12181_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Symmetrically averaged (Icosahedral Symmetry) Pyruvate Dehydrogenase Complex core from Ch. thermophilum, Half-map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Symmetrically averaged (Icosahedral Symmetry) Pyruvate Dehydrogenase Complex core...
| ファイル | emd_12181_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Symmetrically averaged (Icosahedral Symmetry) Pyruvate Dehydrogenase Complex core from Ch. thermophilum, Half-map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Native 60-mer core of Pyruvate Dehydrogenase Complex
| 全体 | 名称: Native 60-mer core of Pyruvate Dehydrogenase Complex |
|---|---|
| 要素 |
|
-超分子 #1: Native 60-mer core of Pyruvate Dehydrogenase Complex
| 超分子 | 名称: Native 60-mer core of Pyruvate Dehydrogenase Complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 3 MDa |
-分子 #1: Acetyltransferase component of pyruvate dehydrogenase complex
| 分子 | 名称: Acetyltransferase component of pyruvate dehydrogenase complex タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: dihydrolipoyllysine-residue acetyltransferase |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 48.777488 KDa |
| 配列 | 文字列: MLAQVLRRQA LQHVRLARAA APSLTRWYAS YPPHTIVKMP ALSPTMTSGN IGAWQKKPGD AITPGEVLVE IETDKAQMDF EFQEEGVLA KILKETGEKD VAVGSPIAVL VEEGTDINAF QNFTLEDAGG DAAAPAAPAK EELAKAETAP TPASTSAPEP E ETTSTGKL ...文字列: MLAQVLRRQA LQHVRLARAA APSLTRWYAS YPPHTIVKMP ALSPTMTSGN IGAWQKKPGD AITPGEVLVE IETDKAQMDF EFQEEGVLA KILKETGEKD VAVGSPIAVL VEEGTDINAF QNFTLEDAGG DAAAPAAPAK EELAKAETAP TPASTSAPEP E ETTSTGKL EPALDREPNV SFAAKKLAHE LDVPLKALKG TGPGGKITEE DVKKAASAPA AAAAAPGAAY QDIPISNMRK TI ATRLKES VSENPHFFVT SELSVSKLLK LRQALNSSAE GRYKLSVNDF LIKAIAVACK RVPAVNSSWR DGVIRQFDTV DVS VAVATP TGLITPIVKG VEAKGLETIS ATVKELAKKA RDGKLKPEDY QGGTISISNM GMNPAVERFT AIINPPQAAI LAVG TTKKV AVPVENEDGT TGVEWDDQIV VTASFDHKVV DGAVGAEWMR ELKKVVENPL ELLL UniProtKB: Dihydrolipoyllysine-residue acetyltransferase component of pyruvate dehydrogenase complex, mitochondrial |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 200.0 mM / 構成要素 - 式: NH4CH2COOH / 構成要素 - 名称: Ammonium acetate |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: For plunging, blot force 2 and blotting time of 6 sec were applied.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 温度 | 最低: 77.15 K / 最高: 103.15 K |
| アライメント法 | Coma free - Residual tilt: 14.7 mrad |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 2593 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 44067 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 444-647 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | The initial model was used to generate our model using MODELLER. This inital model was fitted into the density using ChimeraX and refined in real space using PHENIX. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 103.25 |
| 得られたモデル |  PDB-7bgj: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X