+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12042 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human CAK bound to ICEC0942 at 2.5 Angstroms resolution | |||||||||
 マップデータ マップデータ | Post-processed (sharpened, filtered) cryo-EM map, re-boxed into a 128 x 128 x 128 pixel box | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Kinase / protein complex / small molecules inhibitor / CDK-activating kinase / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cyclin-dependent protein kinase activating kinase holoenzyme complex / ventricular system development / snRNA transcription by RNA polymerase II / CAK-ERCC2 complex / transcription factor TFIIK complex / adult heart development / transcription factor TFIIH core complex / transcription factor TFIIH holo complex / cyclin-dependent protein serine/threonine kinase activator activity / [RNA-polymerase]-subunit kinase ...cyclin-dependent protein kinase activating kinase holoenzyme complex / ventricular system development / snRNA transcription by RNA polymerase II / CAK-ERCC2 complex / transcription factor TFIIK complex / adult heart development / transcription factor TFIIH core complex / transcription factor TFIIH holo complex / cyclin-dependent protein serine/threonine kinase activator activity / [RNA-polymerase]-subunit kinase / RNA Polymerase I Transcription Termination / cyclin-dependent protein serine/threonine kinase regulator activity / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / mRNA Capping / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / regulation of G1/S transition of mitotic cell cycle / RNA Polymerase I Transcription Initiation / RNA polymerase II transcribes snRNA genes / Tat-mediated elongation of the HIV-1 transcript / Formation of HIV-1 elongation complex containing HIV-1 Tat / ATP-dependent activity, acting on DNA / cyclin-dependent kinase / Formation of HIV elongation complex in the absence of HIV Tat / cyclin-dependent protein serine/threonine kinase activity / cyclin-dependent protein kinase holoenzyme complex / Cyclin E associated events during G1/S transition / RNA Polymerase II Transcription Elongation / Cyclin A/B1/B2 associated events during G2/M transition / Formation of RNA Pol II elongation complex / Cyclin A:Cdk2-associated events at S phase entry / RNA Polymerase II Pre-transcription Events / RNA polymerase II CTD heptapeptide repeat kinase activity / male germ cell nucleus / nucleotide-excision repair / transcription initiation at RNA polymerase II promoter / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase I Promoter Escape / positive regulation of smooth muscle cell proliferation / NoRC negatively regulates rRNA expression / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / fibrillar center / Formation of Incision Complex in GG-NER / response to calcium ion / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / G1/S transition of mitotic cell cycle / Cyclin D associated events in G1 / RUNX1 regulates transcription of genes involved in differentiation of HSCs / transcription by RNA polymerase II / protein stabilization / regulation of cell cycle / protein kinase activity / cell cycle / cell division / DNA repair / protein serine kinase activity / protein serine/threonine kinase activity / regulation of transcription by RNA polymerase II / negative regulation of apoptotic process / perinuclear region of cytoplasm / positive regulation of transcription by RNA polymerase II / zinc ion binding / nucleoplasm / ATP binding / nucleus / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Greber BJ / Remis J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biophys J / 年: 2021 ジャーナル: Biophys J / 年: 2021タイトル: 2.5 Å-resolution structure of human CDK-activating kinase bound to the clinical inhibitor ICEC0942. 著者: Basil J Greber / Jonathan Remis / Simak Ali / Eva Nogales /   要旨: The human CDK-activating kinase (CAK), composed of CDK7, cyclin H, and MAT1, is involved in the control of transcription initiation and the cell cycle. Because of these activities, it has been ...The human CDK-activating kinase (CAK), composed of CDK7, cyclin H, and MAT1, is involved in the control of transcription initiation and the cell cycle. Because of these activities, it has been identified as a promising target for cancer chemotherapy. A number of CDK7 inhibitors have entered clinical trials, among them ICEC0942 (also known as CT7001). Structural information can aid in improving the affinity and specificity of such drugs or drug candidates, reducing side effects in patients. Here, we have determined the structure of the human CAK in complex with ICEC0942 at 2.5 Å-resolution using cryogenic electron microscopy. Our structure reveals conformational differences of ICEC0942 compared with previous X-ray crystal structures of the CDK2-bound complex, and highlights the critical ability of cryogenic electron microscopy to resolve structures of drug-bound protein complexes without the need to crystalize the protein target. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12042.map.gz emd_12042.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12042-v30.xml emd-12042-v30.xml emd-12042.xml emd-12042.xml | 23.8 KB 23.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_12042.png emd_12042.png | 134.1 KB | ||
| マスクデータ |  emd_12042_msk_1.map emd_12042_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12042.cif.gz emd-12042.cif.gz | 7.2 KB | ||
| その他 |  emd_12042_half_map_1.map.gz emd_12042_half_map_1.map.gz emd_12042_half_map_2.map.gz emd_12042_half_map_2.map.gz | 49.7 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12042 http://ftp.pdbj.org/pub/emdb/structures/EMD-12042 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12042 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12042 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12042_validation.pdf.gz emd_12042_validation.pdf.gz | 923.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12042_full_validation.pdf.gz emd_12042_full_validation.pdf.gz | 922.7 KB | 表示 | |
| XML形式データ |  emd_12042_validation.xml.gz emd_12042_validation.xml.gz | 11.6 KB | 表示 | |
| CIF形式データ |  emd_12042_validation.cif.gz emd_12042_validation.cif.gz | 13.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12042 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12042 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12042 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12042 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7b5oMC  7b5qMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10561 (タイトル: Cryo-EM structure of the human CDK-activating kinase bound to the clinical inhibitor ICEC0942 EMPIAR-10561 (タイトル: Cryo-EM structure of the human CDK-activating kinase bound to the clinical inhibitor ICEC0942Data size: 3.8 TB Data #1: Unaligned movies of human CAK-ICEC0942, dataset 1 [micrographs - multiframe] Data #2: Unaligned movies of human CAK-ICEC0942, dataset 3 [micrographs - multiframe] Data #3: Unaligned movies of human CAK-ICEC0942, dataset 2 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12042.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12042.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed (sharpened, filtered) cryo-EM map, re-boxed into a 128 x 128 x 128 pixel box | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.029 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12042_msk_1.map emd_12042_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
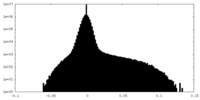
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-map
| ファイル | emd_12042_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-map
| ファイル | emd_12042_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CDK-activating kinase (CAK) in complex with ICEC0942
| 全体 | 名称: CDK-activating kinase (CAK) in complex with ICEC0942 |
|---|---|
| 要素 |
|
-超分子 #1: CDK-activating kinase (CAK) in complex with ICEC0942
| 超分子 | 名称: CDK-activating kinase (CAK) in complex with ICEC0942 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 120 KDa |
-分子 #1: CDK-activating kinase assembly factor MAT1
| 分子 | 名称: CDK-activating kinase assembly factor MAT1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 38.13234 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGSSHHHHHH ENLYFQSNAM DDQGCPRCKT TKYRNPSLKL MVNVCGHTLC ESCVDLLFVR GAGNCPECGT PLRKSNFRVQ LFEDPTVDK EVEIRKKVLK IYNKREEDFP SLREYNDFLE EVEEIVFNLT NNVDLDNTKK KMEIYQKENK DVIQKNKLKL T REQEELEE ...文字列: MGSSHHHHHH ENLYFQSNAM DDQGCPRCKT TKYRNPSLKL MVNVCGHTLC ESCVDLLFVR GAGNCPECGT PLRKSNFRVQ LFEDPTVDK EVEIRKKVLK IYNKREEDFP SLREYNDFLE EVEEIVFNLT NNVDLDNTKK KMEIYQKENK DVIQKNKLKL T REQEELEE ALEVERQENE QRRLFIQKEE QLQQILKRKN KQAFLDELES SDLPVALLLA QHKDRSTQLE MQLEKPKPVK PV TFSTGIK MGQHISLAPI HKLEEALYEY QPLQIETYGP HVPELEMLGR LGYLNHVRAA SPQDLAGGYT SSLACHRALQ DAF SGLFWQ PS UniProtKB: CDK-activating kinase assembly factor MAT1 |
-分子 #2: Cyclin-H
| 分子 | 名称: Cyclin-H / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.695473 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MYHNSSQKRH WTFSSEEQLA RLRADANRKF RCKAVANGKV LPNDPVFLEP HEEMTLCKYY EKRLLEFCSV FKPAMPRSVV GTACMYFKR FYLNNSVMEY HPRIIMLTCA FLACKVDEFN VSSPQFVGNL RESPLGQEKA LEQILEYELL LIQQLNFHLI V HNPYRPFE ...文字列: MYHNSSQKRH WTFSSEEQLA RLRADANRKF RCKAVANGKV LPNDPVFLEP HEEMTLCKYY EKRLLEFCSV FKPAMPRSVV GTACMYFKR FYLNNSVMEY HPRIIMLTCA FLACKVDEFN VSSPQFVGNL RESPLGQEKA LEQILEYELL LIQQLNFHLI V HNPYRPFE GFLIDLKTRY PILENPEILR KTADDFLNRI ALTDAYLLYT PSQIALTAIL SSASRAGITM ESYLSESLML KE NRTCLSQ LLDIMKSMRN LVKKYEPPRS EEVAVLKQKL ERCHSAELAL NVITKKRKGY EDDDYVSKKS KHEEEEWTDD DLV ESL UniProtKB: Cyclin-H |
-分子 #3: Cyclin-dependent kinase 7
| 分子 | 名称: Cyclin-dependent kinase 7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: cyclin-dependent kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 43.65107 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MASWSHPQFE KGGGSGGGSG GGSWSHPQFE KSGGGSENLY FQSNAMALDV KSRAKRYEKL DFLGEGQFAT VYKARDKNTN QIVAIKKIK LGHRSEAKDG INRTALREIK LLQELSHPNI IGLLDAFGHK SNISLVFDFM ETDLEVIIKD NSLVLTPSHI K AYMLMTLQ ...文字列: MASWSHPQFE KGGGSGGGSG GGSWSHPQFE KSGGGSENLY FQSNAMALDV KSRAKRYEKL DFLGEGQFAT VYKARDKNTN QIVAIKKIK LGHRSEAKDG INRTALREIK LLQELSHPNI IGLLDAFGHK SNISLVFDFM ETDLEVIIKD NSLVLTPSHI K AYMLMTLQ GLEYLHQHWI LHRDLKPNNL LLDENGVLKL ADFGLAKSFG SPNRAYTHQV VTRWYRAPEL LFGARMYGVG VD MWAVGCI LAELLLRVPF LPGDSDLDQL TRIFETLGTP TEEQWPDMCS LPDYVTFKSF PGIPLHHIFS AAGDDLLDLI QGL FLFNPC ARITATQALK MKYFSNRPGP TPGCQLPRPN CPVETLKEQS NPALAIKRKR TEALEQGGLP KKLIF UniProtKB: Cyclin-dependent kinase 7 |
-分子 #4: (3R,4R)-4-[[[7-[(phenylmethyl)amino]-3-propan-2-yl-pyrazolo[1,5-a...
| 分子 | 名称: (3R,4R)-4-[[[7-[(phenylmethyl)amino]-3-propan-2-yl-pyrazolo[1,5-a]pyrimidin-5-yl]amino]methyl]piperidin-3-ol タイプ: ligand / ID: 4 / コピー数: 1 / 式: I74 |
|---|---|
| 分子量 | 理論値: 394.513 Da |
| Chemical component information |  ChemComp-I74: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 73 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.9 構成要素:
| ||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | Incubated with 50 uM ICEC0942 for 5 min |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 撮影したグリッド数: 1 / #0 - 実像数: 3571 / #0 - 平均露光時間: 2.0 sec. / #0 - 平均電子線量: 69.0 e/Å2 #0 - 詳細: Movies (69 frames) acquired in 3 x 3 pattern by image shift #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 撮影したグリッド数: 2 / #1 - 実像数: 5302 / #1 - 平均露光時間: 2.0 sec. / #1 - 平均電子線量: 69.0 e/Å2 #1 - 詳細: Movies (70 frames) acquired in 3 x 3 pattern by image shift |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 72886 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Real space refinement in PHENIX; ligand coordinates from PHENIX-OPLS3e refinement |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-7b5o:  PDB-7b5q: |
 ムービー
ムービー コントローラー
コントローラー































 Z
Z Y
Y X
X