+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10059 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nucleosome-CHD4 complex structure (two CHD4 copies) | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Complex / ATPase / chromatin / nucleosome / CHD family / TRANSCRIPTION | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cerebellar granule cell to Purkinje cell synapse / terminal button organization / NuRD complex / regulation of cell fate specification / NGF-stimulated transcription / regulation of stem cell differentiation / ATP-dependent chromatin remodeler activity / regulation of synapse assembly / RNA Polymerase I Transcription Initiation / site of DNA damage ...cerebellar granule cell to Purkinje cell synapse / terminal button organization / NuRD complex / regulation of cell fate specification / NGF-stimulated transcription / regulation of stem cell differentiation / ATP-dependent chromatin remodeler activity / regulation of synapse assembly / RNA Polymerase I Transcription Initiation / site of DNA damage / Transcriptional regulation of brown and beige adipocyte differentiation by EBF2 / Regulation of TP53 Activity through Acetylation / Regulation of PTEN gene transcription / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / transcription coregulator binding / Regulation of endogenous retroelements by KRAB-ZFP proteins / double-strand break repair via homologous recombination / Regulation of endogenous retroelements by Piwi-interacting RNAs (piRNAs) / HDACs deacetylate histones / helicase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / RNA polymerase II transcription regulator complex / histone deacetylase binding / structural constituent of chromatin / transcription corepressor activity / nucleosome / heterochromatin formation / nucleosome assembly / histone binding / Potential therapeutics for SARS / RNA polymerase II-specific DNA-binding transcription factor binding / chromosome, telomeric region / chromatin remodeling / protein heterodimerization activity / negative regulation of gene expression / negative regulation of DNA-templated transcription / centrosome / chromatin binding / chromatin / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / protein-containing complex / ATP hydrolysis activity / DNA binding / zinc ion binding / nucleoplasm / ATP binding / nucleus / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
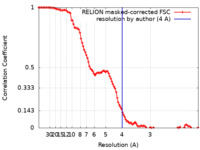
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Farnung L / Ochmann M | |||||||||||||||
| 資金援助 |  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Nucleosome-CHD4 chromatin remodeler structure maps human disease mutations. 著者: Lucas Farnung / Moritz Ochmann / Patrick Cramer /  要旨: Chromatin remodeling plays important roles in gene regulation during development, differentiation and in disease. The chromatin remodeling enzyme CHD4 is a component of the NuRD and ChAHP complexes ...Chromatin remodeling plays important roles in gene regulation during development, differentiation and in disease. The chromatin remodeling enzyme CHD4 is a component of the NuRD and ChAHP complexes that are involved in gene repression. Here, we report the cryo-electron microscopy (cryo-EM) structure of CHD4 engaged with a nucleosome core particle in the presence of the non-hydrolysable ATP analogue AMP-PNP at an overall resolution of 3.1 Å. The ATPase motor of CHD4 binds and distorts nucleosomal DNA at superhelical location (SHL) +2, supporting the 'twist defect' model of chromatin remodeling. CHD4 does not induce unwrapping of terminal DNA, in contrast to its homologue Chd1, which functions in gene activation. Our structure also maps CHD4 mutations that are associated with human cancer or the intellectual disability disorder Sifrim-Hitz-Weiss syndrome. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10059.map.gz emd_10059.map.gz | 7.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10059-v30.xml emd-10059-v30.xml emd-10059.xml emd-10059.xml | 30.9 KB 30.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10059_fsc.xml emd_10059_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10059.png emd_10059.png | 67.5 KB | ||
| マスクデータ |  emd_10059_msk_1.map emd_10059_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10059.cif.gz emd-10059.cif.gz | 7.9 KB | ||
| その他 |  emd_10059_additional_1.map.gz emd_10059_additional_1.map.gz emd_10059_additional_2.map.gz emd_10059_additional_2.map.gz emd_10059_half_map_1.map.gz emd_10059_half_map_1.map.gz emd_10059_half_map_2.map.gz emd_10059_half_map_2.map.gz | 80.4 MB 95.9 MB 80.8 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10059 http://ftp.pdbj.org/pub/emdb/structures/EMD-10059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10059 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10059_validation.pdf.gz emd_10059_validation.pdf.gz | 877.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10059_full_validation.pdf.gz emd_10059_full_validation.pdf.gz | 877.2 KB | 表示 | |
| XML形式データ |  emd_10059_validation.xml.gz emd_10059_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_10059_validation.cif.gz emd_10059_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10059 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10059 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10059 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10059 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ryuMC  6ryrC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10411 (タイトル: Single Particle Cryo-EM Reconstructions of NCP-CHD4 complexes EMPIAR-10411 (タイトル: Single Particle Cryo-EM Reconstructions of NCP-CHD4 complexesData size: 721.2 Data #1: Unaligned multi-frame micrographs of NCP-CHD4 Dataset 1 [micrographs - multiframe] Data #2: Averaged and aligned micrographs Dataset 1 [micrographs - single frame] Data #3: Unaligned multi-frame micrographs of NCP-CHD4 Dataset 2 [micrographs - multiframe] Data #4: Averaged and aligned micrographs NCP-CHD4 Dataset 2 [micrographs - single frame] Data #5: Unaligned multi-frame micrographs of NCP-CHD4 Dataset 3 [micrographs - multiframe] Data #6: Averaged and aligned micrographs Dataset 3 [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10059.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10059.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10059_msk_1.map emd_10059_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
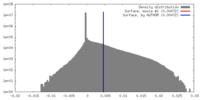
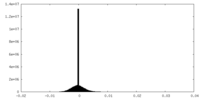
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_10059_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
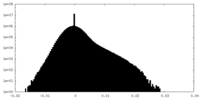
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_10059_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10059_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
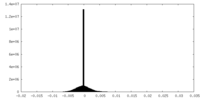
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10059_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
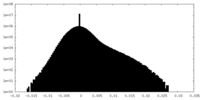
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Nucleosome-CHD4 complex
+超分子 #1: Nucleosome-CHD4 complex
+超分子 #2: Histone
+超分子 #3: DNA
+超分子 #4: Chromodomain-helicase-DNA-binding protein 4
+分子 #1: Histone H3.2
+分子 #2: Histone H4
+分子 #3: Histone H2A type 1
+分子 #4: Histone H2B 1.1
+分子 #7: Chromodomain-helicase-DNA-binding protein 4,CHD4,Chromodomain-hel...
+分子 #5: DNA (149-MER)
+分子 #6: DNA (149-MER)
+分子 #8: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
+分子 #9: MAGNESIUM ION
+分子 #10: ZINC ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)



















































 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)