+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1lxd | ||||||
|---|---|---|---|---|---|---|---|
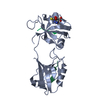
| タイトル | CRYSTAL STRUCTURE OF THE RAS INTERACTING DOMAIN OF RALGDS, A GUANINE NUCLEOTIDE DISSOCIATION STIMULATOR OF RAL PROTEIN | ||||||
 要素 要素 | RALGDSB | ||||||
 キーワード キーワード | PHOSPHORYLATION / RALGDS / RAS BINDING / UBIQUITIN FOLD / CDC25 FAMILY / SIGNAL TRANSDUCTION / CROSS-TALK | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報p38MAPK events / GTPase regulator activity / RAF/MAP kinase cascade / small GTPase-mediated signal transduction / brush border / guanyl-nucleotide exchange factor activity / Ras protein signal transduction / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.4 Å 多波長異常分散 / 解像度: 2.4 Å | ||||||
 データ登録者 データ登録者 | Huang, L. / Weng, X.W. / Hofer, F. / Martin, G.S. / Kim, S.H. | ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 1997 ジャーナル: Nat.Struct.Biol. / 年: 1997タイトル: Three-dimensional structure of the Ras-interacting domain of RalGDS. 著者: Huang, L. / Weng, X. / Hofer, F. / Martin, G.S. / Kim, S.H. #1:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996タイトル: Crystallization and Preliminary Crystallographic Analysis of the Ras Binding Domain of Ralgds, a Guanine Nucleotide Dissociation Stimulator of the Ral Protein 著者: Huang, L. / Jancarik, J. / Kim, S.-H. / Hofer, F. / Martin, G.S. #2:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1994 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1994タイトル: Activated Ras Interacts with the Ral Guanine Nucleotide Dissociation Stimulator 著者: Hofer, F. / Fields, S. / Schneider, C. / Martin, G.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1lxd.cif.gz 1lxd.cif.gz | 59.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1lxd.ent.gz pdb1lxd.ent.gz | 43.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1lxd.json.gz 1lxd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1lxd_validation.pdf.gz 1lxd_validation.pdf.gz | 447.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1lxd_full_validation.pdf.gz 1lxd_full_validation.pdf.gz | 453 KB | 表示 | |
| XML形式データ |  1lxd_validation.xml.gz 1lxd_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  1lxd_validation.cif.gz 1lxd_validation.cif.gz | 13.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lx/1lxd https://data.pdbj.org/pub/pdb/validation_reports/lx/1lxd ftp://data.pdbj.org/pub/pdb/validation_reports/lx/1lxd ftp://data.pdbj.org/pub/pdb/validation_reports/lx/1lxd | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (-0.219734, -0.826478, -0.518316), ベクター: |
- 要素
要素
| #1: タンパク質 | 分子量: 11350.771 Da / 分子数: 2 / 断片: C-TERMINAL DOMAIN WHICH BINDS TO ACTIVE RAS 変異: N-TERMINAL GS INHERITED FROM THE LINKER SEQUENCE OF THE CLONING VECTOR 由来タイプ: 組換発現 / 由来: (組換発現)  遺伝子 (発現宿主): C-TERMINAL DOMAIN OF RALGDS FUSED TO GLUTATHIONINE S TRANSFERASE 発現宿主:  #2: 水 | ChemComp-HOH / | Has protein modification | Y | 配列の詳細 | TERMINAL RESIDUE LYS 100 IN THE PDB FILE IS LYS 864 IN THE RAT RALGDS SEQUENCE. | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.8 Å3/Da / 溶媒含有率: 30 % 解説: THIS STRUCTURE WAS SOLVED USING THE MAD DATA ON THE SELENOMETHIONINE MUTANT OF THE PROTEIN AT X4A NSLS. BUT THE STRUCTURE WAS REFINED AGAINST THE NATIVE DATA ABOVE. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 8.5 詳細: PROTEIN WAS CRYSTALLIZED FROM 20% PEG 8000, 0.1 M TRIS PH 8.5, AND 0.2 M CALCIUM ACETATE. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X12B / 波長: 1.008 / ビームライン: X12B / 波長: 1.008 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1996年11月10日 / 詳細: MIRRORS |
| 放射 | モノクロメーター: SI(111) / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.008 Å / 相対比: 1 |
| 反射 | 解像度: 2.4→10 Å / Num. obs: 6485 / % possible obs: 99.3 % / Observed criterion σ(I): 0 / 冗長度: 4 % / Biso Wilson estimate: 20 Å2 / Rsym value: 0.09 / Net I/σ(I): 16.7 |
| 反射 シェル | 解像度: 2.4→2.48 Å / 冗長度: 4 % / Mean I/σ(I) obs: 5.92 / Rsym value: 0.251 / % possible all: 99.5 |
| 反射 | *PLUS Rmerge(I) obs: 0.09 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 2.4→6 Å / Rfactor Rfree error: 0.04 / Data cutoff high absF: 15000 / Data cutoff low absF: 0 / 交差検証法: THROUGHOUT / σ(F): 2 / 詳細: H19A.PEP IS THE PEPTIDE BOND FIL 多波長異常分散 / 解像度: 2.4→6 Å / Rfactor Rfree error: 0.04 / Data cutoff high absF: 15000 / Data cutoff low absF: 0 / 交差検証法: THROUGHOUT / σ(F): 2 / 詳細: H19A.PEP IS THE PEPTIDE BOND FIL
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 15 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati d res low obs: 6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.4→6 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: NO RESTRAINTS. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.4→2.48 Å / Rfactor Rfree error: 0.045 / Total num. of bins used: 10
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.85 / 分類: refinement X-PLOR / バージョン: 3.85 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rfree: 0.31 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj




