登録情報 データベース : EMDB / ID : EMD-9575タイトル Cryo-EM structure of zika virus complexed with Fab C10 at pH 8.0 複合体 : zika virus complexed with C10 Fab at pH 8.0複合体 : zika virusタンパク質・ペプチド : structural protein Eタンパク質・ペプチド : strutural protein M複合体 : C10 Fabタンパク質・ペプチド : C10 IgG heavy chain variable regionタンパク質・ペプチド : C10 IgG light chain variable regionリガンド : 2-acetamido-2-deoxy-beta-D-glucopyranose / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 / Homo sapiens (ヒト)手法 / / 解像度 : 4.0 Å Zhang S / Kostyuchenko V 資金援助 Organization Grant number 国 Ministry of Education Tier 3 grant MOE2012-T3-1-008 National Research Foundation Investigatorship award NRF-NRFI2016-01 National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) AI100625 and AI 107731
ジャーナル : Nat Commun / 年 : 2016タイトル : Neutralization mechanism of a highly potent antibody against Zika virus.著者 : Shuijun Zhang / Victor A Kostyuchenko / Thiam-Seng Ng / Xin-Ni Lim / Justin S G Ooi / Sebastian Lambert / Ter Yong Tan / Douglas G Widman / Jian Shi / Ralph S Baric / Shee-Mei Lok / 要旨 : The rapid spread of Zika virus (ZIKV), which causes microcephaly and Guillain-Barré syndrome, signals an urgency to identify therapeutics. Recent efforts to rescreen dengue virus human antibodies ... The rapid spread of Zika virus (ZIKV), which causes microcephaly and Guillain-Barré syndrome, signals an urgency to identify therapeutics. Recent efforts to rescreen dengue virus human antibodies for ZIKV cross-neutralization activity showed antibody C10 as one of the most potent. To investigate the ability of the antibody to block fusion, we determined the cryoEM structures of the C10-ZIKV complex at pH levels mimicking the extracellular (pH8.0), early (pH6.5) and late endosomal (pH5.0) environments. The 4.0 Å resolution pH8.0 complex structure shows that the antibody binds to E proteins residues at the intra-dimer interface, and the virus quaternary structure-dependent inter-dimer and inter-raft interfaces. At pH6.5, antibody C10 locks all virus surface E proteins, and at pH5.0, it locks the E protein raft structure, suggesting that it prevents the structural rearrangement of the E proteins during the fusion event-a vital step for infection. This suggests antibody C10 could be a good therapeutic candidate. 履歴 登録 2016年10月26日 - ヘッダ(付随情報) 公開 2016年11月9日 - マップ公開 2016年11月30日 - 更新 2024年11月13日 - 現状 2024年11月13日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
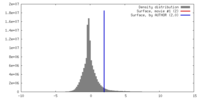
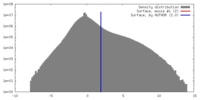
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Zika virus (ジカ熱ウイルス) /
Zika virus (ジカ熱ウイルス) /  Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 シンガポール,
シンガポール,  米国, 3件
米国, 3件  引用
引用 ジャーナル: Nat Commun / 年: 2016
ジャーナル: Nat Commun / 年: 2016

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_9575.map.gz
emd_9575.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-9575-v30.xml
emd-9575-v30.xml emd-9575.xml
emd-9575.xml EMDBヘッダ
EMDBヘッダ emd_9575.png
emd_9575.png emd-9575.cif.gz
emd-9575.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-9575
http://ftp.pdbj.org/pub/emdb/structures/EMD-9575 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9575
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9575 emd_9575_validation.pdf.gz
emd_9575_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_9575_full_validation.pdf.gz
emd_9575_full_validation.pdf.gz emd_9575_validation.xml.gz
emd_9575_validation.xml.gz emd_9575_validation.cif.gz
emd_9575_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9575
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9575 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9575
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9575 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_9575.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_9575.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Zika virus (ジカ熱ウイルス)
Zika virus (ジカ熱ウイルス)
 Zika virus (ジカ熱ウイルス) / 株: Mr 766
Zika virus (ジカ熱ウイルス) / 株: Mr 766
 Zika virus (ジカ熱ウイルス) / 株: Mr 766
Zika virus (ジカ熱ウイルス) / 株: Mr 766 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)