+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9251 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CENP-A nucleosome bound by two copies of CENP-C(CD) and two copies CENP-N(NT) | |||||||||
 マップデータ マップデータ | CENP-A nucleosome bound by two copies of CENP-C(CD) and two copies CENP-N(NT) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | centromere / CENP-A / kinetochore / nucleosome / NUCLEAR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報spindle attachment to meiosis I kinetochore / centromeric DNA binding / CENP-A containing chromatin assembly / protein localization to chromosome, centromeric region / kinetochore assembly / attachment of mitotic spindle microtubules to kinetochore / inner kinetochore / condensed chromosome, centromeric region / establishment of mitotic spindle orientation / mitotic cytokinesis ...spindle attachment to meiosis I kinetochore / centromeric DNA binding / CENP-A containing chromatin assembly / protein localization to chromosome, centromeric region / kinetochore assembly / attachment of mitotic spindle microtubules to kinetochore / inner kinetochore / condensed chromosome, centromeric region / establishment of mitotic spindle orientation / mitotic cytokinesis / chromosome, centromeric region / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / pericentric heterochromatin / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / nucleosomal DNA binding / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / Resolution of Sister Chromatid Cohesion / telomere organization / Meiotic synapsis / RNA Polymerase I Promoter Opening / Assembly of the ORC complex at the origin of replication / SUMOylation of chromatin organization proteins / Regulation of endogenous retroelements by the Human Silencing Hub (HUSH) complex / DNA methylation / Condensation of Prophase Chromosomes / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Regulation of endogenous retroelements by KRAB-ZFP proteins / Defective pyroptosis / chromosome segregation / Regulation of endogenous retroelements by Piwi-interacting RNAs (piRNAs) / HDACs deacetylate histones / Nonhomologous End-Joining (NHEJ) / RHO GTPases Activate Formins / RNA Polymerase I Promoter Escape / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / NoRC negatively regulates rRNA expression / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / G2/M DNA damage checkpoint / HDMs demethylate histones / B-WICH complex positively regulates rRNA expression / DNA Damage/Telomere Stress Induced Senescence / heterochromatin formation / PKMTs methylate histone lysines / Metalloprotease DUBs / Meiotic recombination / kinetochore / Pre-NOTCH Transcription and Translation / RMTs methylate histone arginines / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / Separation of Sister Chromatids / UCH proteinases / nucleosome / nucleosome assembly / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / chromatin organization / mitotic cell cycle / RUNX1 regulates transcription of genes involved in differentiation of HSCs / HATs acetylate histones / Processing of DNA double-strand break ends / midbody / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression / chromosome, telomeric region / nuclear body / Ub-specific processing proteases / protein heterodimerization activity / Amyloid fiber formation / negative regulation of cell population proliferation / cell division / chromatin binding / protein-containing complex / DNA binding / RNA binding / extracellular exosome / extracellular region / nucleoplasm / identical protein binding / membrane / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
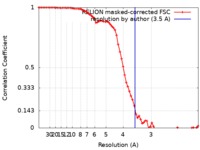
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Allu PK / Black BE | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Curr Biol / 年: 2019 ジャーナル: Curr Biol / 年: 2019タイトル: Structure of the Human Core Centromeric Nucleosome Complex. 著者: Praveen Kumar Allu / Jennine M Dawicki-McKenna / Trevor Van Eeuwen / Moriya Slavin / Merav Braitbard / Chen Xu / Nir Kalisman / Kenji Murakami / Ben E Black /   要旨: Centromeric nucleosomes are at the interface of the chromosome and the kinetochore that connects to spindle microtubules in mitosis. The core centromeric nucleosome complex (CCNC) harbors the ...Centromeric nucleosomes are at the interface of the chromosome and the kinetochore that connects to spindle microtubules in mitosis. The core centromeric nucleosome complex (CCNC) harbors the histone H3 variant, CENP-A, and its binding proteins, CENP-C (through its central domain; CD) and CENP-N (through its N-terminal domain; NT). CENP-C can engage nucleosomes through two domains: the CD and the CENP-C motif (CM). CENP-C is part of the CCNC by virtue of its high specificity for CENP-A nucleosomes and ability to stabilize CENP-A at the centromere. CENP-C is thought to engage a neighboring nucleosome, either one containing conventional H3 or CENP-A, and a crystal structure of a nucleosome complex containing two copies of CENP-C was reported. Recent structures containing a single copy of CENP-N bound to the CENP-A nucleosome in the absence of CENP-C were reported. Here, we find that one copy of CENP-N is lost for every two copies of CENP-C on centromeric chromatin just prior to kinetochore formation. We present the structures of symmetric and asymmetric forms of the CCNC that vary in CENP-N stoichiometry. Our structures explain how the central domain of CENP-C achieves its high specificity for CENP-A nucleosomes and how CENP-C and CENP-N sandwich the histone H4 tail. The natural centromeric DNA path in our structures corresponds to symmetric surfaces for CCNC assembly, deviating from what is observed in prior structures using artificial sequences. At mitosis, we propose that CCNC asymmetry accommodates its asymmetric connections at the chromosome/kinetochore interface. VIDEO ABSTRACT. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9251.map.gz emd_9251.map.gz | 4.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9251-v30.xml emd-9251-v30.xml emd-9251.xml emd-9251.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9251_fsc.xml emd_9251_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9251.png emd_9251.png | 232.5 KB | ||
| Filedesc metadata |  emd-9251.cif.gz emd-9251.cif.gz | 6.8 KB | ||
| その他 |  emd_9251_additional.map.gz emd_9251_additional.map.gz | 23.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9251 http://ftp.pdbj.org/pub/emdb/structures/EMD-9251 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9251 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9251 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9251_validation.pdf.gz emd_9251_validation.pdf.gz | 430.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9251_full_validation.pdf.gz emd_9251_full_validation.pdf.gz | 429.7 KB | 表示 | |
| XML形式データ |  emd_9251_validation.xml.gz emd_9251_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_9251_validation.cif.gz emd_9251_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9251 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9251 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9251 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9251 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9251.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9251.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CENP-A nucleosome bound by two copies of CENP-C(CD) and two copies CENP-N(NT) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
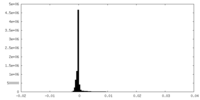
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: CENP-A nucleosome bound by two copies of CENP-C(CD)...
| ファイル | emd_9251_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CENP-A nucleosome bound by two copies of CENP-C(CD) and two copies CENP-N(NT) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
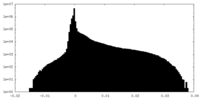
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CENP-A chromatin complex bound with CENP-C and CENP-N of CCAN kin...
| 全体 | 名称: CENP-A chromatin complex bound with CENP-C and CENP-N of CCAN kinetochore components |
|---|---|
| 要素 |
|
-超分子 #1: CENP-A chromatin complex bound with CENP-C and CENP-N of CCAN kin...
| 超分子 | 名称: CENP-A chromatin complex bound with CENP-C and CENP-N of CCAN kinetochore components タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#8 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 276 KDa |
-分子 #1: Histone H3-like centromeric protein A
| 分子 | 名称: Histone H3-like centromeric protein A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.993037 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HQHSRRRQGW LKEIRKLQKS THLLIRKLPF SRLAREICVK FTRGVDFNWQ AQALLALQEA AEAFLVHLFE DAYLLTLHAG RVTLFPKDV QLARRIRGLE EGL UniProtKB: Histone H3-like centromeric protein A |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.604521 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KGLGKGGAKR HRKVLRDNIQ GITKPAIRRL ARRGGVKRIS GLIYEETRGV LKVFLENVIR DAVTYTEHAK RKTVTAMDVV YALKRQGRT LYGFG UniProtKB: Histone H4 |
-分子 #3: Histone H2A type 1-C
| 分子 | 名称: Histone H2A type 1-C / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.494393 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KAKSRSSRAG LQFPVGRVHR LLRKGNYAER VGAGAPVYLA AVLEYLTAEI LELAGNAARD NKKTRIIPRH LQLAIRNDEE LNKLLGRVT IAQGGVLPNI QSVLLP UniProtKB: Histone H2A type 1-C |
-分子 #4: Histone H2B type 2-F
| 分子 | 名称: Histone H2B type 2-F / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.249723 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RKESYSVYVY KVLKQVHPDT GISSKAMGIM NSFVNDIFER IAGEASRLAH YNKRSTITSR EIQTAVRLLL PGELAKHAVS EGTKAVTKY TSS UniProtKB: Histone H2B type 2-F |
-分子 #7: Centromere protein C
| 分子 | 名称: Centromere protein C / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 2.508834 KDa |
| 配列 | 文字列: TKSRRISRRP SDWWVVKSEE UniProtKB: Centromere protein C |
-分子 #8: Centromere protein N
| 分子 | 名称: Centromere protein N / タイプ: protein_or_peptide / ID: 8 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.052885 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDETVAEFIK RTILKIPMNE LTTILKAWDF LSENQLQTVN FRQRKESVVQ HLIHLCEEKR ASISDAALLD IIYMQFHQHQ KVWDVFQMS KGPGEDVDLF DMKQFKNSFK KILQRALKNV TVSFRETEEN AVWIRIAWGT QYTKPNQYKP TYVVYYSQTP Y AFTSSSML ...文字列: MDETVAEFIK RTILKIPMNE LTTILKAWDF LSENQLQTVN FRQRKESVVQ HLIHLCEEKR ASISDAALLD IIYMQFHQHQ KVWDVFQMS KGPGEDVDLF DMKQFKNSFK KILQRALKNV TVSFRETEEN AVWIRIAWGT QYTKPNQYKP TYVVYYSQTP Y AFTSSSML RRNTPLLGQA LTIASKHHQI VKMDLRSRYL DSLKAIVFKQ YNQT UniProtKB: Centromere protein N |
-分子 #5: DNA (147-MER)
| 分子 | 名称: DNA (147-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.141918 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DA)(DA)(DA)(DT)(DA)(DT)(DC) (DC)(DA)(DC)(DC)(DT)(DG)(DC)(DA)(DG)(DA) (DT)(DT)(DC)(DT)(DA)(DC)(DC)(DA)(DA) (DA)(DA)(DG)(DT)(DG)(DT)(DA)(DT)(DT)(DT) (DG) (DG)(DA)(DA)(DA)(DC) ...文字列: (DA)(DT)(DC)(DA)(DA)(DA)(DT)(DA)(DT)(DC) (DC)(DA)(DC)(DC)(DT)(DG)(DC)(DA)(DG)(DA) (DT)(DT)(DC)(DT)(DA)(DC)(DC)(DA)(DA) (DA)(DA)(DG)(DT)(DG)(DT)(DA)(DT)(DT)(DT) (DG) (DG)(DA)(DA)(DA)(DC)(DT)(DG)(DC) (DT)(DC)(DC)(DA)(DT)(DC)(DA)(DA)(DA)(DA) (DG)(DG) (DC)(DA)(DT)(DG)(DT)(DT)(DC) (DA)(DG)(DC)(DT)(DC)(DT)(DG)(DT)(DG)(DA) (DG)(DT)(DG) (DA)(DA)(DA)(DC)(DT)(DC) (DC)(DA)(DT)(DC)(DA)(DT)(DC)(DA)(DC)(DA) (DA)(DA)(DG)(DA) (DA)(DT)(DA)(DT)(DT) (DC)(DT)(DG)(DA)(DG)(DA)(DA)(DT)(DG)(DC) (DT)(DT)(DC)(DC)(DG) (DT)(DT)(DT)(DG) (DC)(DC)(DT)(DT)(DT)(DT)(DA)(DT)(DA)(DT) (DG)(DA)(DA)(DC)(DT)(DT) (DC)(DC)(DT) (DC)(DG)(DA)(DT) |
-分子 #6: DNA (147-MER)
| 分子 | 名称: DNA (147-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.582188 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DG)(DA)(DA)(DG) (DT)(DT)(DC)(DA)(DT)(DA)(DT)(DA)(DA)(DA) (DA)(DG)(DG)(DC)(DA)(DA)(DA)(DC)(DG) (DG)(DA)(DA)(DG)(DC)(DA)(DT)(DT)(DC)(DT) (DC) (DA)(DG)(DA)(DA)(DT) ...文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DG)(DA)(DA)(DG) (DT)(DT)(DC)(DA)(DT)(DA)(DT)(DA)(DA)(DA) (DA)(DG)(DG)(DC)(DA)(DA)(DA)(DC)(DG) (DG)(DA)(DA)(DG)(DC)(DA)(DT)(DT)(DC)(DT) (DC) (DA)(DG)(DA)(DA)(DT)(DA)(DT)(DT) (DC)(DT)(DT)(DT)(DG)(DT)(DG)(DA)(DT)(DG) (DA)(DT) (DG)(DG)(DA)(DG)(DT)(DT)(DT) (DC)(DA)(DC)(DT)(DC)(DA)(DC)(DA)(DG)(DA) (DG)(DC)(DT) (DG)(DA)(DA)(DC)(DA)(DT) (DG)(DC)(DC)(DT)(DT)(DT)(DT)(DG)(DA)(DT) (DG)(DG)(DA)(DG) (DC)(DA)(DG)(DT)(DT) (DT)(DC)(DC)(DA)(DA)(DA)(DT)(DA)(DC)(DA) (DC)(DT)(DT)(DT)(DT) (DG)(DG)(DT)(DA) (DG)(DA)(DA)(DT)(DC)(DT)(DG)(DC)(DA)(DG) (DG)(DT)(DG)(DG)(DA)(DT) (DA)(DT)(DT) (DT)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10 mM HEPES, 50 mN NaCl, 1 mM EDTA, 1mM DTT |
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 300.0 kPa / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC / 詳細: Blot for 8 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 2 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)