+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8616 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 70S ribosome bound with cognate ternary complex base-paired to A site codon (Structure II) | |||||||||||||||
 マップデータ マップデータ | 70S ribosome bound with cognate ternary complex base-paired to A site codon (Structure II): filtered map from Frealign (FFILT setting in Frealign = T), used for model building after B-factor sharpening. This volume not B-factor sharpened. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Ribosome / EF-Tu / tRNA | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報guanyl-nucleotide exchange factor complex / negative regulation of cytoplasmic translational initiation / guanosine tetraphosphate binding / stringent response / ornithine decarboxylase inhibitor activity / transcription antitermination factor activity, RNA binding / misfolded RNA binding / Group I intron splicing / RNA folding / translational elongation ...guanyl-nucleotide exchange factor complex / negative regulation of cytoplasmic translational initiation / guanosine tetraphosphate binding / stringent response / ornithine decarboxylase inhibitor activity / transcription antitermination factor activity, RNA binding / misfolded RNA binding / Group I intron splicing / RNA folding / translational elongation / transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis / negative regulation of cytoplasmic translation / translation elongation factor activity / four-way junction DNA binding / translational termination / DnaA-L2 complex / translation repressor activity / negative regulation of DNA-templated DNA replication initiation / negative regulation of translational initiation / regulation of mRNA stability / mRNA regulatory element binding translation repressor activity / ribosome assembly / assembly of large subunit precursor of preribosome / positive regulation of RNA splicing / transcription elongation factor complex / cytosolic ribosome assembly / regulation of DNA-templated transcription elongation / DNA endonuclease activity / response to reactive oxygen species / transcription antitermination / regulation of cell growth / translational initiation / DNA-templated transcription termination / maintenance of translational fidelity / response to radiation / mRNA 5'-UTR binding / ribosomal small subunit biogenesis / small ribosomal subunit rRNA binding / large ribosomal subunit / ribosome biogenesis / ribosome binding / regulation of translation / ribosomal small subunit assembly / small ribosomal subunit / 5S rRNA binding / large ribosomal subunit rRNA binding / transferase activity / cytosolic small ribosomal subunit / ribosomal large subunit assembly / cytoplasmic translation / cytosolic large ribosomal subunit / tRNA binding / molecular adaptor activity / negative regulation of translation / rRNA binding / ribosome / structural constituent of ribosome / translation / response to antibiotic / negative regulation of DNA-templated transcription / GTPase activity / mRNA binding / GTP binding / DNA binding / RNA binding / zinc ion binding / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||||||||
 データ登録者 データ登録者 | Loveland AB / Demo G | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Ensemble cryo-EM elucidates the mechanism of translation fidelity. 著者: Anna B Loveland / Gabriel Demo / Nikolaus Grigorieff / Andrei A Korostelev /  要旨: Gene translation depends on accurate decoding of mRNA, the structural mechanism of which remains poorly understood. Ribosomes decode mRNA codons by selecting cognate aminoacyl-tRNAs delivered by ...Gene translation depends on accurate decoding of mRNA, the structural mechanism of which remains poorly understood. Ribosomes decode mRNA codons by selecting cognate aminoacyl-tRNAs delivered by elongation factor Tu (EF-Tu). Here we present high-resolution structural ensembles of ribosomes with cognate or near-cognate aminoacyl-tRNAs delivered by EF-Tu. Both cognate and near-cognate tRNA anticodons explore the aminoacyl-tRNA-binding site (A site) of an open 30S subunit, while inactive EF-Tu is separated from the 50S subunit. A transient conformation of decoding-centre nucleotide G530 stabilizes the cognate codon-anticodon helix, initiating step-wise 'latching' of the decoding centre. The resulting closure of the 30S subunit docks EF-Tu at the sarcin-ricin loop of the 50S subunit, activating EF-Tu for GTP hydrolysis and enabling accommodation of the aminoacyl-tRNA. By contrast, near-cognate complexes fail to induce the G530 latch, thus favouring open 30S pre-accommodation intermediates with inactive EF-Tu. This work reveals long-sought structural differences between the pre-accommodation of cognate and near-cognate tRNAs that elucidate the mechanism of accurate decoding. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8616.map.gz emd_8616.map.gz | 100.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8616-v30.xml emd-8616-v30.xml emd-8616.xml emd-8616.xml | 83.8 KB 83.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8616.png emd_8616.png | 155.8 KB | ||
| Filedesc metadata |  emd-8616.cif.gz emd-8616.cif.gz | 15 KB | ||
| その他 |  emd_8616_half_map_1.map.gz emd_8616_half_map_1.map.gz emd_8616_half_map_2.map.gz emd_8616_half_map_2.map.gz | 101.7 MB 101.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8616 http://ftp.pdbj.org/pub/emdb/structures/EMD-8616 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8616 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8616 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8616_validation.pdf.gz emd_8616_validation.pdf.gz | 912.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8616_full_validation.pdf.gz emd_8616_full_validation.pdf.gz | 911.6 KB | 表示 | |
| XML形式データ |  emd_8616_validation.xml.gz emd_8616_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_8616_validation.cif.gz emd_8616_validation.cif.gz | 22 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8616 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8616 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8616 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8616 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5uylMC  8615C  8617C  8618C  8619C  8620C  5uykC  5uymC  5uynC  5uypC  5uyqC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8616.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8616.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 70S ribosome bound with cognate ternary complex base-paired to A site codon (Structure II): filtered map from Frealign (FFILT setting in Frealign = T), used for model building after B-factor sharpening. This volume not B-factor sharpened. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
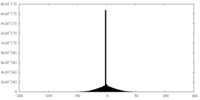
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: 70S ribosome bound with cognate ternary complex base-paired...
| ファイル | emd_8616_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 70S ribosome bound with cognate ternary complex base-paired to A site codon (Structure II): half map (1) suitable for resolution calculation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
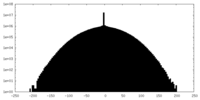
| 密度ヒストグラム |
-ハーフマップ: 70S ribosome bound with cognate ternary complex base-paired...
| ファイル | emd_8616_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 70S ribosome bound with cognate ternary complex base-paired to A site codon (Structure II): half map (2) suitable for resolution calculation | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
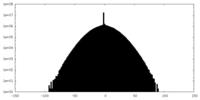
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : 70S ribosome bound with cognate ternary complex not base-paired t...
+超分子 #1: 70S ribosome bound with cognate ternary complex not base-paired t...
+分子 #1: 50S ribosomal protein L2
+分子 #2: 50S ribosomal protein L3
+分子 #3: 50S ribosomal protein L4
+分子 #4: 50S ribosomal protein L5
+分子 #5: 50S ribosomal protein L6
+分子 #6: 50S ribosomal protein L9
+分子 #7: 50S ribosomal protein L10
+分子 #8: 50S ribosomal protein L11
+分子 #9: 50S ribosomal protein L13
+分子 #10: 50S ribosomal protein L14
+分子 #11: 50S ribosomal protein L15
+分子 #12: 50S ribosomal protein L16
+分子 #13: 50S ribosomal protein L17
+分子 #14: 50S ribosomal protein L18
+分子 #15: 50S ribosomal protein L19
+分子 #16: 50S ribosomal protein L20
+分子 #17: 50S ribosomal protein L21
+分子 #18: 50S ribosomal protein L22
+分子 #19: 50S ribosomal protein L23
+分子 #20: 50S ribosomal protein L24
+分子 #21: 50S ribosomal protein L25
+分子 #22: 50S ribosomal protein L27
+分子 #23: 50S ribosomal protein L28
+分子 #24: 50S ribosomal protein L29
+分子 #25: 50S ribosomal protein L30
+分子 #26: 50S ribosomal protein L31
+分子 #27: 50S ribosomal protein L32
+分子 #28: 50S ribosomal protein L33
+分子 #29: 50S ribosomal protein L34
+分子 #30: 50S ribosomal protein L35
+分子 #31: 50S ribosomal protein L36
+分子 #32: 30S ribosomal protein S2
+分子 #33: 30S ribosomal protein S3
+分子 #34: 30S ribosomal protein S4
+分子 #35: 30S ribosomal protein S5
+分子 #36: 30S ribosomal protein S6
+分子 #37: 30S ribosomal protein S7
+分子 #38: 30S ribosomal protein S8
+分子 #39: 30S ribosomal protein S9
+分子 #40: 30S ribosomal protein S10
+分子 #41: 30S ribosomal protein S11
+分子 #42: 30S ribosomal protein S12
+分子 #43: 30S ribosomal protein S13
+分子 #44: 30S ribosomal protein S14
+分子 #45: 30S ribosomal protein S15
+分子 #46: 30S ribosomal protein S16
+分子 #47: 30S ribosomal protein S17
+分子 #48: 30S ribosomal protein S18
+分子 #49: 30S ribosomal protein S19
+分子 #50: 30S ribosomal protein S20
+分子 #51: 30S ribosomal protein S21
+分子 #52: 50S ribosomal protein L1
+分子 #59: Elongation factor Tu 2
+分子 #53: 16S ribosomal RNA
+分子 #54: 23S ribosomal RNA
+分子 #55: 5S ribosomal RNA
+分子 #56: tRNAfMet
+分子 #57: mRNA
+分子 #58: tRNAPhe
+分子 #60: N-FORMYLMETHIONINE
+分子 #61: PHENYLALANINE
+分子 #62: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 275 K / 装置: GATAN CRYOPLUNGE 3 詳細: 2 uL of complex was applied to each grid. After a 10-second incubation, the grids were blotted for 2 to 4 seconds.. |
| 詳細 | 250 nM 50S, 250 nM 30S, 1.25 micromolar mRNA, 500 nM fMet-tRNAfMet, 1 micromolar EF-T, 500 micromolar GDPCP, 1 micromolar Phe-tRNAPhe |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 2 / 実像数: 3928 / 平均露光時間: 0.4 sec. / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 60976 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60976 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
|---|---|
| 得られたモデル |  PDB-5uyl: |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)