+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7a5v | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of a human gamma-aminobutyric acid receptor, the GABA(A)R-beta3 homopentamer, in complex with histamine and megabody Mb25 in lipid nanodisc | |||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / Pentameric ligand-gated ion channel / Neurotrasmitter receptor / GABA(A) receptor | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to histamine / GABA receptor activation / GABA-A receptor activity / GABA-gated chloride ion channel activity / GABA-A receptor complex / inhibitory synapse assembly / synaptic transmission, GABAergic / gamma-aminobutyric acid signaling pathway / postsynaptic specialization membrane / neurotransmitter receptor activity ...cellular response to histamine / GABA receptor activation / GABA-A receptor activity / GABA-gated chloride ion channel activity / GABA-A receptor complex / inhibitory synapse assembly / synaptic transmission, GABAergic / gamma-aminobutyric acid signaling pathway / postsynaptic specialization membrane / neurotransmitter receptor activity / roof of mouth development / Signaling by ERBB4 / chloride channel complex / transmembrane transporter complex / chloride transmembrane transport / cytoplasmic vesicle membrane / postsynaptic membrane / dendritic spine / neuron projection / synapse / signal transduction / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.7 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Nakane, T. / Kotecha, A. / Sente, A. / Yamashita, K. / McMullan, G. / Masiulis, S. / Brown, P.M.G.E. / Grigoras, I.T. / Malinauskaite, L. / Malinauskas, T. ...Nakane, T. / Kotecha, A. / Sente, A. / Yamashita, K. / McMullan, G. / Masiulis, S. / Brown, P.M.G.E. / Grigoras, I.T. / Malinauskaite, L. / Malinauskas, T. / Miehling, J. / Yu, L. / Karia, D. / Pechnikova, E.V. / de Jong, E. / Keizer, J. / Bischoff, M. / McCormack, J. / Tiemeijer, P. / Hardwick, S.W. / Chirgadze, D.Y. / Murshudov, G. / Aricescu, A.R. / Scheres, S.H.W. | |||||||||||||||||||||
| 資金援助 |  英国, 6件 英国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Single-particle cryo-EM at atomic resolution. 著者: Takanori Nakane / Abhay Kotecha / Andrija Sente / Greg McMullan / Simonas Masiulis / Patricia M G E Brown / Ioana T Grigoras / Lina Malinauskaite / Tomas Malinauskas / Jonas Miehling / Tomasz ...著者: Takanori Nakane / Abhay Kotecha / Andrija Sente / Greg McMullan / Simonas Masiulis / Patricia M G E Brown / Ioana T Grigoras / Lina Malinauskaite / Tomas Malinauskas / Jonas Miehling / Tomasz Uchański / Lingbo Yu / Dimple Karia / Evgeniya V Pechnikova / Erwin de Jong / Jeroen Keizer / Maarten Bischoff / Jamie McCormack / Peter Tiemeijer / Steven W Hardwick / Dimitri Y Chirgadze / Garib Murshudov / A Radu Aricescu / Sjors H W Scheres /    要旨: The three-dimensional positions of atoms in protein molecules define their structure and their roles in biological processes. The more precisely atomic coordinates are determined, the more chemical ...The three-dimensional positions of atoms in protein molecules define their structure and their roles in biological processes. The more precisely atomic coordinates are determined, the more chemical information can be derived and the more mechanistic insights into protein function may be inferred. Electron cryo-microscopy (cryo-EM) single-particle analysis has yielded protein structures with increasing levels of detail in recent years. However, it has proved difficult to obtain cryo-EM reconstructions with sufficient resolution to visualize individual atoms in proteins. Here we use a new electron source, energy filter and camera to obtain a 1.7 Å resolution cryo-EM reconstruction for a human membrane protein, the β3 GABA receptor homopentamer. Such maps allow a detailed understanding of small-molecule coordination, visualization of solvent molecules and alternative conformations for multiple amino acids, and unambiguous building of ordered acidic side chains and glycans. Applied to mouse apoferritin, our strategy led to a 1.22 Å resolution reconstruction that offers a genuine atomic-resolution view of a protein molecule using single-particle cryo-EM. Moreover, the scattering potential from many hydrogen atoms can be visualized in difference maps, allowing a direct analysis of hydrogen-bonding networks. Our technological advances, combined with further approaches to accelerate data acquisition and improve sample quality, provide a route towards routine application of cryo-EM in high-throughput screening of small molecule modulators and structure-based drug discovery. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7a5v.cif.gz 7a5v.cif.gz | 128.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7a5v.ent.gz pdb7a5v.ent.gz | 96.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7a5v.json.gz 7a5v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7a5v_validation.pdf.gz 7a5v_validation.pdf.gz | 1.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7a5v_full_validation.pdf.gz 7a5v_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  7a5v_validation.xml.gz 7a5v_validation.xml.gz | 34.3 KB | 表示 | |
| CIF形式データ |  7a5v_validation.cif.gz 7a5v_validation.cif.gz | 50.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/a5/7a5v https://data.pdbj.org/pub/pdb/validation_reports/a5/7a5v ftp://data.pdbj.org/pub/pdb/validation_reports/a5/7a5v ftp://data.pdbj.org/pub/pdb/validation_reports/a5/7a5v | HTTPS FTP |
-関連構造データ
| 関連構造データ |  11657MC  7a4mC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10500 (タイトル: High resolution structure of GABAAR beta3 homopentamer EMPIAR-10500 (タイトル: High resolution structure of GABAAR beta3 homopentamerData size: 13.0 TB Data #1: CFEG, Falcon4, with 5 eV filter [micrographs - multiframe] Data #2: CFEG, Falcon4, with 5 eV filter, without objective aperture [micrographs - multiframe] Data #3: XFEG, Falcon3, without filter [micrographs - multiframe] Data #4: XFEG, Falcon4, with filter but retracted slit [micrographs - multiframe] Data #5: XFEG, Falcon4, with 3 eV filter [micrographs - multiframe] Data #6: XFEG, Falcon4, with 5 eV filter [micrographs - multiframe] Data #7: XFEG, Falcon4, with 3 eV filter [micrographs - multiframe] Data #8: XFEG, K3, without filter [micrographs - multiframe]) |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-タンパク質 / 抗体 , 2種, 2分子 AO
| #1: タンパク質 | 分子量: 45744.285 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GABRB3 / 細胞株 (発現宿主): HEK293S / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GABRB3 / 細胞株 (発現宿主): HEK293S / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P28472 Homo sapiens (ヒト) / 参照: UniProt: P28472 |
|---|---|
| #2: 抗体 | 分子量: 56300.629 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
-糖 , 3種, 3分子 
| #3: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |
|---|---|
| #4: 多糖 | alpha-D-mannopyranose-(1-3)-[alpha-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-4)-2-acetamido-2- ...alpha-D-mannopyranose-(1-3)-[alpha-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |
| #7: 糖 | ChemComp-NAG / |
-非ポリマー , 6種, 212分子 

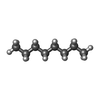
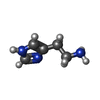







| #5: 化合物 | ChemComp-R16 / | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| #6: 化合物 | ChemComp-D10 / #8: 化合物 | ChemComp-OCT / | #9: 化合物 | ChemComp-HSM / | #10: 化合物 | #11: 水 | ChemComp-HOH / | |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.485 MDa / 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||
| 緩衝液 | pH: 7.6 | ||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: UltrAuFoil R1.2/1.3 | ||||||||||||||||||||||||
| 急速凍結 | 装置: LEICA PLUNGER / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 287 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 165000 X / 最大 デフォーカス(公称値): 1100 nm / 最小 デフォーカス(公称値): 300 nm / Cs: 2.7 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 40 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) |
| 画像スキャン | 横: 4096 / 縦: 4096 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0270 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C5 (5回回転対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 1.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 371693 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 1.7→292.21 Å / Cor.coef. Fo:Fc: 0.862 / SU B: 0.591 / SU ML: 0.017 / ESU R: 0.009 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 50.129 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 4231 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj










