#1: タンパク質 A B C D Sulfofructosephosphate aldolase / SFP aldolase詳細 配列 配列領域 機能 構造部位 (4) 変異 (80) 構造検証 (4)
分子量 : 34076.262 Da
/ 分子数 : 4
/ 由来タイプ : 組換発現
由来 : (組換発現)
Salmonella enterica (サルモネラ菌)遺伝子: A0N81_18250, A3M31_18775, A6870_09715, A6898_11780, A7749_21790, A7F95_15550, AIO56_15225, AIY46_16340, AL463_07200, AUD13_20420, AWT52_20455, AWU43_13980, AXB23_12525, B1Q59_20525, B5654_ ... 遺伝子 : A0N81_18250, A3M31_18775, A6870_09715, A6898_11780, A7749_21790, A7F95_15550, AIO56_15225, AIY46_16340, AL463_07200, AUD13_20420, AWT52_20455, AWU43_13980, AXB23_12525, B1Q59_20525, B5654_18495, B9864_14245, BH399_15915, BM489_16210, BUX38_13710, BWB44_15835, BZG86_17325, BZG97_21310, C0X54_15110, CF411_19890, CGY76_15895, CIU94_12215, CJT17_15765, CKB50_07315, CKU74_14590, COP74_11865, COQ67_11805, CQW68_17310, CRH85_14725, CRQ37_15620, CRX97_18560, CS198_12645, CSK90_20940, CTB03_19360, D0C46_16695, D1239_15905, D3346_03620, D3P91_16440, D5403_16535, D5563_18085, D5K09_17370, D5K19_15290, D5S75_07410, D5T72_18210, D5U49_16110, D5U61_18460, D5U63_11355, D6093_17370, D6272_19205, D6518_20345, D6570_13930, D6969_14660, D6E28_04120, D6E86_17625, D6F20_16795, D6F49_14185, D6G02_17665, D6G26_10200, D6G47_17665, D6G76_19295, D6H24_18045, D6H57_18255, D6I51_13955, D6I58_08830, D6J18_10675, D6N21_14905, D6N55_08625, D6O25_17540, D6O30_15095, D6Y46_16705, D7B84_19925, D7B88_17660, D7C05_16595, D7C06_21680, D7C84_14020, D7C85_15860, D7D21_15750, D7D22_05990, D7D38_09665, D7D43_21660, D7E22_19080, D7E26_17595, D7E90_22935, D7F69_17605, D7G05_16290, D7G28_16865, D7G59_21240, D7J34_06690, D7L19_20755, D7M32_23435, D7T23_13960, D7U23_11360, D7U29_12545, D7V14_12170, D7V56_03055, D7W03_19080, D7W42_15665, D7W92_17045, D7X02_18505, D8I75_22320, D8I93_09135, D8N07_15105, D8N11_16775, D8N46_13960, D8N55_16780, D8O41_20680, D8P26_14535, D8P48_20440, D8P50_21965, D8P66_11165, D8T80_18610, D8U57_08880, D8V04_12990, D8V52_15680, D8W17_18845, D8W37_13540, D8X82_11100, D8X94_09915, D9788_20490, D9789_20795, D9813_16860, D9893_11870, D9904_16025, D9908_21180, D9A34_21035, D9A73_12625, D9B25_17115, D9B94_19645, D9B97_15535, D9N56_15950, D9N82_18885, D9R37_17245, D9R54_15605, DKH31_16885, DKT79_16205, DLG27_11245, DLG46_06205, DLG52_07510, DLG63_07605, DLG68_10135, DLO13_17295, DLO43_18780, DM517_15195, DM524_16185, DM771_11020, DO307_21625, DP889_19510, DPH90_12840, DPI33_15970, DQT99_17410, DS006_04045, DSC90_17425, DT233_14295, DTC77_09285, DTD68_12010, DTS56_10715, DV697_20955, DWE28_16300, DXG53_11555, DXJ54_22650, DXN62_09570, DXS05_10875, EA021_14685, EA079_18825, EA089_19020, EA474_08655, EA495_19590, EA530_18665, EA547_20545, EA580_19185, EA608_19155, EA613_20370, EA865_08835, EA919_10115, EAA74_11055, EAB27_13270, EAB35_16100, EAB61_09075, EAB95_19485, EAC03_16205, EAC50_16505, EAC62_17350, EAC94_18900, EAD37_15045, EAD40_09145, EAD57_13665, EAD76_19515, EAM93_17780, EAN15_16450, EAN28_13585, EAP40_16925, EAP69_20610, EAP86_19400, EAT69_18110, EAT75_16760, EAT82_14950, EAU16_09940, EAU32_20705, EAU35_15685, EAU46_12835, EAU53_10110, EAU58_12765, EAU70_20970, EAV62_11145, EAW74_11570, EAW95_17755, EAX13_03905, EAX14_14635, EAX36_11700, EAX53_13770, EBB31_16095, EBF85_18205, EBG44_16225, EBG45_13980, EBG75_07135, EBH58_15795, EBH87_22355, EBI58_18035, EE513_15450, EF325_19645, EF388_12305, EF393_20830, EF628_09690, EF991_17315, EFC63_19880, EFC67_20365, EFE59_16535, EFG85_09970, EFH54_15625, EFH94_15335, EFJ16_17715, EFY29_17600, EFZ26_04800, EFZ60_09705, EG093_16475, EG164_13955, EG208_16115, EG454_09425, EG623_11865, EG641_09825, EG692_19510, EG762_13995, EG767_06865, EG904_07125, EG952_08925, EG958_15370, EGA35_15790, EGA57_09435, EGC01_19260, EGC99_17865, EGD06_09555, EGD07_07640, EGD31_09160, EGD39_14760, EGI61_15585, EGS29_19525, EGY78_10120, EGZ16_20780, EGZ23_10235, EH065_16465, EH070_09515, EH342_16890, EH378_03350, EH386_15285, EH444_09295, EHA32_16515, EHA97_05130, EHG07_19400, EHG48_13385, EHG76_05255, EHN34_10355, EHN57_18120, EHN63_15685, EHN69_20070, EHN71_15015, EHN74_20955, EHN75_15295, EHO38_16865, EHT01_15035, EHT24_15825, EHU01_19845, EHU13_18495, EHZ75_17125, EI217_12345, EID62_19165, EIM38_09355, EIN80_15985, EIP55_14465, EIQ89_18750, EIR18_16870, EIR57_14645, EIR68_20810, EIR69_10290, EIR74_09395, EIR86_11165, EIS08_13390, EIS13_04610, EIU76_04060, EIU90_06585, EIV01_12260, EIW14_04360, EIW18_14780, F0673_17300, F0Y85_06995, F1H08_17590, F2162_17330, F2262_17185, F2D15_19045, F3368_19600, F3E97_16225, F3Z27_11460, F6G43_17665, F6H70_17780, F7365_11585, F7450_19930, F7M71_12365, F8658_14000, FE26_19770, FJR52_18110, FLX20_17775, FQR50_10080, FR099_15765, FRH21_16835, FRK18_12280, FY665_16255, FZ154_17610, FZ547_18070, GB762_17280, GC435_07645, GC879_16555, GCA81_13885, GCH85_20920, GCR52_14595, IX01_17020, R929_17375プラスミド : pET28a
/ 発現宿主 :
Escherichia coli BL21(DE3) (大腸菌)参照 :
,
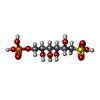
#2: 化合物 A -301C -301ChemComp-U8W / (2~{S},3~{S},4~{R})-2,3,4,5-tetrakis(oxidanyl)-6-phosphonooxy-hexane-1-sulfonic acid 詳細 外部データベース 構造部位
分子量 : 326.215 Da
/ 分子数 : 2
/ 由来タイプ : 合成
/ 式 : C
6 H
15 O
11 PS
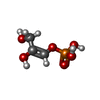
#3: 化合物 B -301D -301ChemComp-U8Z / [(~{E})-2,3-bis(oxidanyl)prop-1-enyl] dihydrogen phosphate 詳細 外部データベース 構造部位
分子量 : 170.058 Da
/ 分子数 : 2
/ 由来タイプ : 合成
/ 式 : C
3 H
7 O
6 P
#4: 水 ChemComp-HOH / water 詳細 外部データベース
分子量 : 18.015 Da
/ 分子数 : 1000
/ 由来タイプ : 天然
/ 式 : H
2 O
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Salmonella enterica (サルモネラ菌)
Salmonella enterica (サルモネラ菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.5 Å
分子置換 / 解像度: 1.5 Å  データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: Acs Cent.Sci. / 年: 2021
ジャーナル: Acs Cent.Sci. / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7ne2.cif.gz
7ne2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7ne2.ent.gz
pdb7ne2.ent.gz PDB形式
PDB形式 7ne2.json.gz
7ne2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7ne2_validation.pdf.gz
7ne2_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7ne2_full_validation.pdf.gz
7ne2_full_validation.pdf.gz 7ne2_validation.xml.gz
7ne2_validation.xml.gz 7ne2_validation.cif.gz
7ne2_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ne/7ne2
https://data.pdbj.org/pub/pdb/validation_reports/ne/7ne2 ftp://data.pdbj.org/pub/pdb/validation_reports/ne/7ne2
ftp://data.pdbj.org/pub/pdb/validation_reports/ne/7ne2 リンク
リンク 集合体
集合体
 要素
要素 Salmonella enterica (サルモネラ菌)
Salmonella enterica (サルモネラ菌)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.9763 Å
/ ビームライン: I03 / 波長: 0.9763 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj