+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7542 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the nvTRPM2 channel in complex with Ca2+ | |||||||||
 マップデータ マップデータ | Structure of the nvTRPM2 channel in complex with Ca2+ | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ligand-gated sodium channel activity / ADP-ribose diphosphatase activity / ligand-gated calcium channel activity / monoatomic cation transmembrane transport / plasma membrane => GO:0005886 / sodium ion transmembrane transport / monoatomic cation channel activity / calcium ion transmembrane transport / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Nematostella vectensis (イソギンチャク) Nematostella vectensis (イソギンチャク) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.07 Å | |||||||||
 データ登録者 データ登録者 | Zhang Z / Toth B / Szollosi A / Chen J / Csanady L | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: Structure of a TRPM2 channel in complex with Ca explains unique gating regulation. 著者: Zhe Zhang / Balázs Tóth / Andras Szollosi / Jue Chen / László Csanády /   要旨: Transient receptor potential melastatin 2 (TRPM2) is a Ca-permeable cation channel required for immune cell activation, insulin secretion, and body heat control. TRPM2 is activated by cytosolic Ca, ...Transient receptor potential melastatin 2 (TRPM2) is a Ca-permeable cation channel required for immune cell activation, insulin secretion, and body heat control. TRPM2 is activated by cytosolic Ca, phosphatidyl-inositol-4,5-bisphosphate and ADP ribose. Here, we present the ~3 Å resolution electron cryo-microscopic structure of TRPM2 from , 63% similar in sequence to human TRPM2, in the Ca-bound closed state. Compared to other TRPM channels, TRPM2 exhibits unique structural features that correlate with its function. The pore is larger and more negatively charged, consistent with its high Ca selectivity and larger conductance. The intracellular Ca binding sites are connected to the pore and cytosol, explaining the unusual dependence of TRPM2 activity on intra- and extracellular Ca. In addition, the absence of a post-filter motif is likely the cause of the rapid inactivation of human TRPM2. Together, our cryo-EM and electrophysiology studies provide a molecular understanding of the unique gating mechanism of TRPM2. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7542.map.gz emd_7542.map.gz | 202.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7542-v30.xml emd-7542-v30.xml emd-7542.xml emd-7542.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7542.png emd_7542.png | 201.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7542 http://ftp.pdbj.org/pub/emdb/structures/EMD-7542 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7542 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7542 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7542_validation.pdf.gz emd_7542_validation.pdf.gz | 537.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7542_full_validation.pdf.gz emd_7542_full_validation.pdf.gz | 537 KB | 表示 | |
| XML形式データ |  emd_7542_validation.xml.gz emd_7542_validation.xml.gz | 7 KB | 表示 | |
| CIF形式データ |  emd_7542_validation.cif.gz emd_7542_validation.cif.gz | 8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7542 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7542 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7542 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7542 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7542.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7542.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the nvTRPM2 channel in complex with Ca2+ | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
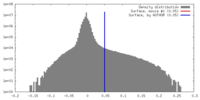
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : nvTRPM2 tetramer
| 全体 | 名称: nvTRPM2 tetramer |
|---|---|
| 要素 |
|
-超分子 #1: nvTRPM2 tetramer
| 超分子 | 名称: nvTRPM2 tetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Nematostella vectensis (イソギンチャク) Nematostella vectensis (イソギンチャク) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Predicted protein
| 分子 | 名称: Predicted protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Nematostella vectensis (イソギンチャク) Nematostella vectensis (イソギンチャク) |
| 分子量 | 理論値: 176.663438 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGKDSFTPLY DGGDSSHVHL NKFGSNQLSQ SKKSWIARNF SRRECIRFVP KSHDVSRCKC GRPRERHSQQ ALESGQGSEE WNVASCTTK HPTNAYGEID FEGYGGQKRA PYLRMSHDTD ANLVITLMLK RWNLEIPNLV ISVTGGAKSF VLKPRLREMF R RGLIKAAK ...文字列: MGKDSFTPLY DGGDSSHVHL NKFGSNQLSQ SKKSWIARNF SRRECIRFVP KSHDVSRCKC GRPRERHSQQ ALESGQGSEE WNVASCTTK HPTNAYGEID FEGYGGQKRA PYLRMSHDTD ANLVITLMLK RWNLEIPNLV ISVTGGAKSF VLKPRLREMF R RGLIKAAK TTGAWIITGG TNTGVMKHVG EAVKEQQLMF GSDTQVNVIG IATWGIVDKQ SDLISEKNGK YPALYSMEPT PG HQGAMLD PNHSHFFLVD DGTEGKYGVE IGMRSRIEEA IMKVKTDSRS EAGSIGVPVV LLVLEGGPNT VATMYELIKK KVP AVVIDG SGRAASVVGF AYNHTIKRNV DGQTINVIDP QYEDEVRAKV VEVFGAKGAD KTYSMIKDVL EDEKMISVYS LDGE ISQDI DLAILKALLK ANRSSPVAQL NLALAWNRID LAKSDIFTEE QQWTTETLSA AMLTALLDDK AEFAELFLQN GLSMR EFLS LDILCKLYAE VPGNTTIKPL LQKEMGKRQV KTIDMDVVGE VIEELMGDMF ESYYRKDGHY FGELASYAEG LVLKNR KSS KDLLANINRI DPLPTPYLDV FLWAVLCNRR ELARVLWEAG REPMAAALMA SRLLKRMASR AQEDNTITDI SSDLYDH AR LFEERAVGVL DECFNENETL SQTLLVRELD HYSRMTALEL AVSAESQDFI AHTSCQVLLT RLWMGTMAMN TRWWKVLV C LYLPVLIFPI IYFVPDEQHE RQAAEREHQK SLNQKSSKVK SHKEKNDAPV VPVYRSKEEK AVSNDEEARV GTENEEEDF QLEDYIPEIR EDDSMEVIMR NKKLGFCDRI MHFYSAPFSK FVGNVVGYLA FIFLYAYVVL FNFPRFDPAK TLGGIHPTEI VLYFWVFTI LIEEIRQLAA KPPKYIKDKV SVYFSDTWNF VDIFSLTVFI IAIILRFFTN SRIFTASRII LSLDIIFFIV R SLQIFSVN RLLGPKLVMI QKMMQDLAQF IIILAVFTIA YGIALHAVMF PSPGIYARNN TWVTITSVVQ YPYWQMYGEL FL DEIQGEK PKEFGEVDPD GRWLSPLLLA IYMVFTNILL LNLLIAIFNY TFERVQEDSD KVWKFQRYDL VQEYHSRPVF APP LVLLGH ILIFIRWVWR MCRCGHPPRG STMKIGLSPA EMEQMDNWEF QAAEMYIHQQ QQKNSGTLEE RVRALGDRVD CINS QLNRV LDSMSGTRAH ALTDGNGLEG GHDSEGRLAR MEVELSSNSE SLQKILALLQ QQPPVKGQAA VPIQLTLLHY KARSS PYPG STAKRFAVQD NMVDWQVPFP DYKPVNYTAP VVLANPVWAD KDLMAMSPRP ELPYNQMDHT CNVNRVSYNG TYVVKD GLP LNPMGRTGMQ GRGLLGRFGP NHAADPVVTR WKRTSAGVML QGGKKVLEFV AIQRKDNNQW AIPGGMVEPG QLVTQAL KA EFGEEAMAKL NVSQEEKERI AKQIERLFQQ GQEIYKGYVD DPRNTDNAWM ETVAVNFHDD KGDLFGDITL QAGDDAAA V RWQRVSGNIP LYASHVSILE KVAKMRDAAF SNSLEVLFQ |
-分子 #3: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 3 / コピー数: 4 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #4: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(tri...
| 分子 | 名称: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(trimethylammonio)ethyl phosphate タイプ: ligand / ID: 4 / コピー数: 56 / 式: POV |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-POV: |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 4 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #6: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 6 / コピー数: 3 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 1550 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.7 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6co7: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)