+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7086 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | active dimer | |||||||||
 マップデータ マップデータ | Primary consensus dimer map in C2. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PIKK / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of type B pancreatic cell development / positive regulation of cytoplasmic translational initiation / positive regulation of pentose-phosphate shunt / RNA polymerase III type 1 promoter sequence-specific DNA binding / RNA polymerase III type 2 promoter sequence-specific DNA binding / T-helper 1 cell lineage commitment / Activation of the mRNA upon binding of the cap-binding complex and eIFs, and subsequent binding to 43S / regulation of locomotor rhythm / positive regulation of wound healing, spreading of epidermal cells / eukaryotic initiation factor 4E binding ...regulation of type B pancreatic cell development / positive regulation of cytoplasmic translational initiation / positive regulation of pentose-phosphate shunt / RNA polymerase III type 1 promoter sequence-specific DNA binding / RNA polymerase III type 2 promoter sequence-specific DNA binding / T-helper 1 cell lineage commitment / Activation of the mRNA upon binding of the cap-binding complex and eIFs, and subsequent binding to 43S / regulation of locomotor rhythm / positive regulation of wound healing, spreading of epidermal cells / eukaryotic initiation factor 4E binding / cellular response to leucine starvation / regulation of membrane permeability / TFIIIC-class transcription factor complex binding / heart valve morphogenesis / negative regulation of lysosome organization / RNA polymerase III type 3 promoter sequence-specific DNA binding / TORC2 complex / TORC1 complex / positive regulation of transcription of nucleolar large rRNA by RNA polymerase I / regulation of autophagosome assembly / calcineurin-NFAT signaling cascade / nucleus localization / TORC1 signaling / voluntary musculoskeletal movement / positive regulation of odontoblast differentiation / regulation of osteoclast differentiation / positive regulation of keratinocyte migration / cellular response to L-leucine / MTOR signalling / Amino acids regulate mTORC1 / cellular response to nutrient / energy reserve metabolic process / Energy dependent regulation of mTOR by LKB1-AMPK / negative regulation of cell size / ruffle organization / protein serine/threonine kinase inhibitor activity / positive regulation of osteoclast differentiation / cellular response to osmotic stress / enzyme-substrate adaptor activity / negative regulation of protein localization to nucleus / anoikis / cardiac muscle cell development / negative regulation of cold-induced thermogenesis / positive regulation of transcription by RNA polymerase III / negative regulation of calcineurin-NFAT signaling cascade / regulation of myelination / regulation of cell size / Macroautophagy / positive regulation of oligodendrocyte differentiation / small GTPase-mediated signal transduction / negative regulation of macroautophagy / positive regulation of actin filament polymerization / lysosome organization / positive regulation of myotube differentiation / protein kinase activator activity / behavioral response to pain / oligodendrocyte differentiation / Constitutive Signaling by AKT1 E17K in Cancer / mTORC1-mediated signalling / germ cell development / CD28 dependent PI3K/Akt signaling / cellular response to nutrient levels / positive regulation of phosphoprotein phosphatase activity / social behavior / HSF1-dependent transactivation / positive regulation of TOR signaling / TOR signaling / neuronal action potential / positive regulation of translational initiation / response to amino acid / positive regulation of G1/S transition of mitotic cell cycle / regulation of macroautophagy / endomembrane system / 'de novo' pyrimidine nucleobase biosynthetic process / positive regulation of lamellipodium assembly / positive regulation of epithelial to mesenchymal transition / positive regulation of lipid biosynthetic process / heart morphogenesis / cardiac muscle contraction / regulation of cellular response to heat / translation repressor activity / negative regulation of translational initiation / positive regulation of stress fiber assembly / 14-3-3 protein binding / positive regulation of TORC1 signaling / positive regulation of endothelial cell proliferation / cytoskeleton organization / translation initiation factor binding / T cell costimulation / cellular response to amino acid starvation / phagocytic vesicle / positive regulation of glycolytic process / cellular response to starvation / negative regulation of autophagy / positive regulation of mitotic cell cycle / protein serine/threonine kinase activator activity / response to nutrient levels / response to nutrient / post-embryonic development / VEGFR2 mediated vascular permeability 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Pavletich NP / Yang H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Mechanisms of mTORC1 activation by RHEB and inhibition by PRAS40. 著者: Haijuan Yang / Xiaolu Jiang / Buren Li / Hyo J Yang / Meredith Miller / Angela Yang / Ankita Dhar / Nikola P Pavletich /  要旨: The mechanistic target of rapamycin complex 1 (mTORC1) controls cell growth and metabolism in response to nutrients, energy levels, and growth factors. It contains the atypical kinase mTOR and the ...The mechanistic target of rapamycin complex 1 (mTORC1) controls cell growth and metabolism in response to nutrients, energy levels, and growth factors. It contains the atypical kinase mTOR and the RAPTOR subunit that binds to the Tor signalling sequence (TOS) motif of substrates and regulators. mTORC1 is activated by the small GTPase RHEB (Ras homologue enriched in brain) and inhibited by PRAS40. Here we present the 3.0 ångström cryo-electron microscopy structure of mTORC1 and the 3.4 ångström structure of activated RHEB-mTORC1. RHEB binds to mTOR distally from the kinase active site, yet causes a global conformational change that allosterically realigns active-site residues, accelerating catalysis. Cancer-associated hyperactivating mutations map to structural elements that maintain the inactive state, and we provide biochemical evidence that they mimic RHEB relieving auto-inhibition. We also present crystal structures of RAPTOR-TOS motif complexes that define the determinants of TOS recognition, of an mTOR FKBP12-rapamycin-binding (FRB) domain-substrate complex that establishes a second substrate-recruitment mechanism, and of a truncated mTOR-PRAS40 complex that reveals PRAS40 inhibits both substrate-recruitment sites. These findings help explain how mTORC1 selects its substrates, how its kinase activity is controlled, and how it is activated by cancer-associated mutations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7086.map.gz emd_7086.map.gz | 186.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7086-v30.xml emd-7086-v30.xml emd-7086.xml emd-7086.xml | 26 KB 26 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7086.png emd_7086.png | 62.2 KB | ||
| Filedesc metadata |  emd-7086.cif.gz emd-7086.cif.gz | 8.8 KB | ||
| その他 |  emd_7086_additional_1.map.gz emd_7086_additional_1.map.gz emd_7086_additional_2.map.gz emd_7086_additional_2.map.gz emd_7086_additional_3.map.gz emd_7086_additional_3.map.gz | 186.8 MB 186.8 MB 186.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7086 http://ftp.pdbj.org/pub/emdb/structures/EMD-7086 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7086 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7086 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7086_validation.pdf.gz emd_7086_validation.pdf.gz | 581.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7086_full_validation.pdf.gz emd_7086_full_validation.pdf.gz | 581.3 KB | 表示 | |
| XML形式データ |  emd_7086_validation.xml.gz emd_7086_validation.xml.gz | 6.7 KB | 表示 | |
| CIF形式データ |  emd_7086_validation.cif.gz emd_7086_validation.cif.gz | 7.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7086 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7086 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7086 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7086 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6bcuMC  7087C  5wbhC  5wbiC  5wbjC  5wbkC  5wblC  5wbuC  5wbyC  6bcxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7086.map.gz / 形式: CCP4 / 大きさ: 199.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7086.map.gz / 形式: CCP4 / 大きさ: 199.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary consensus dimer map in C2. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3306 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Focused 3D refinement of monomer part2 (mask 2)...
| ファイル | emd_7086_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
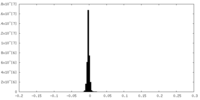
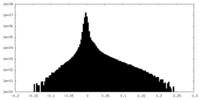
| 注釈 | Focused 3D refinement of monomer part2 (mask 2) used to reconstruct the 3.4 angstroms resolution C2 dimer structure factors with the REFMAC5 composite map option for model refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Focused 3D refinement of monomer part1 (mask 1)...
| ファイル | emd_7086_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
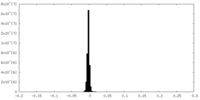
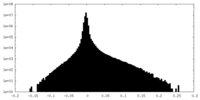
| 注釈 | Focused 3D refinement of monomer part1 (mask 1) used to reconstruct the 3.4 angstroms resolution C2 dimer structure factors with the REFMAC5 composite map option for model refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Focused 3D refinement of monomer part3 (mask 3)...
| ファイル | emd_7086_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused 3D refinement of monomer part3 (mask 3) used to reconstruct the 3.4 angstroms resolution C2 dimer structure factors with the REFMAC5 composite map option for model refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : active complex
| 全体 | 名称: active complex |
|---|---|
| 要素 |
|
-超分子 #1: active complex
| 超分子 | 名称: active complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Serine/threonine-protein kinase mTOR,Serine/threonine-protein kin...
| 分子 | 名称: Serine/threonine-protein kinase mTOR,Serine/threonine-protein kinase mTOR タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 287.399125 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MLGTGPAAAT TAATTS(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)SRNEET(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) ...文字列: MLGTGPAAAT TAATTS(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)SRNEET(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)EMSQE E(UNK)TRFYDQLN HHIFELVSSS DANERKGGIL AIASLIGVEG GNATRI GRF ANYLRNLLPS NDPVVMEMAS KAIGRLAMAG DTFTAEYVEF EVKRALEWLG ADRNEGRRHA AVLVLRELAI SVPTFFF QQ VQPFFDNIFV AVWDPKQAIR EGAVAALRAC LILTTQREPK EMQKPQWYRH TFEEAEKGFD ETLAKEKGMN RDDRIHGA L LILNELVRIS SMEGERLREE MEEITQQQLV HDKYCKDLMG FGTKPRHITP FTSFQAVQPQ QSNALVGLLG YSSHQGLMG FGTSPSPAKS T(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)RNS KNSLIQMTIL NLLPRLA AF RPSAFTDTQY LQDTMNHVLS CVKKEKERTA AFQALGLLSV AVRSEFKVYL PRVLDIIRAA LPPKDFAHKR QKAMQVDA T VFTCISMLAR AMGPGIQQDI KELLEPMLAV GLSPALTAVL YDLSRQIPQL KKDIQDGLLK MLSLVLM(UNK)KP LRHPG MPKG LAHQLASPGL TTLPEAS(UNK)VG SITLALRTLG SFEFEGHSLT QFVRHCADHF LNSEHKEIRM EAARTCSRLL TP SIHLISG HAHVVSQTAV QVVADVLSKL LVVGITDPDP DIRYCVLASL DERFDAHLAQ AENLQALFVA LNDQVFEIRE LAI CTVGRL SSMNPAFVMP FLRKMLIQIL TELEHSGIGR IKEQSARMLG HLVSNAPRLI RPYMEPILKA LILKLKDPDP DPNP GVINN VLATIGELAQ VSGLEMRKWV DELFIIIMDM LQDSSLLAKR QVALWTLGQL VASTGYVVEP YRKYPTLLEV LLNFL KTEQ NQGTRREAIR VLGLLGALDP YKHKVNIGMI DQSRDASAVS LSESKSSQDS SDYSTSEMLV NMGNLPLDEF YPAVSM VAL MRIFRDQSLS HHHTMVVQAI TFIFKSLGLK CVQFLPQVMP TFLNVIRVCD GAIREFLFQQ LGMLVSFVKS HIRPYMD EI VTLMREFWVM NTSIQSTIIL LIEQIVVALG GEFKLYLPQL IPHMLRVFMH DNSPGRIVSI KLLAAIQLFG ANLDDYLH L LLPPIVKLFD APEAPLPSRK AALETVDRLT ESLDFTDYAS RIIHPIVRTL DQSPELRSTA MDTLSSLVFQ LGKKYQIFI PMVNKVLVRH RINHQRYDVL ICRIVKGYTL ADEEEDPLIY QHRMLRSGQG DALASGPVET GPMKKLHVST INLQKAWGAA RRVSKDDWL EWLRRLSLEL LKDSSSPSLR SCWALAQAYN PMARDLFNAA FVSCWSELNE DQQDELIRSI ELALTSQDIA E VTQTLLNL AEFMEHSDKG PLPLRDDNGI VLLGERAAKC RAYAKALHYK ELEFQKGPTP AILESLISIN NKLQQPEAAA GV LEYAMKH FGELEIQATW YEKLHEWEDA LVAYDKKMDT NKDDPELMLG RMRCLEALGE WGQLHQQCCE KWTLVNDETQ AKM ARMAAA AAWGLGQWDS MEEYTCMIPR DTHDGAFYRA VLALHQDLFS LAQQCIDKAR DLLDAELTAM AGESYSRAYG AMVS CHMLS ELEEVIQYKL VPERREIIRQ IWWERLQGCQ RIVEDWQKIL MVRSLVVSPH EDMRTWLKYA SLCGKSGRLA LAHKT LVLL LGVDPSRQLD HPLPTVHPQV TYAYMKNMWK SARKIDAFQH MQHFVQTMQQ QAQHAIATED QQHKQELHKL MARCFL KLG EWQLNLQGIN ESTIPKVLQY YSAATEHDRS WYKAWHAWAV MNFEAVLHYK HQNQARDEKK KLRHASGANI TNATTAA TT AATATTTAST EGSNSESEAE STENSPTPSP LQKKVTEDLS KTLLMYTVPA VQGFFRSISL SRGNNLQDTL RVLTLWFD Y GHWPDVNEAL VEGVKAIQID TWLQVIPQLI ARIDTPRPLV GRLIHQLLTD IGRYHPQALI YPLTVASKST TTARHNAAN KILKNMCEHS NTLVQQAMMV SEELIRVAIL WHEMWHEGLE EASRLYFGER NVKGMFEVLE PLHAMMERGP QTLKETSFNQ AYGRDLMEA QEWCRKYMKS GNVKDLTQAW DLYYHVFRRI SKQLPQLTSL ELQYVSPKLL MCRDLELAVP GTYDPNQPII R IQSIAPSL QVITSKQRPR KLTLMGSNGH EFVFLLKGHE DLRQDERVMQ LFGLVNTLLA NDPTSLRKNL SIQRYAVIPL ST NSGLIGW VPHCDTLHAL IRDYREKKKI LLNIEHRIML RMAPDYDHLT LMQKVEVFEH AVNNTAGDDL AKLLWLKSPS SEV WFDRRT NYTRSLAVMS MVGYILGLGD RHPSNLMLDR LSGKILHIDF GDCFEVAMTR EKFPEKIPFR LTRMLTNAME VTGL DGNYR ITCHTVMEVL REHKDSVMAV LEAFVYDPLL NWRLMDTNTK GNKRSRTRTD SYSAGQSVEI LDGVELGEPA HKKTG TTVP ESIHSFIGDG LVKPEALNKK AIQIINRVRD KLTGRDFSHD DTLDVPTQVE LLIKQATSHE NLCQCYIGWC PFW UniProtKB: Serine/threonine-protein kinase mTOR |
-分子 #2: Target of rapamycin complex subunit LST8
| 分子 | 名称: Target of rapamycin complex subunit LST8 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 35.91009 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MNTSPGTVGS DPVILATAGY DHTVRFWQAH SGICTRTVQH QDSQVNALEV TPDRSMIAAA GYQHIRMYDL NSNNPNPIIS YDGVNKNIA SVGFHEDGRW MYTGGEDCTA RIWDLRSRNL QCQRIFQVNA PINCVCLHPN QAELIVGDQS GAIHIWDLKT D HNEQLIPE ...文字列: MNTSPGTVGS DPVILATAGY DHTVRFWQAH SGICTRTVQH QDSQVNALEV TPDRSMIAAA GYQHIRMYDL NSNNPNPIIS YDGVNKNIA SVGFHEDGRW MYTGGEDCTA RIWDLRSRNL QCQRIFQVNA PINCVCLHPN QAELIVGDQS GAIHIWDLKT D HNEQLIPE PEVSITSAHI DPDASYMAAV NSTGNCYVWN LTGGIGDEVT QLIPKTKIPA HTRYALQCRF SPDSTLLATC SA DQTCKIW RTSNFSLMTE LSIKSGNPGE SSRGWMWGCA FSGDSQYIVT ASSDNLARLW CVETGEIKRE YGGHQKAVVC LAF NDSVLG UniProtKB: Target of rapamycin complex subunit LST8 |
-分子 #3: Regulatory-associated protein of mTOR,Regulatory-associated prote...
| 分子 | 名称: Regulatory-associated protein of mTOR,Regulatory-associated protein of mTOR タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 150.197 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKE SEMLQSPLLG LGEEDEADLT DWNLPLAFMK KRHCEKIEGS KSLAQSWRMK DRMKTVSVAL VLCLNVGVDP PDVVKTTPC ARLECWIDPL SMGPQKALET IGANLQKQYE NWQPRARYKQ SLDPTVDEVK KLCTSLRRNA KEERVLFHYN G HGVPRPTV ...文字列: MDYKDDDDKE SEMLQSPLLG LGEEDEADLT DWNLPLAFMK KRHCEKIEGS KSLAQSWRMK DRMKTVSVAL VLCLNVGVDP PDVVKTTPC ARLECWIDPL SMGPQKALET IGANLQKQYE NWQPRARYKQ SLDPTVDEVK KLCTSLRRNA KEERVLFHYN G HGVPRPTV NGEVWVFNKN YTQYIPLSIY DLQTWMGSPS IFVYDCSNAG LIVKSFKQFA LQREQELEVA AINPNHPLAQ MP LPPSMKN CIQLAACEAT ELLPMIPDLP ADLFTSCLTT PIKIALRWFC MQKCVSLVPG VTLDLIEKIP GRLNDRRTPL GEL NWIFTA ITDTIAWNVL PRDLFQKLFR QDLLVASLFR NFLLAERIMR SYNCTPVSSP RLPPTYMHAM WQAWDLAVDI CLSQ LPTII EEGTAFRHSP FFAEQLTAFQ VWLTMGVENR NPPEQLPIVL QVLLSQVHRL RALDLLGRFL DLGPWAVSLA LSVGI FPYV LKLLQSSARE LRPLLVFIWA KILAVDSSCQ ADLVKDNGHK YFLSVLADPY MPAEHRTMTA FILAVIVNSY HTGQEA CLQ GNLIAICLEQ LNDPHPLLRQ WVAICLGRIW QNFDSARWCG VRDSAHEKLY SLLSDPIPEV RCAAVFALGT FVGNSAE RT DHSTTIDHNV AMMLAQLVSD GSPMVRKELV VALSHLVVQY ESNFCTVALQ FIEEEKNYAL PSPATTEGGS LTPVRDSP C TPRLRSVSSY GNIRAVATAR SLNKSLQNLS LTEESGGAVA FSPGNLSTSS SASSTLGSPE NEEHILSFET IDKMRRASS YSSLNSLIGV SFNSVYTQIW RVLLHLAADP YPEVSDVAMK VLNSIAYKAT VNARPQRVLD TSSLTQSAPA SPTNKGVHIH QAGGSPPAS STSSSSLTND VAKQPVSRDL PSGRPGTTGP AGAQYTPHSH QFPRTRKMFD KGPEQTADDA DDAAGHKSFI S ATVQTGFC DWSARYFAQP VMKIPEEHDL ESQIRKEREW RFLRNSRVRR QAQQVIQKGI TRLDDQIFLN RNPGVPSVVK FH PFTPCIA VADKDSICFW DWEKGEKLDY FHNGNPRYTR VTAMEYLNGQ DCSLLLTATD DGAIRVWKNF ADLEKNPEMV TAW QGLSDM LPTTRGAGMV VDWEQETGLL MSSGDVRIVR IWDTDREMKV QDIPTGADSC VTSLSCDSHR SLIVAGLGDG SIRV YDRRM ALSECRVMTY REHTAWVVKA SLQKRPDGHI VSVSVNGDVR IFDPRMPESV NVLQIVKGLT ALDIHPQADL IACGS VNQF TAIYNSSGEL INNIKYYDGF MGQRVGAISC LAFHPHWPHL AVGSNDYYIS VYSVEKRVR UniProtKB: Regulatory-associated protein of mTOR, Regulatory-associated protein of mTOR |
-分子 #4: Eukaryotic translation initiation factor 4E-binding protein 1
| 分子 | 名称: Eukaryotic translation initiation factor 4E-binding protein 1 タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.951344 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GSGRMSGGSS CSQTPSRAIP ATRRVVLGDG VQLPPGDYST TPGGTLFSTT PGGTRIIYDR KFLMECRNSP VTKTPPRDLP TIPGVTSPS SDEPPMEASQ SHLRNSPEDK RAGGEESQFE MDI UniProtKB: Eukaryotic translation initiation factor 4E-binding protein 1 |
-分子 #5: GTP-binding protein Rheb
| 分子 | 名称: GTP-binding protein Rheb / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.877826 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GSGRMPQSKS RKIAILGYRS VGKSSLTIQF VEGQFVDSYD PTIENTFTKL ITVNGQEYHL QLVDTAGQDE YSIFPQTYSI DINGYILVY SVTSIKSFEV IKVIHGKLLD MVGKVQIPIM LVGNKKDLHM ERVISYEEGK ALAESWNAAF LESSAKENQT A VDVFRRII LEAEKMDGAA SQGKSSCSVM UniProtKB: GTP-binding protein Rheb |
-分子 #6: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #8: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 8 / コピー数: 2 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 56.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 2) / 使用した粒子像数: 198237 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6bcu: |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X