登録情報 データベース : PDB / ID : 6zg4タイトル Structure of M1-StaR-T4L in complex with HTL0009936 at 2.35A Muscarinic acetylcholine receptor M1,Endolysin,Muscarinic acetylcholine receptor M1 キーワード / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 2.33 Å データ登録者 Rucktooa, P. / Cooke, R.M. ジャーナル : Cell / 年 : 2021タイトル : From structure to clinic: Design of a muscarinic M1 receptor agonist with potential to treatment of Alzheimer's disease.著者: Brown, A.J.H. / Bradley, S.J. / Marshall, F.H. / Brown, G.A. / Bennett, K.A. / Brown, J. / Cansfield, J.E. / Cross, D.M. / de Graaf, C. / Hudson, B.D. / Dwomoh, L. / Dias, J.M. / Errey, J.C. ... 著者 : Brown, A.J.H. / Bradley, S.J. / Marshall, F.H. / Brown, G.A. / Bennett, K.A. / Brown, J. / Cansfield, J.E. / Cross, D.M. / de Graaf, C. / Hudson, B.D. / Dwomoh, L. / Dias, J.M. / Errey, J.C. / Hurrell, E. / Liptrot, J. / Mattedi, G. / Molloy, C. / Nathan, P.J. / Okrasa, K. / Osborne, G. / Patel, J.C. / Pickworth, M. / Robertson, N. / Shahabi, S. / Bundgaard, C. / Phillips, K. / Broad, L.M. / Goonawardena, A.V. / Morairty, S.R. / Browning, M. / Perini, F. / Dawson, G.R. / Deakin, J.F.W. / Smith, R.T. / Sexton, P.M. / Warneck, J. / Vinson, M. / Tasker, T. / Tehan, B.G. / Teobald, B. / Christopoulos, A. / Langmead, C.J. / Jazayeri, A. / Cooke, R.M. / Rucktooa, P. / Congreve, M.S. / Weir, M. / Tobin, A.B. 履歴 登録 2020年6月18日 登録サイト / 処理サイト 改定 1.0 2021年10月6日 Provider / タイプ 改定 1.1 2021年12月1日 Group / カテゴリ / citation_authorItem _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.title / _citation.year 改定 1.2 2021年12月8日 Group / カテゴリ / citation_authorItem _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.pdbx_database_id_PubMed / _citation.title 改定 1.3 2024年1月24日 Group / Refinement descriptionカテゴリ / chem_comp_bond / pdbx_initial_refinement_model改定 1.4 2024年10月16日 Group カテゴリ / pdbx_modification_featureItem
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) Enterobacteria phage T4 (ファージ)
Enterobacteria phage T4 (ファージ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.33 Å
分子置換 / 解像度: 2.33 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell / 年: 2021
ジャーナル: Cell / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6zg4.cif.gz
6zg4.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6zg4.ent.gz
pdb6zg4.ent.gz PDB形式
PDB形式 6zg4.json.gz
6zg4.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6zg4_validation.pdf.gz
6zg4_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6zg4_full_validation.pdf.gz
6zg4_full_validation.pdf.gz 6zg4_validation.xml.gz
6zg4_validation.xml.gz 6zg4_validation.cif.gz
6zg4_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/zg/6zg4
https://data.pdbj.org/pub/pdb/validation_reports/zg/6zg4 ftp://data.pdbj.org/pub/pdb/validation_reports/zg/6zg4
ftp://data.pdbj.org/pub/pdb/validation_reports/zg/6zg4 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト), (組換発現)
Homo sapiens (ヒト), (組換発現)  Enterobacteria phage T4 (ファージ)
Enterobacteria phage T4 (ファージ)
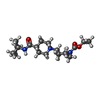








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 1.03319 Å
/ ビームライン: X06SA / 波長: 1.03319 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj














